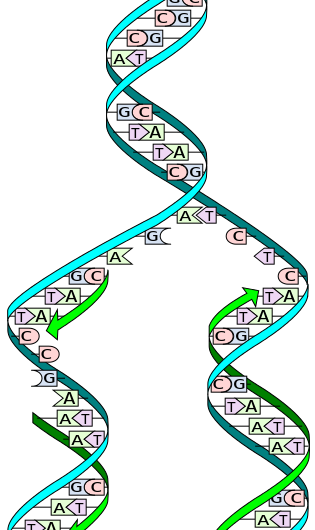
Lors de la réplication, les deux brins sont séparés. Chaque brin de la molécule d'ADN d'origine sert alors de matrice pour la production de son homologue complémentaire. Crédit :Wikimedia Commons
L'analyse de l'ADN est utile pour un certain nombre d'applications vitales. Cela comprend le diagnostic et le suivi des maladies, identification des criminels, et étudier la fonction d'un segment d'ADN ciblé. Cependant, les méthodes utilisées pour les analyses nécessitent souvent plus d'ADN que ce qui peut être disponible dans un échantillon typique. 'Par conséquent, une amplification est nécessaire, mais pas toujours simple. La méthode d'amplification ou de photocopie la plus largement utilisée est la réaction en chaîne par polymérase (PCR). Une nouvelle méthode PCR pourrait aider le processus d'amplification, et ainsi développer des tests robustes qui n'auraient pas été possibles auparavant.
L'acide désoxyribonucléique (ADN) est une molécule présente dans le noyau des cellules et porte les « instructions » pour le développement et le fonctionnement des organismes vivants. Il est souvent comparé à un ensemble de plans car il contient les instructions nécessaires pour construire des cellules. Ces instructions sont divisées en segments le long d'un brin d'ADN.
Réplication
L'ADN est constitué d'une double hélice de deux brins complémentaires constitués de nucléotides, une structure souvent comparée à une échelle. Quand il est temps de répliquer, les deux brins d'ADN – ou « côtés » de l'échelle – se déroulent et se séparent. Une enzyme appelée ADN polymérase lit les brins individuels et fait correspondre les bases complémentaires - les « échelons » de l'échelle - au brin d'origine. De nouveaux brins sont produits des deux côtés de l'ADN d'origine, faire deux doubles hélices d'ADN identiques composées d'un brin original et d'un nouveau brin. Ce processus se produit dans tous les organismes vivants et constitue la base de l'hérédité biologique.
PCR
La réplication peut également être traitée artificiellement. Parfois appelée « photocopie moléculaire, " la réaction en chaîne par polymérase (PCR) est une technique rapide et relativement peu coûteuse utilisée pour amplifier, ou faire de nombreuses copies de, petits segments d'ADN. Ceci est nécessaire car les méthodes utilisées pour analyser l'ADN ou déterminer la séquence de paires de bases d'ADN nécessitent plus d'ADN que ce qui peut être dans un échantillon typique. D'où, le but de la PCR est d'amplifier une région spécifique d'un brin d'ADN.
Pour effectuer la PCR, les deux brins de la double hélice d'ADN sont physiquement séparés en appliquant une température élevée (93-98 °C) dans un processus appelé fusion de l'ADN. Dans la deuxième étape, la température est abaissée et les deux brins d'ADN, maintenant séparé, deviennent des modèles pour les soi-disant amorces. Les amorces sont de courtes molécules d'ADN simple brin, complémentaire de la région d'ADN ciblée pour l'amplification. Après avoir abaissé la température, les amorces ajoutées au processus PCR trouvent et se fixent à sa cible spécifique. Une enzyme appelée ADN polymérase allongera - ou construira - un nouveau brin d'ADN à partir de l'amorce en utilisant la molécule d'ADN simple brin sous-jacente comme matrice.
Construire une échelle
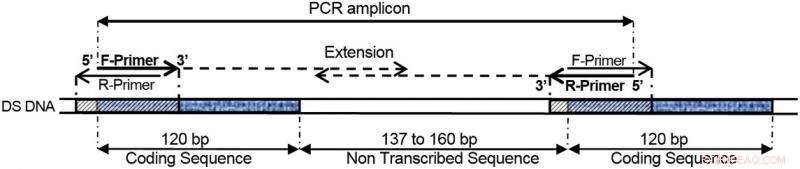
COMPAS-PCR utilisant des amorces hautement complémentaires appliquées aux gènes de répétition directe de l'ADN ribosomique 5S, comme le montre cet exemple. Crédit : Institut norvégien de recherche sur l'eau (NIVA)
Comme mentionné ci-dessus, Les amorces sont de courtes molécules d'ADN simple brin. Celles-ci ont généralement une longueur d'environ 20 nucléotides. Cette chaîne de nucléotides, s'attache spécifiquement au début du brin modèle par appariement de bases - en trouvant les bases complémentaires. L'ADN polymérase est alors capable d'ajouter le prochain nucléotide complémentaire. La polymérase continue d'ajouter plus de nucléotides complémentaires à l'ADN matrice jusqu'à ce qu'un nouveau double brin d'ADN soit terminé, ou pour utiliser la métaphore ci-dessus ; une nouvelle échelle est construite, avec une face d'origine et une face neuve.
Des amorces doubles permettent de définir précisément l'aire d'une molécule d'ADN à amplifier par la PCR. Ces deux amorces flanquantes spécifient où la nouvelle chaîne doit commencer et se terminer.
Un petit changement de paradigme
Une règle centrale fortement ancrée dans l'esprit du scientifique moléculaire est que les amorces ne doivent être complémentaires que de la séquence cible, et non entre eux. Ceci pour éviter que les amorces ne s'utilisent les unes les autres comme matrices et désactivent ainsi une éventuelle implication supplémentaire dans l'amplification de cible traditionnelle.
Dans une étude récente, publié dans la revue scientifique PLOS Un , Le chercheur scientifique principal Marc Anglès d'Auriac de l'Institut norvégien de recherche sur l'eau (NIVA) montre qu'il est possible d'utiliser des amorces très complémentaires tout en évitant les conséquences fâcheuses mentionnées ci-dessus. La nouvelle méthode a été nommée COMPAS-PCR, abréviation de COMPlementary Primer Asymmetric PCR.
En bref, Anglès d'Auriac a observé que des amorces complémentaires entre elles et une cible (triple overlay) pouvaient encore définir un produit d'amplification lorsqu'elles faisaient partie d'un motif ou d'une structure d'ADN répété. Ceci est montré dans la figure ci-dessous. Plus loin, l'obstacle physique à l'amplification de la cible dû à la complémentarité des amorces a été atténué par l'introduction de concentrations d'amorces asymétriques. Asymétrique à cet égard signifie un nombre impair de molécules entre les deux amorces. Une amorce est constituée d'un faible nombre de molécules, l'autre avec un nombre élevé.
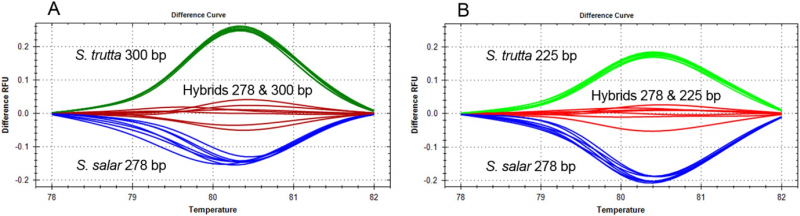
Analyse de Melt haute résolution de deux COMPAS-PCR duplex à trois amorces pour la différenciation de S. salar, S. trutta et hybrides. Crédit : Institut norvégien de recherche sur l'eau (NIVA)
"Si vous utilisez des concentrations égales, comme vous le faites habituellement pour la PCR, les amorces, présents en quantités égales, se colleront les uns aux autres, " dit Anglès d'Auriac.
"Avec des concentrations asymétriques, l'amorce en excès ou fortement concentrée a un certain nombre de molécules non collées avec l'amorce limitante, et est donc disponible pour l'amplification de la cible."
Travailler seul
"Cette approche contre-intuitive va isoler, mais aussi protéger, l'amorce basse concentration, " dit Anglès d'Auriac.
L'apprêt limitant étant collé, et ainsi protégé, il est possible que le primaire haute concentration agisse seul, C'est, faire une copie simple brin de l'ADN cible. Lorsque suffisamment d'amplicons simple brin sont produits, ils peuvent servir de matériau de matrice pour l'amorce à faible concentration. L'amorce limitante est libérée, et la réaction passe à l'amplification PCR exponentielle classique.
« Cela élargit les possibilités d'application de la PCR pour le scientifique, Anglès d'Auriac élucide."
Structures répétitives
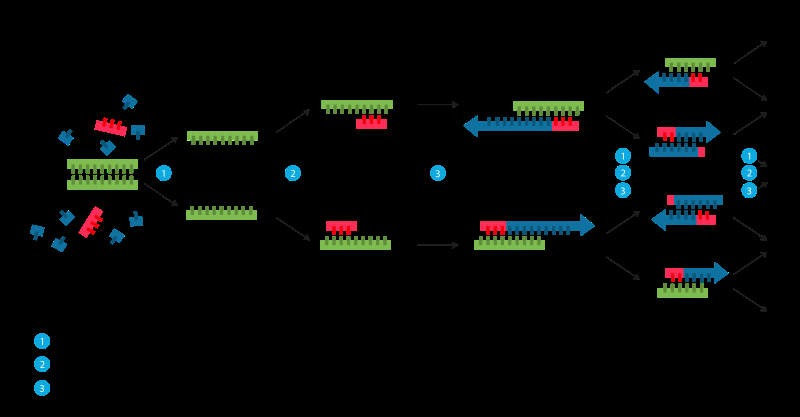
Réaction en chaîne par polymérase (PCR) . Crédit :Wikimédia
Dans de nombreux organismes, une fraction importante de l'ADN génomique est hautement répétitive, avec plus de la moitié de la séquence constituée d'éléments répétitifs chez l'homme.
C'est en effet une structure d'ADN répétitive qui a poussé Anglès d'Auriac à sortir des sentiers battus et à mettre en avant le principe COMPAS-PCR, un petit changement de paradigme dans le domaine de la photocopie moléculaire.
Dans le processus d'effectuer un diagnostic basé sur l'ADN pour les salmonidés, en particulier l'identification de la truite fario (Salmo trutta) et du saumon atlantique (Salmo salar) étroitement apparentés, Anglès d'Auriac a eu du mal à séparer ces espèces – y compris les hybrides entre elles. Une identification rapide et précise permettrait d'améliorer la surveillance et les études des écosystèmes fluviaux, car l'identification des hybrides est importante pour estimer la santé écologique des bassins fluviaux. Après avoir utilisé COMPAS-PCR, Anglès d'Auriac a pu appliquer une méthode d'analyse de produit PCR appelée analyse de fusion à haute résolution dans l'identification de la truite fario, Saumon atlantique et leurs hybrides en un seul test.
Anglès d'Auriac souligne cependant que le principe COMPAS-PCR ne se limite pas à l'identification des espèces de poissons. L'utilisation d'amorces presque entièrement complémentaires ciblant la même séquence peut s'appliquer à toutes les copies adjacentes les unes aux autres, dans des motifs d'ADN d'intérêt en tant que séquences cibles.
« Les séquences répétées d'ADN représenteraient plus de 50 % du génome humain et sont présentes dans de nombreuses familles de gènes « de ménage » telles que le gène ribosomique 5S utilisé dans cette étude. Par conséquent, les principes généraux de COMPAS-PCR permettront de développer de nouvelles stratégies d'amplification d'ADN tirant parti de ces structures d'ADN répétées, " conclut Marc Anglès d'Auriac.