Les bactéries existaient depuis des millénaires avant l’homme et nous infectent depuis le début. Bien que nous puissions traiter les infections grâce aux produits pharmaceutiques, les bactéries continuent de devenir résistantes aux traitements en raison de leur évolution rapide. Les infections bactériennes restent l'une des principales causes de morbidité et de mortalité en 2024, entraînant près de huit millions de décès annuels dans le monde.
Une caractéristique clé partagée par toutes les bactéries infectieuses est appelée chimiotaxie. La chimiotaxie est un processus polyvalent qui permet aux bactéries de nager vers des molécules riches en énergie, de trouver des niches préférées pour l'infection, d'éviter les espèces nuisibles, de changer de vitesse et de s'arrêter complètement pour former des biofilms. La chimiotaxie est également essentielle à la virulence chez les animaux et constitue une cible potentielle pour de nouveaux traitements. Mais d'abord, le processus lui-même doit être mieux compris.
Le laboratoire de Tina Iverson, professeure Louise B. McGavock et professeur de pharmacologie, en collaboration avec des chercheurs de l'Université de Californie à San Francisco ; Université de Stanford; et l'Institut Weizmann des Sciences en Israël ont publié de nouveaux travaux dans Nature Microbiology , fournissant de nouvelles informations sur la chimiotaxie.
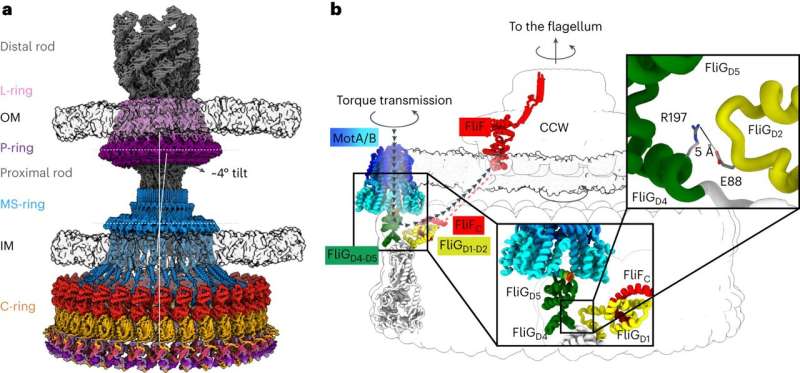
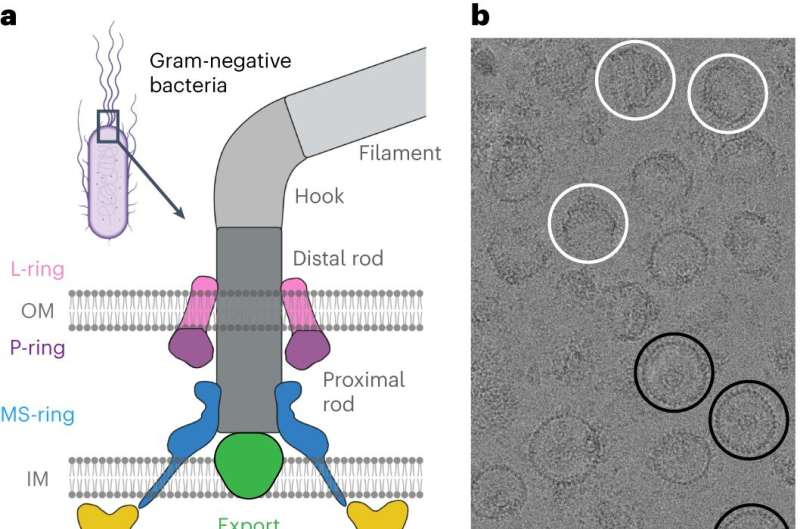
La chimiotaxie nécessite un petit moteur pour faire tourner un flagelle, un appendice pileux sur une bactérie qui tourne pour assurer la propulsion, comme un moteur de bateau. La rotation du flagelle dans le sens des aiguilles d'une montre ou dans le sens inverse à des vitesses différentes permet aux bactéries de se rapprocher ou de s'éloigner de différents stimuli. Les recherches actuelles n'ont pas abouti à une architecture convenue des composants centraux du moteur qui alimente le flagelle, ce qui a entravé la compréhension et la capacité des chercheurs à cibler la chimiotaxie avec des médicaments.
Les travaux actuels, menés par Prashant Singh, associé de recherche principal au laboratoire d'Iverson, présentent de nouvelles informations sur la manière dont un composant moteur appelé interrupteur inverse la rotation et transmet le couple au flagelle.
Pour ce faire, les chercheurs ont pris comme modèle Salmonella enterica, une bactérie responsable d’environ 60 000 décès par an dans le monde. Après avoir isolé et purifié les moteurs de S. enterica stabilisés dans différentes configurations de nage, les collaborateurs ont exploité la puissance du Titan Krios de Vanderbilt, un microscope cryoélectronique de 10 millions de dollars acquis par l'École de médecine des sciences fondamentales et mis à disposition via le Cryo du Centre de biologie structurale. -Facilité EM.
Les structures ont fourni aux chercheurs des informations sur la manière dont le moteur bactérien alimente la rotation du flagelle dans le sens horaire et antihoraire, ce qui permet à une bactérie de nager droit ou de changer de direction en nageant. Cela les a également aidés à comprendre comment les protéines se lient au moteur pour aider à réguler le mouvement bactérien.
Ces résultats sont applicables à un large éventail d’infections. Par exemple, la machinerie de chimiotaxie de Salmonella est presque identique à celle d’Escherichia coli, responsable de plus de 250 000 infections par an rien qu’aux États-Unis. Étant donné que la chimiotaxie est nécessaire à l'infection, la perturbation sélective des interactions qui permettent aux agents pathogènes de former un réservoir au sein d'un organisme peut aider à prévenir les infections récurrentes sans avoir d'impact sur le microbiome normal.
Le laboratoire d'Iverson travaille actuellement à identifier comment une gamme élargie de différents partenaires protéiques se lient au moteur flagellaire pendant la chimiotaxie, et espère que cela permettra de trouver des moyens de perturber la chimiotaxie pendant l'infection.
Plus d'informations : Prashant K. Singh et al, Les structures CryoEM révèlent comment le flagelle bactérien tourne et change de direction, Nature Microbiology (2024). DOI : 10.1038/s41564-024-01674-1
Informations sur le journal : Microbiologie naturelle
Fourni par l'Université Vanderbilt