Résumé graphique. Crédit :Cellulaire (2022). DOI :10.1016/j.cell.2022.08.003
Des études clés de la dernière décennie ont montré que le microbiome intestinal, la collection de centaines d'espèces bactériennes qui vivent dans le système digestif humain, influence le développement neuronal, la réponse aux immunothérapies anticancéreuses et d'autres aspects de la santé. Mais ces communautés sont complexes et sans moyens systématiques d'étudier les constituants, les cellules et molécules exactes liées à certaines maladies restent un mystère.
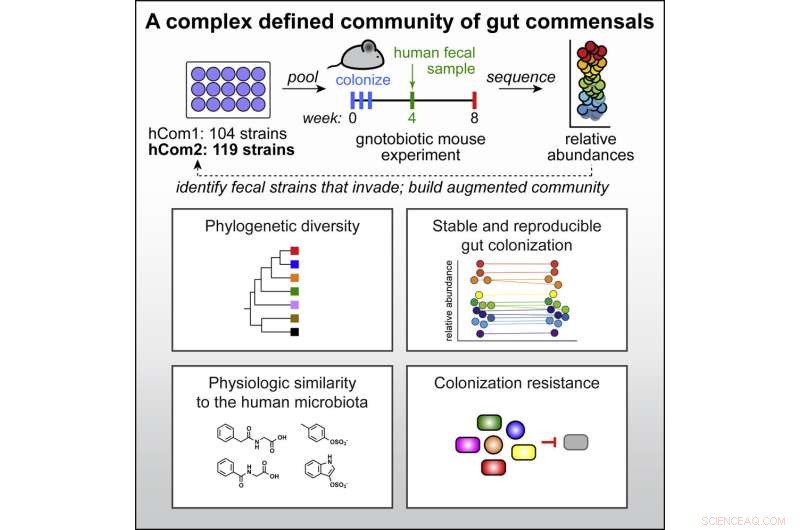
Les chercheurs de l'Université de Stanford ont construit le microbiome synthétique le plus complexe et le mieux défini, créant une communauté de plus de 100 espèces bactériennes qui ont été transplantées avec succès chez des souris. La possibilité d'ajouter, de supprimer et de modifier des espèces individuelles permettra aux scientifiques de mieux comprendre les liens entre le microbiome et la santé, et éventuellement de développer des thérapies de microbiome de première classe.
De nombreuses études clés sur le microbiome ont été réalisées à l'aide de greffes fécales, qui introduisent l'intégralité du microbiome naturel d'un organisme à un autre. Alors que les scientifiques font systématiquement taire un gène ou suppriment une protéine d'une cellule spécifique ou même d'une souris entière, il n'existe aucun ensemble d'outils de ce type pour supprimer ou modifier une espèce parmi des centaines dans un échantillon fécal donné.
"Une grande partie de ce que nous savons sur la biologie, nous ne le saurions pas sans la capacité de manipuler des systèmes biologiques complexes par morceaux", a déclaré Michael Fischbach, chercheur à l'Institut Sarafan ChEM-H et auteur correspondant de l'étude, publiée dans Cellule le 6 septembre.
Fischbach, qui est professeur agrégé de bio-ingénierie, de microbiologie et d'immunologie, et d'autres ont vu une solution :créer un microbiome à partir de zéro en cultivant individuellement puis en mélangeant ses bactéries constitutives.
Construire l'arche
Chaque cellule du microbiome occupe une niche fonctionnelle spécifique, effectuant des réactions qui décomposent et accumulent des molécules. Pour construire un microbiome, l'équipe devait s'assurer que le mélange final était non seulement stable, en maintenant un équilibre sans qu'aucune espèce ne domine le reste, mais aussi fonctionnel, exécutant toutes les actions d'un microbiome complet et naturel. La sélection des espèces à inclure dans leur communauté synthétique était également difficile compte tenu de la variation naturelle entre les individus; deux personnes choisies au hasard partagent moins de la moitié de leurs gènes microbiens.
Les chercheurs ont décidé de construire leur colonie à partir des bactéries les plus répandues et se sont tournés vers le Human Microbiome Project (HMP), une initiative des National Institutes of Health pour séquencer les génomes microbiens complets de plus de 300 adultes.
"Nous recherchions l'arche de Noé des espèces bactériennes dans l'intestin humain, en essayant de trouver celles qui étaient presque toujours présentes chez n'importe quel individu", a déclaré Fischbach.
Ils ont sélectionné plus de 100 souches bactériennes présentes chez au moins 20 % des individus HMP. L'ajout de quelques espèces nécessaires pour certaines études ultérieures les a amenés à 104 espèces, qu'ils ont cultivées dans des stocks individuels, puis mélangées en une seule culture combinée pour créer ce qu'ils appellent la communauté humaine une, ou hCom1.
Bien que convaincus que les souches puissent coexister en laboratoire, le véritable test était de savoir si leur nouvelle colonie prendrait racine dans l'intestin. Ils ont introduit hCom1 chez des souris soigneusement conçues pour ne pas avoir de bactéries présentes. hCom1 était remarquablement stable, avec 98 % des espèces constitutives colonisant l'intestin de ces souris sans germes, et les niveaux d'abondance relative de chaque espèce restant constants sur deux mois.
Invasion étrangère
Pour rendre leur colonie plus complète, les chercheurs voulaient s'assurer que toutes les fonctions vitales du microbiome seraient assurées par une ou plusieurs espèces. They relied on a theory called colonization resistance, which explains that any bacterium, when introduced into an existing colony, will only survive if it can fill a niche not already occupied.
By introducing a complete microbiome, in the form of a human fecal sample, to their colony and tracking any new species that took up residence, they could build a more complete community.
Some were skeptical that this would work. "The bacterial species in hCom1 had lived together for only a few weeks," said Fischbach. "Here we were, introducing a community that had coexisted for a decade. Some people thought they would decimate our colony."
Remarkably, hCom1 held its own, and only about 10% of cells in the final community came from the fecal transplant.
They found over 20 new bacterial species that inserted themselves in at least two of their three fecal transplant studies. Adding those to their initial community and removing those that failed to take root in mouse guts gave them a new community of 119 strains, dubbed hCom2. This second iteration, still made from individually growing and then mixing the constituents, made mice even more resistant to fecal challenges than the first.
Final challenge
To demonstrate the utility of their synthetic microbiome, the team took hCom2-colonized mice and challenged them with a sample of E. coli. These mice, like those that were colonized with a natural microbiome, resisted infection.
Prior studies have shown that a healthy natural microbiome leads to protection, but Fischbach and colleagues could take this a step further by iteratively eliminating or modifying certain strains to determine which ones specifically conferred protection. They found several key bacteria and plan to conduct further studies to narrow down to the most critical species.
Fischbach believes that hCom2, or future versions of it, will enable similar reductionist studies that reveal the bacterial agents involved in other areas, like immunotherapy responses.
"We built this consortium for the broader research community. We want to get this into as many hands as possible to have an impact on the field," said Fischbach.
He also envisions that this method of building a microbiome from the ground up will make engineered microbiome-based therapies possible in the future. As the director of the Stanford Microbiome Therapies Initiative (MITI), an initiative launched in 2019 by Sarafan ChEM-H and the Department of Bioengineering, he aims to construct engineered communities that could one day be transplanted into people to treat or prevent a variety of diseases. Ecological principles at play in gut microbiome