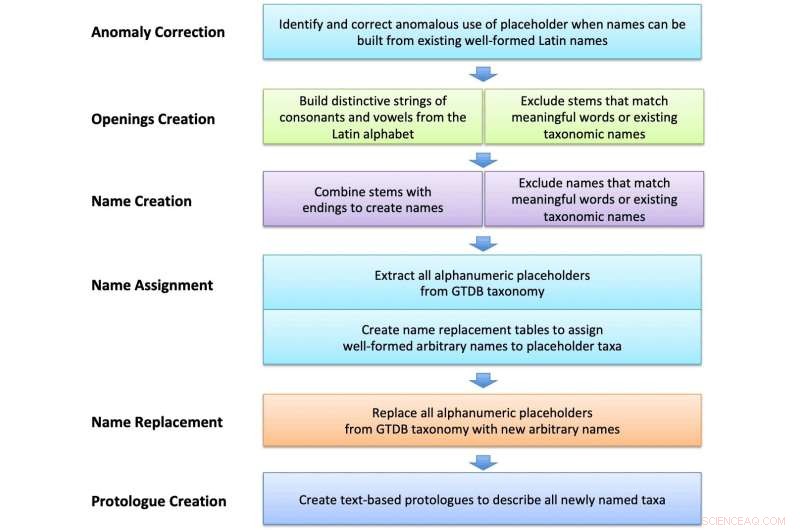
Flux de travail schématique pour la création et l'attribution de noms latins arbitraires. Les chaînes construites à partir de lettres latines ont été sélectionnées et organisées pour exclure les composants indésirables et les mots significatifs. Ceux-ci ont été combinés avec des suffixes ou des mots latins pour créer des noms, qui ont ensuite été utilisés pour nommer les taxons d'espace réservé dans les fichiers GTDB et créer des protologues textuels. Crédit :Journal international de microbiologie systématique et évolutive (2022). DOI :10.1099/ijsem.0.005482
Dans un article récemment publié dans le International Journal of Systematic and Evolutionary Microbiology , des chercheurs au Royaume-Uni et en Autriche ont nommé plus de 65 000 types différents de microbes. L'étude, dirigée par le professeur Mark Pallen du Quadram Institute de Norwich, s'appuie sur une longue tradition de création de noms latins bien formés mais arbitraires pour de nouvelles espèces, mais applique cette approche à une échelle sans précédent dans l'histoire de la taxonomie.
Le professeur Pallen soutient que la microbiologie est victime de son propre succès, avec des dizaines de milliers de nouvelles espèces découvertes ces dernières années, mais la plupart restent sans nom. Dans le passé, les bactéries ont reçu des noms descriptifs ou ont été nommées d'après des personnes ou des lieux. Cette approche fournit actuellement environ un millier de nouveaux noms d'espèces par an. Cependant, avec un arriéré de plus de 50 000 espèces bien classées mais sans nom, à ce rythme de progression, il faudrait au moins un demi-siècle pour nommer toutes ces bactéries sans nom - à ce moment-là, les scientifiques seraient confrontés au problème de nommer les millions d'espèces supplémentaires. découvert entre-temps.
Pallen et ses collègues ont adopté une approche efficace, à haut débit et à grande échelle de données, en utilisant un programme informatique pour générer des dizaines de milliers de noms distinctifs mais faciles à utiliser qui ne ressemblent à aucun mot existant. Pour se conformer à l'exigence selon laquelle les noms de bactéries doivent être en latin, l'équipe a combiné des chaînes arbitraires de lettres de l'alphabet latin avec des suffixes féminins grammaticalement bien formés. Le résultat est un ensemble de noms qui rappellent la familiarité et la gravité du latin, même s'ils manquent de tout pedigree significatif. Les exemples incluent Dupisella tifacia pour une bactérie de l'intestin de mouton, Hopelia gocarosa pour une bactérie vivant dans les eaux souterraines suédoises ou Saxicetta apufaria d'un lac salé russe.
Bien que cela puisse sembler radical, la formation de noms de manière arbitraire a en fait une longue tradition, remontant au premier code de nomenclature taxonomique en 1869 et même au père de la taxonomie, le naturaliste suédois Linnaeus. Ce qui est différent ici, c'est le sens étonnant de l'échelle, avec un catalogue de noms qui s'étend sur plus de dix mille pages dans ce qui représente la plus grande dénomination d'espèces jamais publiée dans une seule publication.
Parce qu'ils ont été appliqués à des espèces découvertes par séquençage d'ADN mais pas encore cultivées en laboratoire, les nouveaux noms restent pour l'instant provisoires plutôt permanents. Cependant, comme le code de nomenclature insiste sur le fait que les bactériologistes visent la stabilité des noms et qu'aucune approche concurrente de dénomination à l'échelle n'est en cours, il semble probable que la grande majorité des nouveaux noms seront utilisés pendant des années, voire des siècles.
En conclusion, Pallen dit "J'étais membre du groupe de travail qui nous a donné des lettres grecques pour les variantes COVID, qui ont été rapidement adoptées par la communauté scientifique. J'espère que les noms proposés ici seront également rapidement adoptés et largement utilisés. C'est juste La première étape. L'ère de la découverte microbienne est loin d'être révolue, mais il sera facile de créer de futurs noms en masse en utilisant les principes que nous avons établis ici. Un système automatisé pour générer un million de nouveaux noms de bactéries