À l'aide de Cas13 guidé par la structure, des chercheurs de l'Université Rice et de l'Université du Connecticut ont modifié un outil d'édition de gènes pour servir de test de diagnostic très sensible pour la présence du virus SARS-CoV-2. Ils ont utilisé un capteur électrochimique standard pour fournir des résultats. Crédit :Jie Yang/Université Rice
Une méthode technique basée sur CRISPR qui trouve l'ARN du SRAS-CoV-2, le virus qui cause le COVID-19, promet de rendre le test de cette maladie et d'autres maladies rapide et facile.
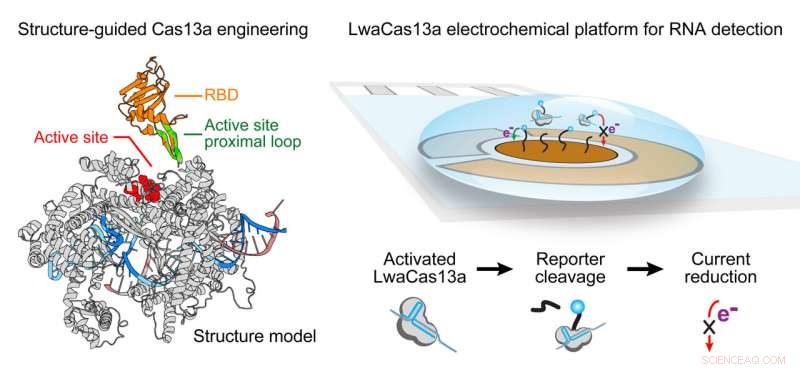
Des collaborateurs de l'Université Rice et de l'Université du Connecticut ont en outre conçu le système CRISPR-Cas13 d'édition d'ARN pour augmenter leur puissance de détection d'infimes quantités de virus SARS-CoV-2 dans des échantillons biologiques sans l'étape fastidieuse d'extraction et d'amplification d'ARN nécessaire dans test PCR de référence.
La nouvelle plate-forme a connu un grand succès par rapport à la PCR, trouvant 10 positifs sur 11 et aucun faux positif pour le virus lors de tests sur des échantillons cliniques directement à partir d'écouvillons nasaux. Les chercheurs ont montré que leur technique trouve des signes de SRAS-CoV-2 dans l'attomolaire (10 -18 ) concentration.
L'étude menée par l'ingénieur chimiste et biomoléculaire Xue Sherry Gao de la George R. Brown School of Engineering de Rice et les chercheurs postdoctoraux Jie Yang de Rice et Yang Song du Connecticut apparaît dans Nature Chemical Biology .
Cas13, comme son cousin plus connu Cas9, fait partie du système par lequel les bactéries se défendent naturellement contre les phages envahisseurs. Depuis sa découverte, CRISPR-Cas9 a été adapté par des scientifiques pour éditer des génomes d'ADN vivants et s'avère très prometteur pour traiter et même guérir des maladies.

Le chercheur postdoctoral de l'Université Rice, Jie Yang, a mené un effort pour adapter les outils d'édition du génome Cas13 pour servir de détecteur très sensible de la présence du virus SARS-CoV-2, qui cause le COVID-19. Crédit :Jeff Fitlow/Rice University
Et il peut être utilisé d'autres manières. Cas13 seul peut être amélioré avec de l'ARN guide pour trouver et couper des séquences d'ARN cibles, mais aussi pour trouver une "collatérale", dans ce cas la présence de virus comme le SARS-CoV-2.
"La protéine Cas13 conçue dans ce travail peut être facilement adaptée à d'autres plates-formes précédemment établies", a déclaré Gao. "La stabilité et la robustesse des variantes Cas13 conçues les rendent plus adaptées aux diagnostics au point de service dans les zones à faibles ressources lorsque les machines PCR coûteuses ne sont pas disponibles."
Yang a déclaré que le Cas13 de type sauvage, tiré d'une bactérie, Leptotrichia wadei, ne peut pas détecter le niveau attomolaire d'ARN viral dans un délai de 30 à 60 minutes, mais la version améliorée créée à Rice fait le travail en environ une demi-heure et détecte le SRAS -CoV-2 à des concentrations beaucoup plus faibles que les tests précédents.
Elle a dit que la clé est une boucle en épingle à cheveux flexible et bien cachée près du site actif de Cas13. "C'est au milieu de la protéine près du site catalytique qui détermine l'activité de Cas13", a déclaré Yang. "Étant donné que Cas13 est vaste et dynamique, il était difficile de trouver un site pour insérer un autre domaine fonctionnel."
Les chercheurs ont fusionné sept domaines de liaison d'ARN différents à la boucle, et deux des complexes étaient clairement supérieurs. Lorsqu'ils trouvaient leurs cibles, les protéines devenaient fluorescentes, révélant la présence du virus.
"Nous avons pu voir que l'activité accrue était cinq ou six fois supérieure à celle du Cas13 de type sauvage", a déclaré Yang. "Ce nombre semble petit, mais c'est assez étonnant avec une seule étape d'ingénierie des protéines.
"But that was still not enough for detection, so we moved the whole assay from a fluorescence plate reader, which is quite large and not available in low-resource settings, to an electrochemical sensor, which has higher sensitivity and can be used for point-of-care diagnostics," she said.
With the off-the-shelf sensor, Yang said the engineered protein was five orders of magnitude more sensitive in detecting the virus compared to the wild-type protein.
The lab wants to adapt its technology to paper strips like those in home COVID-19 antibody tests, but with much higher sensitivity and accuracy. "We hope that will make testing more convenient and with lower cost for many targets," Gao said.
The researchers are also investigating improved detection of the Zika, dengue and Ebola viruses and predictive biomarkers for cardiovascular disease. Their work could lead to rapid diagnosis of the severity of COVID-19.
"Different viruses have different sequences," Yang said. "We can design guide RNA to target a specific sequence that we can then detect, which is the power of the CRISPR-Cas13 system."
But because the project began just as the pandemic took hold, SARS-CoV-2 was a natural focus. "The technology is quite amenable to all the targets," she said. "This makes it a very good option to detect all kinds of mutations or different coronaviruses."
"We are very excited about this work as a combinational effort of structure biology, protein engineering and biomedical device development," Gao added. "I greatly appreciate all the efforts from my lab members and collaborators."
Co-authors of the paper are Rice postdoctoral researcher Xiangyu Deng, undergraduate Jeffrey Vanegas and graduate student Zheng You; graduate students Yuxuan Zhang and Zhengyan Weng of the University of Connecticut; microbiology supervisor Lori Avery and Kevin Dieckhaus, a professor of medicine, of UConn Health; Yi Zhang, an assistant professor of biomedical engineering at the University of Connecticut; and Yang Gao, an assistant professor of biosciences at Rice. Compact CRISPR system enables portable COVID-19 testing