
Modèle moléculaire montrant dCas9 lié à une molécule guide de fusion ARN-ARN origami qui apporte des facteurs de transcription à une séquence promotrice. Crédit :Cody Geary, Université d'Aarhus
Le développement d'outils pour un contrôle précis des processus biologiques a été l'un des principaux piliers du domaine désormais mature de la biologie synthétique. Ces outils scientifiques empruntent des principes à une multitude de domaines de recherche qui, lorsqu'ils sont combinés, permettent des applications uniques potentiellement transformatrices pour la société moderne.
La traduction des innovations modernes de la nanotechnologie de l'ARN dans le contexte biologique possède un immense potentiel en raison de la compatibilité avec le repliement et l'expression dans les cellules, mais elle impose également des défis uniques tels que des conditions de performance strictes et l'instabilité inhérente des molécules d'ARN.
Cependant, une approche récente de conception d'ARN structurel développée dans le laboratoire d'Andersen, appelée "origami d'ARN", tente de résoudre ce problème. Cette approche tente de générer des dispositifs complexes à base d'ARN artificiels qui sont stables dans les cellules, interagissent avec d'autres biomolécules, y compris d'autres ARN et protéines, et permettent des applications uniques, en particulier dans le contexte de la régulation des gènes.
Démontré par deux approches distinctes récemment publiées dans Nucleic Acids Research , l'origami d'ARN est présenté comme une plate-forme de conception d'ARN sophistiquée qui, lorsqu'elle est appliquée dans le contexte cellulaire, génère des molécules uniques pour une régulation basée sur la biologie synthétique.
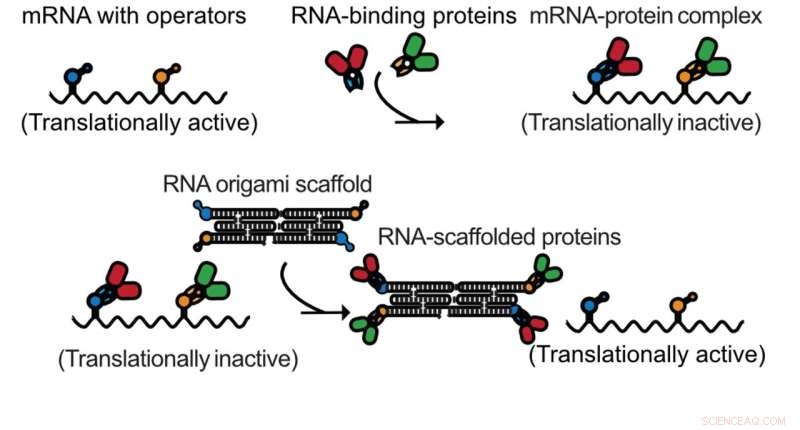
Un ARNm avec des opérateurs est inhibé par les protéines qu'ils expriment. Les molécules d'origami d'ARN servent d'éponges qui lient les protéines et rendent à nouveau les ARNm actifs en traduction. Crédit :ACS Biologie synthétique (2022)
Les éponges à ARN régulent la production d'enzymes chez les bactéries
Dans la première approche, l'origami d'ARN a été utilisé pour obtenir un contrôle précis des niveaux de production de protéines lorsqu'elles sont exprimées dans des bactéries. Des cassettes d'expression de protéines auto-inhibantes ont été fabriquées en installant un site de liaison fort pour la protéine exprimée dans son propre gène. Ensuite, l'origami d'ARN décoré des mêmes sites de liaison aux protéines a été exprimé en grand excès.
De cette façon, l'origami d'ARN sert d'éponge protéique qui séquestre les protéines dans la cellule et permet l'expression de la protéine auto-inhibée. Il a été démontré que ce concept général permet la régulation de plusieurs protéines simultanément et active les voies enzymatiques pour de meilleurs rendements de produits.
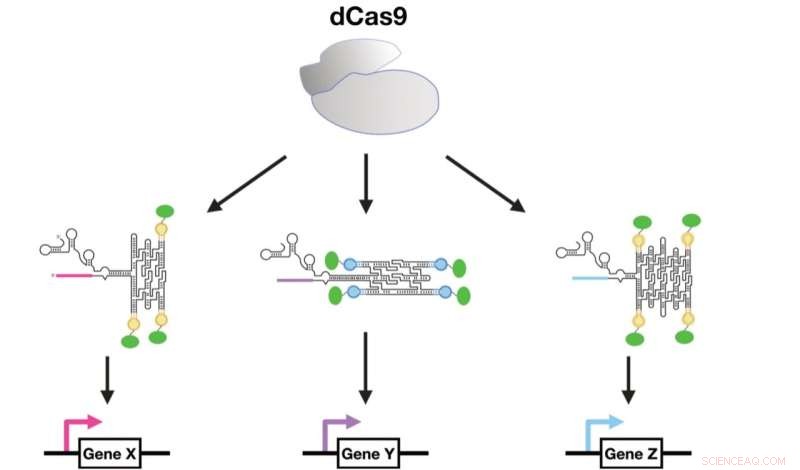
CRISPR-dCas9 fonctionne comme un régulateur principal des molécules de fusion sgRNA - RNA origami qui apportent des facteurs de transcription à une séquence promotrice. Graphismes de George Pothoulakis. Crédit :Recherche sur les acides nucléiques (2022). DOI :10.1093/nar/gkac470
Régulateurs basés sur CRISPR pour les usines chimiques de levure
Dans la deuxième approche, l'origami d'ARN a été combiné avec CRISPR, l'une des techniques de biologie moléculaire modernes les plus populaires, pour réguler l'expression des gènes dans la levure. Les origamis d'ARN ont été intégrés dans les petits ARN qui guident CRISPR-Cas9 pour cibler des séquences spécifiques dans le génome d'ADN.
Les échafaudages d'origami d'ARN ont été décorés avec des sites de liaison aux protéines capables de recruter des facteurs de transcription. En ciblant les échafaudages d'ARN sur les régions promotrices, les facteurs de transcription ont activé l'expression des gènes. Il a été démontré que la force d'expression peut être réglée par l'orientation de l'échafaudage et la quantité de facteurs de transcription recrutés. Enfin, il a été démontré que les voies multi-enzymes pouvaient être contrôlées pour une production à haut rendement de la violacéine, un médicament anticancéreux. Système de contrôle qualité des protéines synthétiques chez les bactéries