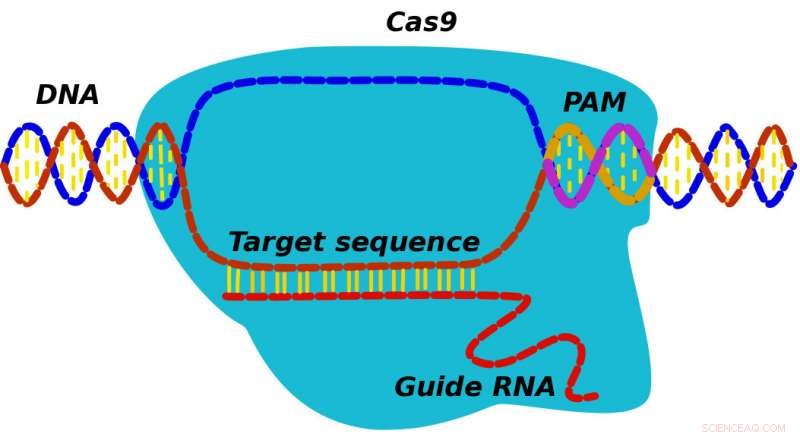
Lorsqu'une protéine CRISPR-Cas9 trouve sa cible, il trouve d'abord une séquence d'espacement connue sous le nom de PAM, puis recherche l'ADN adjacent pour voir s'il correspond à l'ARN guide de Cas9. Un nouveau modèle développé par des chercheurs de l'Université Rice pourrait aider à découvrir des détails sur le mécanisme par lequel CRISPR-Cas9 peut remplacer les mutations par un ADN correct. Crédit :Alexey Shvets/Université Rice
Des chercheurs de l'Université Rice ont développé un modèle informatique pour quantifier le mécanisme par lequel les protéines CRISPR-Cas9 trouvent leurs cibles d'édition du génome.
Anatoli Kolomeisky, un professeur Rice de chimie et de génie chimique et biomoléculaire, et l'ancien élève Alexey Shvets a adapté un système qu'ils ont développé plus tôt pour montrer comment les protéines trouvent généralement leurs cibles biologiques. Ils espèrent que le modèle révisé aidera à percer les mystères restants de CRISPR.
A l'état naturel, CRISPR, qui signifie « répétitions palindromiques courtes regroupées régulièrement espacées, " est le mécanisme biologique par lequel les bactéries se protègent des infections virales. Les bactéries incorporent une copie de l'ADN étranger et construisent un enregistrement de tous ceux qui envahissent. Elles se réfèrent à cet enregistrement lorsque de nouveaux envahisseurs sont détectés et l'utilisent pour les détruire.
Dans les années récentes, les chercheurs ont commencé à adapter le mécanisme pour l'utiliser dans l'édition du génome, qui a le potentiel de guérir les maladies et d'améliorer les organismes, y compris les humains. Mais une pierre d'achoppement a été le risque que les protéines CRISPR-Cas9, l'un des systèmes utilisant l'approche CRISPR, coupera et remplacera les mauvaises séquences cibles, introduire des mutations.
Le modèle Rice décrit dans le Journal biophysique trouvé probable que CRISPR-Cas9 localise les bonnes cibles plus efficacement lorsque ces modifications hors cible sont autorisées, parce que les protéines ne perdent pas de temps à se dissocier des cibles hors cible pour continuer la recherche.
Cela peut ou peut ne pas être une bonne chose, mais c'est certainement digne d'étude, dit Kolomeisky.
« Le taux d'erreur (coupe hors cible) est parfois de 10 à 20 %, " a-t-il dit. " Nous avons deux idées à ce sujet :l'une est que les virus mutent très rapidement et peut-être que les bactéries essaient de couper des cibles qui ne sont que légèrement mutées afin d'être plus flexibles. L'autre est qu'il existe des protéines qui peuvent corriger les erreurs, donc s'il n'y a pas beaucoup de mauvaises coupes, le système peut les tolérer.
Kolomeisky a déclaré que son modèle est une étape simple vers la compréhension de la dynamique de l'édition CRISPR. "CRISPR-Cas9 est la variante la plus populaire car elle n'a qu'une seule protéine et est plus facile, biologiquement, travailler avec, " il a dit.
Le laboratoire Rice a développé son modèle original pour apprendre comment les protéines glissent le long de l'ADN pour trouver des cibles et déclencher des processus comme la transcription des gènes. Kolomeisky a noté que la pionnière de CRISPR, Jennifer Doudna, a découvert que CRISPR-Cas9 ne cherche pas de la même manière. "Elle a trouvé qu'il ne glisse nulle part sur l'ADN, " il a dit.
Au lieu, selon Doudna et son équipe, la protéine reconnaît initialement les séquences PAM à trois nucléotides (pour protospacer adjacent motif) qui marquent l'emplacement des cibles potentielles. "CRISPR trouve et se lie à PAM, puis son ARN associé explore l'ADN adjacent pour voir s'il s'agit de la cible, " dit Kolomeisky. " Si c'est le cas, la protéine commence à couper. Si non, il se désolidarise et regarde ailleurs."
Dans les expériences ultérieures de Doudna avec suppression des séquences PAM, Les protéines CRISPR-Cas9 n'ont pas du tout pu trouver leurs cibles. Les PAMs ont donc un rôle important et ne sont pas seulement un espaceur générique, il a dit. "Dès que j'ai lu ceci, J'ai compris que nous pouvions également utiliser notre modèle ici."
Le modèle théorique examine les processus de premier passage - ceux qui se produisent lorsqu'un système franchit un seuil physique ou chimique, comme trouver un PAM pertinent - pour suivre les protéines CRISPR-Cas9 insérées dans une cellule lorsqu'elles examinent d'abord les séquences PAM, puis, tout en étant lié aux PAM, rechercher la cible d'ADN qui correspond à l'ARN de Cas9.
Ils ont découvert que les CRISPR qui évitent les coupures hors cible en se dissociant du «mauvais» ADN prennent plus de temps à s'installer que ceux qui coupent simplement les cibles. "Aller au mauvais PAM prend du temps, " a déclaré Kolomeisky. " Notre calcul montre que CRISPR peut trouver de vraies cibles plus rapidement lorsqu'il coupe parfois aux mauvais endroits. La fraction qui va aux bonnes cibles peut être plus petite, mais vous finirez par les couper.
"C'est un modèle simple et exactement résoluble, " a dit Kolomeisky. " Si quelqu'un veut tester, le modèle peut fournir des prédictions spécifiques et, dans certains cas, proposer des tendances pour ce qui devrait être observé. nucléotide par nucléotide.
"La chose la plus impressionnante à propos de CRISPR n'est pas la découverte d'un système immunitaire chez les bactéries mais le fait que cela a créé une révolution dans la biotechnologie, car cela signifie que dans n'importe quelle cellule, nous pouvons couper n'importe quel ADN à un endroit spécifique, très précisément, " a déclaré Kolomeisky. " J'espère que nos travaux stimuleront des études plus fondamentales, car j'aime beaucoup la méthode CRISPR. Mais je ne suis pas content quand les gens l'appliquent sans comprendre comment cela fonctionne au niveau moléculaire."
Shvets est maintenant chercheur postdoctoral au Massachusetts Institute of Technology. Kolomeisky est professeur de chimie et de génie chimique et biomoléculaire.