
Conception et mise en œuvre de l'imagerie MATRIEX :(a) Schéma expérimental du système d'imagerie MATRIEX. Les deux objets 3D ronds dans le coin inférieur gauche sont les vues supérieure et inférieure de la chambre de la tête de souris utilisée pour l'imagerie in vivo. (Ti:Sa) : Laser pulsé ultrarapide Ti :Saphir ; PC :cellule de Pockels ; BE :extenseur de faisceau; SM1 et SM2 :miroirs de balayage x–y ; SL :objectif de balayage ; TL :lentille tubulaire; DM :miroir dichroïque; CL :lentille de collecte; PMT :tube photomultiplicateur; FAIRE :objectif sec; MO :objectifs miniaturisés. (b) Photographie montrant une vue d'ensemble oblique du système d'imagerie MATRIEX réel. (c) La photographie de l'image supérieure montre une vue agrandie des trois MO attachés aux barres de manipulation au-dessus de la chambre principale ; la photographie du bas a été prise directement au-dessus des MO avec un appareil photo pour smartphone. Tous les MO utilisés dans cette figure sont du même modèle :« version standard ». (d, e) Illustrations du grossissement à deux étages et du couplage multiaxes. Les images carrées sont de véritables images à deux photons prises de billes de 20 µm. Chaque cercle rouge indique un FOV. Le modèle de DO utilisé dans les panneaux (d-f) est le Olympus MPlan ×4/0.1, et tous les MO de cette figure sont du même modèle personnalisé. (f) Illustration montrant l'absence de diaphonie inter-FOV sous les MO adjacents. Les images ont été prises sur une plaque fluorescente uniforme. Les cercles rouges indiquent les zones d'analyse utilisées pour comparer le contraste de l'image entre deux conditions; la condition de gauche montre la plaque fluorescente sous les deux MO, et la condition du côté droit montre la plaque de fluorescence sous un seul MO. (g) Test de la résolution optique de l'assemblage composé avec des billes de 0,51 µm. Courbes :ajustements gaussiens de points de données bruts. Les profils d'intensité de fluorescence sur ou hors axe ont été mesurés lorsque l'axe de la MO était aligné avec l'axe de la DO ou en dehors de l'axe de la DO (2 mm pour la DO de ×4 ou ×5, 3 mm pour le DO de ×2.5, et 4 mm pour le DO de ×2), respectivement. Crédit :Lumière :Science &Applications, doi:10.1038/s41377-019-0219-x
L'imagerie par microscopie à balayage laser à deux photons est couramment appliquée pour étudier l'activité neuronale à des résolutions cellulaires et subcellulaires dans le cerveau des mammifères. De telles études sont encore confinées à une seule région fonctionnelle du cerveau. Dans un récent rapport, Mengke Yang et ses collègues du Brain Research Instrument Innovation Center, Institut des neurosciences, Centre pour la neuroscience des systèmes et la technologie de fabrication avancée des systèmes optiques en Chine, L'Allemagne et le Royaume-Uni ont développé une nouvelle technique appelée explorateur in vitro multizone à deux photons en temps réel (MATRIEX). La méthode a permis à l'utilisateur de cibler plusieurs régions du cerveau fonctionnel avec un champ de vision (FOV) d'environ 200 µm de diamètre pour effectuer un Ca à deux photons. 2+ l'imagerie avec une résolution unicellulaire simultanément dans toutes les régions.
Yang et al. réalisé une imagerie fonctionnelle en temps réel des activités d'un seul neurone dans le cortex visuel primaire, cortex moteur primaire et région hippocampique CA1 pendant les états anesthésiés et éveillés chez la souris. La technique MATRIEX peut configurer de manière unique plusieurs FOV microscopiques à l'aide d'un seul appareil de balayage laser. Par conséquent, la technique peut être mise en œuvre en tant que module optique complémentaire dans le cadre d'un balayage monofaisceau conventionnel existant, microscopes à deux photons sans modifications supplémentaires. Le MATRIEX peut être appliqué pour explorer l'activité neuronale multizone in vivo pour la fonction des circuits neuronaux à l'échelle du cerveau avec une résolution unicellulaire.
La microscopie laser à deux photons est née dans les années 1990 pour devenir populaire parmi les neuroscientifiques intéressés par l'étude des structures et des fonctions neurales in vivo. Un avantage majeur de l'imagerie à deux photons et à trois photons pour les cerveaux vivants est la résolution optique obtenue à travers des tissus cérébraux densément marqués qui diffusent fortement la lumière, au cours de laquelle des pixels d'image coupés optiquement peuvent être numérisés et acquis avec une diaphonie minimale. Cependant, les avantages ont également causé des inconvénients importants à la méthode en empêchant la vue simultanée de deux objets à une distance spécifique. Les chercheurs avaient auparavant mis en œuvre de nombreuses stratégies pour étendre les limites, mais les méthodes étaient difficiles à mettre en œuvre dans les laboratoires de recherche en neurosciences. Néanmoins, il existe une demande de plus en plus élevée en neurosciences pour étudier les fonctions neuronales à l'échelle du cerveau avec une résolution unicellulaire in vivo.

GAUCHE :Schéma expérimental du système d'imagerie MATRIEX. Les deux objets 3D ronds dans le coin inférieur gauche sont les vues supérieure et inférieure de la chambre de la tête de souris utilisée pour l'imagerie in vivo. (Ti:Sa) : Laser pulsé ultrarapide Ti :Saphir ; PC :cellule de Pockels ; BE :extenseur de faisceau; SM1 et SM2 :miroirs de balayage x–y ; SL :objectif de balayage ; TL :lentille tubulaire; DM :miroir dichroïque; CL :lentille de collecte; PMT :tube photomultiplicateur; FAIRE :objectif sec; MO :objectifs miniaturisés. DROITE :Illustrations du grossissement à deux niveaux et du couplage multiaxes. Les images carrées sont de véritables images à deux photons prises de billes de 20 µm. Chaque cercle rouge indique un FOV. Crédit :Lumière :Science &Applications, doi:10.1038/s41377-019-0219-x
Dans une approche directe, les scientifiques peuvent placer deux microscopes au-dessus du même cerveau animal pour imager simultanément le cortex et le cervelet. Mais de tels efforts peuvent conduire à des augmentations substantielles de la complexité et des coûts. Les attentes élevées existantes en matière de performances et de faisabilité posent donc une question d'ingénierie très difficile sur la façon dont un seul système d'imagerie peut obtenir simultanément des images microscopiques en direct de plusieurs régions du cerveau in vivo. Pour répondre à la question, Yang et al. introduit une nouvelle méthode qui combine un grossissement à deux étages et un couplage optique multi-axes.
Ils ont réalisé la méthode en utilisant un objectif sec à faible grossissement (OD), avec plusieurs immersions dans l'eau, objectifs miniaturisés (OM) sous l'objectif sec. Les scientifiques ont placé chacun des MO à la position cible et à la profondeur souhaitées dans le tissu cérébral. L'équipe a utilisé le nouvel assemblage d'objets composés de la même manière que l'objectif de microscope immergé dans l'eau d'origine sans modifications supplémentaires du sous-système de numérisation et d'acquisition d'images.
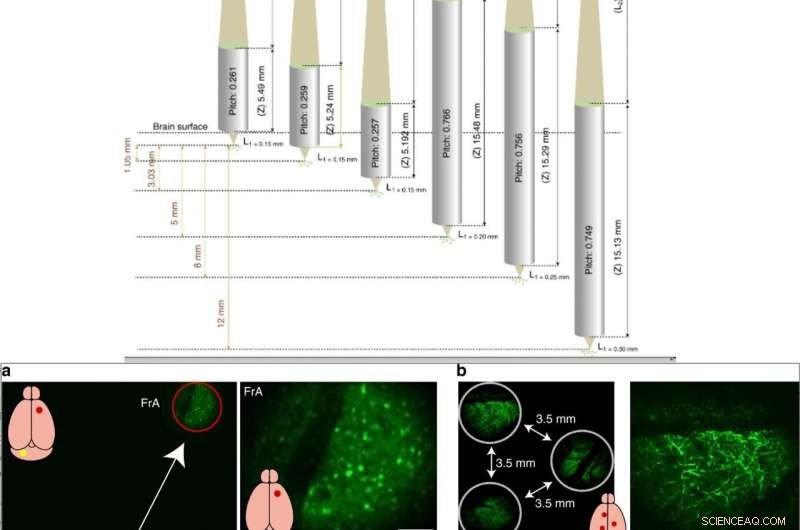
TOP :Configurer les MO avec différents paramètres pour cibler des plans objets à différentes profondeurs pour ensuite être conjugués sur le même plan image. Chaque cylindre gris représente une lentille avec une valeur de pas, distance de travail avant (L1), distance de travail arrière (L2) et longueur (Z). EN BAS :Démonstration d'imagerie MATRIEX :imagerie structurelle dans plusieurs zones cérébrales in vivo. a Image de gauche :une image plein cadre incluant deux FOV dans le cortex frontal d'association (FrA) et le cervelet. Les cercles rouges et jaunes indiquent deux FOV qui sont agrandis numériquement et affichés dans les images en haut à droite et en bas à droite. Une souris transgénique GAD67-GFP (avec les interneurones marqués à l'échelle du cerveau) a été utilisée. Deux MO (« version standard ») ont été placés à la même profondeur sous un DO (Mitutoyo ×2/0,055). b Exemple de configuration de trois FOV dans le cortex d'une souris transgénique Thy1-GFP (avec des neurones corticaux de la couche 5 spécifiquement marqués et avec des dendrites en touffes visibles près de la surface corticale). Trois MO (« version standard ») ont été placés à la même profondeur sous un DO (Olympus ×4/0.1). Crédit :Lumière :Science &Applications, doi:10.1038/s41377-019-0219-x
L'équipe de recherche a d'abord assemblé l'objectif composé MATRIEX. Pour ça, ils ont remplacé l'objectif de microscope à immersion dans l'eau conventionnel par un ensemble d'objectif composé personnalisé, à l'intérieur d'un microscope à balayage laser à deux photons équipé d'un dispositif de balayage conventionnel à faisceau unique. L'assemblage composé contenait plusieurs MO (objectifs miniaturisés) insérés à travers plusieurs craniotomies au cours desquelles les scientifiques ont collé une chambre en plastique imprimée en 3D sur le crâne du modèle de souris. La chambre a approximativement aligné les MO avec le même espace pour ajuster la position latérale et la profondeur. Yang et al. manipulé avec précision les MO individuels pour voir les objets sous tous les MO simultanément dans le même plan d'image.
Ils ont mis en œuvre la méthode MATRIEX selon deux principes; grossissement à deux niveaux et couplage multiaxes. Par exemple, en utilisant un grossissement à deux niveaux avec l'objectif sec (DO) seul, ils ont observé des billes de 20 µm sous forme de minuscules points flous tout en observant des cercles ronds à travers l'assemblage composé. Lors d'un couplage multiaxe, les scientifiques ont couplé un seul DO avec plusieurs MO sur le même plan image. À l'aide d'un simple balayage raster dans un seul cadre rectangulaire, l'équipe de recherche a acquis une image rectangulaire contenant plusieurs FOV circulaires (champ de vues) - où chaque FOV correspondait à un MO avec une diaphonie de pixels inter-FOV minimale.
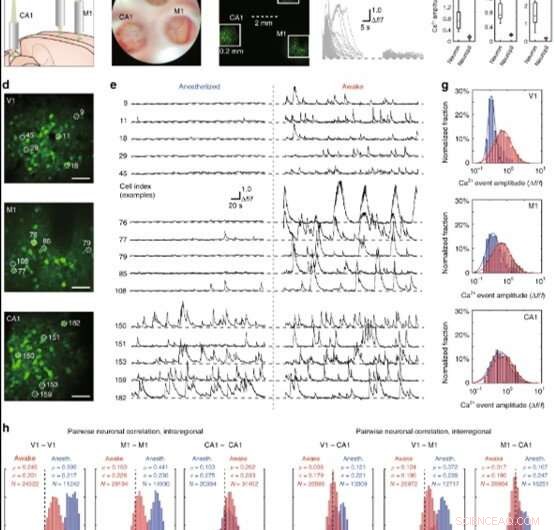
Démonstration d'imagerie MATRIEX :acquisition simultanée de modèles d'activité neuronale en direct dans V1, M1, et l'hippocampe CA1 chez la souris à l'état anesthésié ou à l'état éveillé. Les neurones ont été marqués par un indicateur fluorescent Ca2+ génétiquement codé, GCaMP6f (a) Illustration montrant le positionnement de trois MO sur le V1, Régions M1 et hippocampique CA1 dans un cerveau de souris modèle. (b) Une photographie prise à travers la lentille oculaire du microscope sous un éclairage à fond clair en lumière blanche, dans lequel trois FOV sont facilement visibles. La région supérieure est V1, la région en bas à gauche est CA1, et la région inférieure droite est M1. (c) Une image à deux photons, soit une moyenne de 100 images, acquises par simple balayage raster plein cadre avec un microscope à deux photons. Les cases blanches pleines montrent les trois parties de l'image qui sont agrandies dans le panneau (d). (d) FOV individuels agrandis numériquement montrant des neurones en V1, M1, et CA1, du haut jusqu'en bas. Barre d'échelle :40 μm. (e) Traces de signal Ca2+ time-lapse de cinq exemples de cellules de chaque région, avec chacun étiqueté par l'index de cellule. Les enregistrements de la même cellule chez le même animal à l'état anesthésié (côté gauche) et à l'état éveillé (côté droit) sont présentés. (f) Gauche :traces montrant des événements de signal Ca2+ individuels (séparés à partir de chaque heure d'apparition et superposés) à partir de cellules d'exemple sélectionnées au hasard. Milieu :traces de signal Ca2+ de chacune des zones neuropiles qui sont directement adjacentes à chacune des cellules de l'exemple. À droite :trois boîtes à moustaches comparant l'amplitude de l'événement du signal Ca2+ neuronal à l'amplitude du signal Ca2+ du neuropile adjacent du neurone ; test de somme des rangs de Wilcoxon apparié, ***P < 0,001. (g) Ajustement log-normal des histogrammes de distribution de l'amplitude de l'événement Ca2+ spontané pour les données regroupées de tous les animaux. Les barres rouges et la courbe ajustée montrent la distribution des données enregistrées à l'état éveillé, et les barres bleues et la courbe ajustée montrent la distribution des données enregistrées à l'état anesthésié. (h) Corrélation de l'activité neuronale par paire (coefficients de corrélation de Pearson) pour les données regroupées de tous les animaux. Les barres rouges montrent la répartition des données enregistrées à l'état éveillé, et les barres bleues montrent la distribution des données enregistrées à l'état anesthésié. Crédit :Lumière :Science &Applications, doi:10.1038/s41377-019-0219-x
Les scientifiques ont crédité le grossissement de l'ouverture numérique (NA) pour permettre une meilleure résolution avec l'assemblage composé. Les lentilles associées étaient également flexibles et conçues sur mesure pour une production en série à faible coût afin de faciliter la conception expérimentale. La principale caractéristique de MATRIEX était sa capacité à imager plusieurs objets simultanément à de grands intervalles de profondeur. Pour le souligner, Yang et al. conçu différents MO avec divers paramètres, en les plaçant à une profondeur spécifique où les plans objets correspondants se conjuguent sur le même axe. En pratique, l'équipe de recherche a compensé les écarts mineurs entre la profondeur de l'objet souhaitée et réelle en ajustant les MO individuellement le long de chacun des axes z.
Typiquement, sous le DO (objectif sec) la taille latérale maximale de la zone cible est limitée par la taille maximale du champ de balayage. Par exemple, à l'aide d'un DO avec un grossissement 2x et une zone cible de 12 mm de diamètre, les scientifiques peuvent imager un cerveau entier de souris adulte. Dans cette étude, Yang et al. simultanément imagé le cortex d'association frontale et le cervelet de la souris. En pratique, un objectif d'air 4x était adapté pour obtenir une meilleure résolution pour observer les structures de dendrites fines.

Imagerie calcique simultanée dans le V1, Régions M1 et CA1 utilisant MATRIEX pendant les états anesthésiés et éveillés chez la souris. Voir le film complet sur Crédit : Lumière :Science et applications, doi:10.1038/s41377-019-0219-x
Comme preuve de principe, l'équipe de recherche a utilisé MATRIEX pour effectuer simultanément deux photons Ca 2+ imagerie de neurones marqués par fluorescence dans le cortex visuel primaire (région V1), cortex moteur primaire (région M1) et région hippocampique CA1 de souris. Dans la configuration des trois MO, les scientifiques ont placé deux MO adaptés aux régions V1 et M1, directement au-dessus du cortex et inséré un MO dans la région hippocampique CA1 après avoir enlevé chirurgicalement un tissu cortical. L'équipe a ensuite conçu les lentilles pour les plans objets correspondant à V1, M1 et CA1 pour la conjugaison sur le même plan image. A l'aide d'un microscope biphotonique équipé d'un scanner résonant 12 kHz, les scientifiques ont scanné l'image complète pour observer trois FOV et leurs cellules individuelles après avoir agrandi les trois sections différentes pour résoudre des neurones uniques. Ensuite, ils ont noté la puissance laser à répartir entre plusieurs FOV.
Alors que Yang et al. aurait pu obtenir ces résultats en utilisant l'imagerie conventionnelle à FOV unique dans une seule région du cerveau, la technique MATRIEX leur a fourni des données au-delà de celles offertes avec les techniques d'imagerie à FOV unique. Pris ensemble, ces résultats ont permis une distribution et une transformation très inhomogènes des schémas d'activité spontanée de l'état anesthésié à l'état éveillé chez la souris, couvrant un niveau de circuit à l'échelle du cerveau à une résolution monocellulaire.
De cette façon, Menge Yang et ses collaborateurs ont développé la technique MATRIEX basée sur le principe du grossissement à deux étages et du couplage optique multiaxe. Ils ont mené simultanément deux photons Ca 2+ l'imagerie dans les activités de la population neuronale à différentes profondeurs dans diverses régions (V1, M1 et CA1) chez des souris anesthésiées et éveillées avec une résolution unicellulaire. Surtout, tout microscope biphotonique classique peut être transformé en un microscope MATRIEX, tout en préservant toutes les fonctionnalités d'origine. La clé de la transformation repose sur la conception d'un ensemble objectif composé. Les chercheurs peuvent utiliser différentes, MO soigneusement conçus pour s'adapter à diverses régions du cerveau avec une compatibilité à 100 pour cent entre la technique MATRIEX et la microscopie conventionnelle. L'équipe de recherche s'attend à ce que la technique MATRIEX progresse considérablement en trois dimensions, dynamique des circuits neuronaux à l'échelle du cerveau à une résolution unicellulaire.
© 2019 Réseau Science X