
Invite montrée aux cliniciens pour les annotations. Crédit :IBM
Ces derniers temps ont été témoins de progrès significatifs dans la compréhension du langage naturel par l'IA, telles que la traduction automatique et la réponse aux questions. Une raison essentielle derrière ces développements est la création d'ensembles de données, qui utilisent des modèles d'apprentissage automatique pour apprendre et effectuer une tâche spécifique. La construction de tels ensembles de données dans le domaine ouvert se compose souvent de texte provenant d'articles de presse. Ceci est généralement suivi par la collecte d'annotations humaines à partir de plateformes de crowdsourcing telles que Crowdflower, ou Amazon Mechanical Turk.
Cependant, le langage utilisé dans des domaines spécialisés comme la médecine est totalement différent. Le vocabulaire utilisé par un médecin lorsqu'il rédige une note clinique est assez différent des mots d'un article de presse. Ainsi, les tâches linguistiques dans ces domaines à forte intensité de connaissances ne peuvent pas être externalisées car de telles annotations exigent une expertise du domaine. Cependant, la collecte d'annotations auprès d'experts du domaine est également très coûteuse. De plus, les données cliniques sont sensibles à la confidentialité et ne peuvent donc pas être partagées facilement. Ces obstacles ont inhibé la contribution des ensembles de données linguistiques dans le domaine médical. En raison de ces défis, la validation d'algorithmes performants du domaine ouvert sur des données cliniques n'a pas encore été étudiée.
Afin de combler ces lacunes, nous avons travaillé avec le Massachusetts Institute of Technology pour construire MedNLI, un jeu de données annoté par des médecins, effectuer une tâche d'inférence en langage naturel (NLI) et fondée sur les antécédents médicaux des patients. Plus important encore, nous le mettons à la disposition du public pour que les chercheurs fassent progresser le traitement du langage naturel en médecine.
Nous avons travaillé avec les laboratoires de recherche du MIT Critical Data pour construire un ensemble de données pour l'inférence du langage naturel en médecine. Nous avons utilisé des notes cliniques de leur base de données "Medical Information Mart for Intensive Care" (MIMIC), qui est sans doute la plus grande base de données publiquement disponible sur les dossiers des patients. Les cliniciens de notre équipe ont suggéré que les antécédents médicaux d'un patient contiennent des informations vitales à partir desquelles des inférences utiles peuvent être tirées. Par conséquent, nous avons extrait les antécédents médicaux des notes cliniques dans MIMIC et présenté une phrase de ces antécédents comme prémisse à un clinicien. Il leur a ensuite été demandé d'utiliser leur expertise médicale et de générer trois phrases :une phrase qui était bien vraie pour le patient, étant donné la prémisse; une phrase qui était définitivement fausse, et enfin une phrase qui pourrait être vraie.
En quelques mois, nous avons échantillonné au hasard 4, 683 de ces locaux et a travaillé avec quatre cliniciens pour construire MedNLI, un jeu de données de 14, 049 paires prémisse-hypothèse. Dans le domaine ouvert, d'autres exemples d'ensembles de données construits de la même manière incluent l'ensemble de données Stanford Natural Language Inference, qui a été organisée avec l'aide de 2, 500 travailleurs sur Amazon Mechanical Turk et se compose de 0,5 million de paires prémisse-hypothèse où les phrases prémisses ont été tirées des légendes des photos Flickr. MultiNLI en est un autre et se compose de textes de prémisse de genres spécifiques tels que la fiction, blog, conversations téléphoniques, etc.
Le Dr Leo Anthony Celi (chercheur principal pour MIMIC) et le Dr Alistair Johnson (chercheur scientifique) du MIT Critical Data ont travaillé avec nous pour rendre MedNLI accessible au public. Ils ont créé le référentiel de données dérivées MIMIC, auquel MedNLI a agi en tant que première contribution à l'ensemble de données de traitement du langage naturel. Tout chercheur ayant accès à MIMIC peut également télécharger MedNLI à partir de ce référentiel.
Bien que de taille modeste par rapport aux jeux de données du domaine ouvert, MedNLI est suffisamment grand pour informer les chercheurs alors qu'ils développent de nouveaux modèles d'apprentissage automatique pour l'inférence du langage en médecine. Plus important encore, il présente des défis intéressants qui appellent des idées innovantes. Considérez quelques exemples de MedNLI :
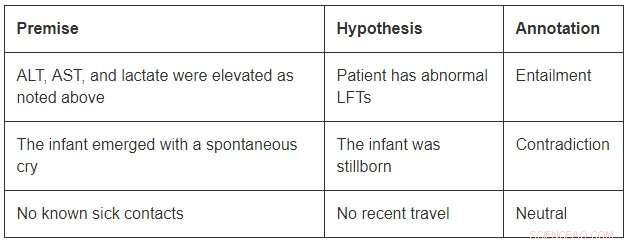
Afin de conclure l'implication dans le premier exemple, on devrait pouvoir développer les abréviations ALT, AST, et LFT ; comprendre qu'ils sont liés; et conclure en outre qu'une mesure élevée est anormale. Le deuxième exemple dépeint une inférence subtile de conclure que l'émergence d'un nourrisson est une description de sa naissance. Finalement, le dernier exemple montre comment la connaissance commune du monde est utilisée pour dériver des inférences.
Les algorithmes d'apprentissage en profondeur à la pointe de la technologie peuvent être très performants sur les tâches linguistiques, car ils ont le potentiel de devenir très efficaces pour apprendre une cartographie précise des entrées aux sorties. Ainsi, la formation sur un grand ensemble de données annoté à l'aide d'annotations participatives est souvent la recette du succès. Cependant, ils manquent encore de capacités de généralisation dans des conditions différentes de celles rencontrées lors de la formation. C'est encore plus difficile dans des domaines spécialisés et à forte intensité de connaissances tels que la médecine, où les données de formation sont limitées et la langue est beaucoup plus nuancée.
Finalement, bien que de grands progrès aient été accomplis dans l'apprentissage d'une tâche linguistique de bout en bout, il existe toujours un besoin de techniques supplémentaires qui peuvent incorporer des bases de connaissances organisées par des experts dans ces modèles. Par exemple, SNOMED-CT est une terminologie médicale organisée par des experts avec plus de 300 000 concepts et relations entre les termes de son ensemble de données. Au sein de MedNLI, nous avons apporté des modifications simples aux architectures de réseaux de neurones profonds existantes pour infuser des connaissances à partir de bases de connaissances telles que SNOMED-CT. Cependant, une grande quantité de connaissances reste encore inexploitée.
Nous espérons que MedNLI ouvrira de nouvelles directions de recherche dans la communauté du traitement du langage naturel.
Cette histoire est republiée avec l'aimable autorisation d'IBM Research. Lisez l'histoire originale ici.