Les lipides sont une classe de biomolécules qui jouent un rôle important dans de nombreux processus cellulaires. Les analyses qui cherchent à caractériser tous les lipides d'un échantillon (appelées lipidomique) sont essentielles à l'étude des systèmes biologiques complexes.
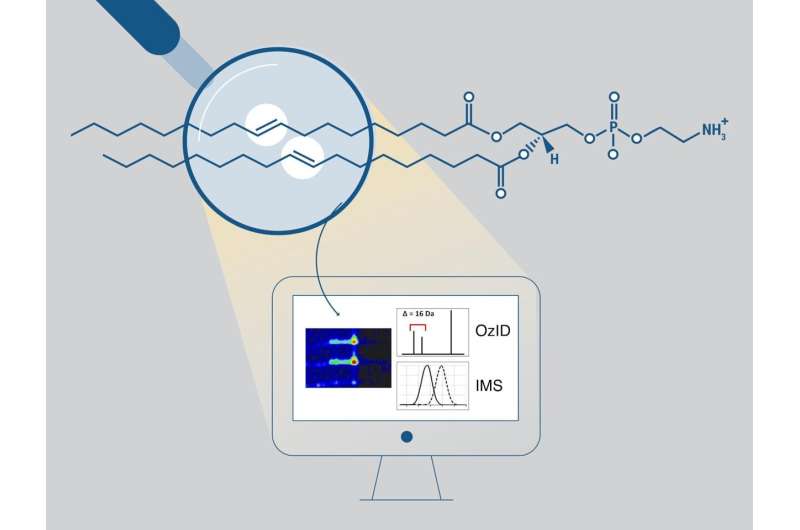
Un défi important en lipidomique consiste à relier la variété des structures des lipides à leurs fonctions biologiques. La position des doubles liaisons au sein des chaînes d’acides gras est particulièrement importante. En effet, ils peuvent affecter les propriétés physiques des membranes cellulaires et moduler les voies de signalisation cellulaire.
Ces informations ne sont pas systématiquement mesurées dans les études lipidomiques car elles nécessitent une configuration expérimentale compliquée qui produit des données complexes. Ainsi, les scientifiques du Pacific Northwest National Laboratory (PNNL) ont développé un flux de travail rationalisé pour déterminer les positions des doubles liaisons. Ce flux de travail utilise à la fois des approches d'automatisation et d'apprentissage automatique.
Leur nouvelle méthode, LipidOz, rationalise l'analyse des données pour déterminer les positions des doubles liaisons. En abordant cet élément clé de l’analyse des lipides, LipidOz offre aux chercheurs une méthode plus efficace et plus précise pour la caractérisation des lipides. L'étude est publiée dans la revue Communications Chemistry .
L'identification sans ambiguïté des lipides est compliquée par la présence de parties moléculaires ayant la même formule chimique mais des configurations physiques différentes. Plus précisément, les différences entre ces parties moléculaires incluent la longueur de la chaîne acyle gras, la position numérotée de manière stéréospécifique (sn) et la position/stéréochimie des doubles liaisons.
Les analyses conventionnelles peuvent déterminer la longueur des chaînes d'acyles gras, le nombre de doubles liaisons et, dans certains cas, la position sn mais pas les positions des doubles liaisons carbone-carbone. Les positions de ces doubles liaisons peuvent être déterminées avec plus de confiance à l'aide d'une réaction d'oxydation en phase gazeuse appelée dissociation induite par l'ozone (OzID), qui produit des fragments caractéristiques.
Cependant, l’analyse des données obtenues à partir de cette réaction est complexe et répétitive, et il existe un manque de support logiciel. L'outil Python open source, LipidOz, détermine et attribue automatiquement les positions des doubles liaisons des lipides en combinant des approches traditionnelles d'automatisation et d'apprentissage en profondeur. De nouvelles recherches démontrent cette capacité pour les mélanges lipidiques standards et les extraits lipidiques complexes, permettant une application pratique d'OzID pour de futures études lipidomiques.
Plus d'informations : Dylan H. Ross et al, LipidOz permet l'élucidation automatisée des positions des doubles liaisons lipidiques carbone-carbone à partir des données de spectrométrie de masse à dissociation induite par l'ozone, Communications Chemistry (2023). DOI : 10.1038/s42004-023-00867-9
Informations sur le journal : Chimie des communications
Fourni par le Laboratoire national du Nord-Ouest du Pacifique