Crédit :Wiley
L'ajout et l'élimination de groupes méthyle sur l'ADN joue un rôle important dans la régulation des gènes. Afin d'étudier plus précisément ces mécanismes, une équipe allemande a développé une nouvelle méthode par laquelle des sites de méthylation spécifiques peuvent être bloqués puis débloqués à un moment précis par irradiation avec de la lumière (photocaging). Comme indiqué dans le journal Angewandte Chemie , le régent requis est produit par voie enzymatique, in situ.
Bien qu'ils semblent très différents et remplissent des fonctions complètement différentes, toutes les cellules de notre corps ont un ADN identique. Cependant, ils n'utilisent pas les mêmes gènes. Certains gènes sont activés et d'autres désactivés, selon le type de cellule et le moment. Les « commutateurs » sont des changements chimiques dans les éléments constitutifs de l'ADN. Ces changements sont appelés modifications épigénétiques. Un mécanisme de régulation important est la méthylation et la déméthylation, signifiant la fixation et l'élimination d'un groupe méthyle (-CH
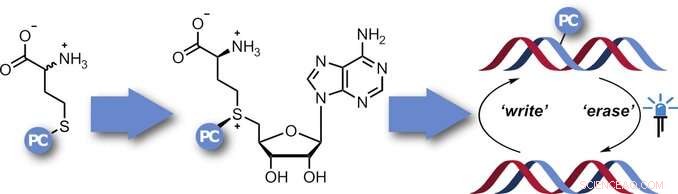
Afin d'étudier de plus près le but et la fonction de cette régulation et de déterminer les schémas de méthylation, il serait utile d'avoir des "outils" pour inhiber spécifiquement la méthylation à des endroits ciblés, puis lever l'inhibition à un moment défini. À cette fin, une équipe dirigée par Andrea Rentmeister a choisi d'utiliser une méthode connue sous le nom de photocaging. Dans cette méthode, une "photocage" est une molécule qui se désagrège lors de l'irradiation, tel qu'un groupe 2-nitrobenzyle. La cage bloque d'abord l'emplacement cible, puis une irradiation ciblée avec de la lumière agit comme un « interrupteur » pour lever le blocus.
L'idée était d'équiper les analogues d'AdoMet d'une photocage qui est ensuite transférée sur les sites de méthylation. Cependant, Les analogues d'AdoMet se décomposent dans des solutions aqueuses et ne peuvent pas pénétrer dans les cellules. Par conséquent, l'équipe de l'université de Münster voulait les produire in situ. Dans le corps, AdoMet est produit à partir de l'acide aminé méthionine par l'action de l'enzyme, méthionine adénosyl transférase (MAT). La synthèse des analogues d'AdoMet nécessite de la méthionine avec une photocage de nitrobenzyle attachée et un MAT qui peut utiliser un tel substrat altéré. A partir d'une enzyme MAT d'un organisme unicellulaire (Cryptosporidium hominis), les chercheurs ont pu modifier soigneusement des acides aminés spécifiques dans l'enzyme pour augmenter la taille de sa cavité de liaison hydrophobe afin qu'elle puisse contenir le groupe nitrobenzyle. Une analyse de la structure cristalline a montré que l'analogue ADoMet est lié dans la cavité de ce MAT de photocaging (PC-MAT). Sur la base de ces informations, l'équipe a également produit un deuxième PC-MAT basé sur une enzyme MAT thermostable de l'archéon Methanocaldococcus jannaschii.
Ces deux PC-MAT sont compatibles avec les ADN et ARN MTases et ont permis d'attacher des photocages à tous les sites de méthylation naturels d'un ADN plasmidique. L'irradiation avec la lumière a supprimé le blocus.