Une équipe de chercheurs a récemment démontré l'utilité d'utiliser un système de criblage déjà établi pour sonder les interactions entre les petites molécules et l'ARN. Leur étude est publiée dans Communications Chemistry. .
L'équipe était dirigée par le professeur Hirohide Saito (Département des frontières des sciences de la vie, CiRA), le Dr Kaoru R. Komatsu (ancien doctorant au CiRA), le professeur agrégé Kazumitsu Onizuka et le professeur Fumi Nagatsugi (Institut de recherche multidisciplinaire). pour les matériaux avancés, Université du Tohoku).
Des récents vaccins à ARNm du SRAS-CoV-2 pour lutter contre la pandémie de COVID-19 au risdiplam, un modificateur d'épissage de l'ARN approuvé par la Food and Drug Administration des États-Unis pour l'amyotrophie spinale, le mot « ARN » est entré dans le langage courant car il représente à la fois de nouvelles classes d'agents thérapeutiques et de cibles médicamenteuses.
Cependant, notre compréhension de la manière dont diverses séquences d'ARN et, par conséquent, structures dictent les interactions avec de petites molécules telles que des composés médicamenteux ou de grandes biomolécules telles que des protéines reste incomplète.
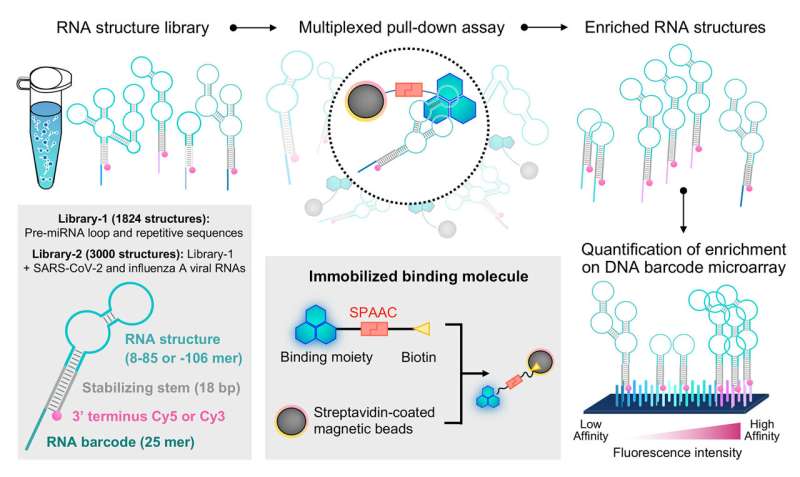
Dans une étude précédente, le professeur Saito et son équipe de recherche ont conçu un système appelé profilage d'éléments d'ARN pliés avec bibliothèque de structures, ou FOREST, pour examiner les détails moléculaires de la façon dont l'ARN interagit avec les protéines connues de liaison à l'ARN.
Pour cette nouvelle étude, grâce à une collaboration avec des chercheurs de l'Université de Tohoku, l'équipe de recherche commune a illustré comment FOREST peut être utilisé pour analyser les petites interactions moléculaires avec l'ARN.
L'équipe de recherche a d'abord validé l'applicabilité de l'approche FOREST aux petites molécules en examinant comment une bibliothèque de structures d'ARN interagit avec de petits interacteurs moléculaires d'ARN connus :les dérivés G-clamp et thiazine orange (TO).
Les chercheurs ont isolé les structures d’ARN liées au G-clamp à partir d’une bibliothèque comprenant plus de 1 800 séquences d’ARN dérivées de pré-miARN humains et d’autres séquences répétitives et de contrôle. Parce que ces structures d’ARN sont liées à un colorant fluorescent et à un code-barres d’ARN, elles pourraient être facilement décodées et quantifiées par une puce à ADN avec des séquences complémentaires aux codes-barres d’ARN pour fournir une analyse quantitative de la façon dont les structures d’ARN interagissent avec la petite molécule spécifique d’intérêt.
Comme prévu, ils ont identifié une liaison préférentielle aux séquences d’ARN simple et double brin contenant de la guanosine (G) (ARNsb et ARNdb, respectivement) par G-clamp. Dans la bibliothèque de structures d'ARN, l'équipe de recherche a choisi des séquences montrant une liaison à affinité élevée, intermédiaire ou faible au G-clamp pour validation par une expérience indépendante basée sur la fluorescence qui mesure directement les constantes de dissociation apparentes des interactions individuelles.
Remarquablement, ils ont observé une bonne corrélation entre l'affinité de liaison relative estimée par l'approche FOREST et les constantes de dissociation apparentes déterminées par le test de liaison basé sur la fluorescence, indiquant la grande robustesse de cette méthode pour quantifier les interactions petites molécules-ARN.
De plus, en mutant une structure spécifique de boucle d'ARN avec plusieurs guanosines à différents endroits, ils ont découvert que la G-clamp n'interagit pas de la même manière avec toutes les guanosines de la boucle, mais qu'un contexte structurel supplémentaire peut influencer l'interaction.
À l’inverse, les dérivés TO sont des sondes couramment utilisées pour les analyses de déplacement d’indicateur fluorescent (FID). Les chercheurs ont ensuite mélangé TO et TO-3 séparément avec une bibliothèque de structures d'ARN élargie contenant des séquences supplémentaires dérivées des ARN du SRAS-CoV-2 et du virus de la grippe A afin de mieux caractériser les dérivés de TO pour les mesures d'ARN.
Comme prévu, alors qu'il n'y avait aucune corrélation entre les profils de liaison des structures d'ARN interagissant avec les dérivés G-clamp et TO, TO et TO-3 partageaient des profils de liaison similaires avec quelques distinctions mineures.
Autres comparaisons entre TO-N3 , À-N3 -2, et TO-3-N3 ont révélé que la position du lieur a une influence modeste sur les profils de liaison à l'ARN. De plus, sur la base de ces profils de liaison, les chercheurs ont discerné certaines préférences de position de base et de boucle que les dérivés TO ont lorsqu'ils interagissent avec les structures d'ARN.
L'équipe de recherche a en outre étendu son analyse des dérivés de TO en comparant les affinités de liaison relatives déterminées par FOREST aux constantes de dissociation apparentes mesurées pour les indicateurs nucléiques fluorescents disponibles dans le commerce, TO-PRO-1 et TO-PRO-3, par le test de liaison basé sur la fluorescence. .
Grâce à cette analyse, ils ont révélé que même si TO-N3 -2 peut décrire plus précisément le profil de liaison de TO-PRO1 par rapport à TO-3-N3 , tous deux TO-N3 -2 et TO-3-N3 simule à peu près aussi bien TO-PRO-3, fournissant ainsi des informations structurelles cruciales pour améliorer les appariements d'ARN cible et d'indicateurs fluorescents pour les tests FID.
À l’aide des profils de liaison déterminés pour les dérivés TO, l’équipe de recherche a sélectionné des combinaisons d’indicateurs fluorescents (TO-PRO-1 ou TO-PRO-3) et de séquences de pré-miARN précédemment démontrées comme étant dérégulées dans les tumeurs présentant des affinités de liaison intermédiaires pour les tests FID.
En guise de preuve de concept, les chercheurs ont examiné une bibliothèque chimique disponible dans le commerce contenant 118 composés pour identifier de petites molécules capables d'interagir avec les pré-miARN associés à la maladie. Grâce à cet effort, ils ont identifié la baicaléine (Bai), la myricétine (Myr), le chlorure de chélérythrine (Che) et l'AS 602801 (AS) comme composés candidats. Alors que Myr et Che sont connus pour lier l'ADN et l'ARN, il s'agissait de la première démonstration de l'AS en tant qu'interacteur d'acide nucléique.
Notamment, les chercheurs ont observé des résultats différents lorsque TO-PRO-1 ou TO-PRO-3 étaient utilisés comme indicateur fluorescent, suggérant ainsi que des indicateurs distincts devraient être utilisés pour éviter les identifications faussement positives et négatives. Un examen plus approfondi de l'AS a confirmé sa liaison à plusieurs pré-miARN humains d'intérêt, mais les chercheurs ont également remarqué que le composé présentait de fortes propriétés lumineuses lors de son interaction avec l'ARN.
L'examen structurel du composé suggère qu'il contient une fraction chimique probablement responsable des propriétés d'éclairage, ce qui en fait un composé intéressant pour un développement ultérieur en un nouvel interacteur d'ARN et une sonde fluorescente.
Dans cette étude, l'effort de recherche conjoint illustre une fois de plus l'applicabilité de la méthodologie FOREST, non seulement pour inspecter les interactions ARN-protéine, mais également pour étudier les moindres détails des interactions entre l'ARN et les petites molécules.
Compte tenu du vaste potentiel de l’ARN en tant que nouvelle approche thérapeutique dans la médecine de nouvelle génération, la capacité de caractériser systémiquement les interactions petites molécules-ARN à grande échelle aura d’énormes impacts sur la recherche fondamentale sur l’ARN et sur la traduction de ces connaissances en thérapies.
Plus d'informations : Ryosuke Nagasawa et al, Analyse à grande échelle des interactions petites molécules-ARN à l'aide de bibliothèques de structures d'ARN multiplexées, Chimie des communications (2024). DOI :10.1038/s42004-024-01181-8
Informations sur le journal : Chimie des communications
Fourni par l'Université de Kyoto