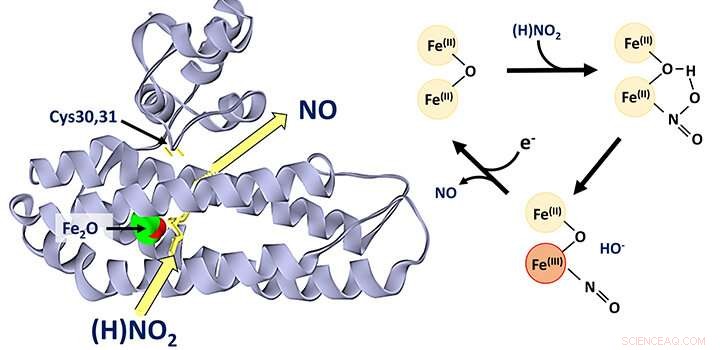
Résumé graphique. Source :Journal of the American Chemical Society (2022). DOI :10.1021/jacs.1c12407
Des chercheurs de l'Université d'East Anglia ont identifié une nouvelle classe d'enzymes impliquées dans la gestion du stress chez les bactéries.
Tous les organismes, des humains aux bactéries, doivent être capables de répondre à un large éventail de stress résultant de changements dans leur environnement.
Parmi ceux-ci, les plus courants sont les stress oxydatifs et nitrosatifs, qui se produisent lorsqu'un organisme est exposé à des concentrations élevées d'oxygène de réaction ou d'espèces azotées réactives, respectivement. Lorsque cela se produit, les composants fragiles de la cellule sont endommagés, entraînant une perte de fonction et, dans certains cas, la mort cellulaire.
Sans surprise, les organismes ont développé une multitude de systèmes de réponse au stress qui détectent et atténuent des stress particuliers.
Les amas fer-soufre, composés de fer et de soufre inorganique, se trouvent dans tous les types de cellules où ils jouent un rôle essentiel dans un large éventail de processus cellulaires. Parce qu'ils sont si réactifs, ils sont souvent les premiers composants cellulaires à être endommagés dans des conditions de stress.
On pense généralement que la protéine di-fer YtfE, largement présente dans les bactéries, fonctionne directement dans la réparation des amas fer-soufre qui ont été endommagés dans des conditions de stress. Cette activité a été proposée de manière variable pour impliquer le don de fer pour la reconstruction des amas fer-soufre, ou l'élimination de l'oxyde nitrique (NO) des amas endommagés.
Récemment, de nouvelles preuves sont apparues à partir d'études sur la fonction YtfE dans les cellules, suggérant que son activité est associée à une augmentation, et non à une diminution, de la concentration de NO. Cela a incité les chercheurs de l'École de chimie à réexaminer la fonction de YtfE.
L'équipe, dirigée par le Dr Jason Crack et le professeur Nick Le Brun, et impliquant le laboratoire du Dr Fraser MacMillan, ainsi que des chercheurs de l'Université de Birmingham et de l'Université de Sheffield, a découvert que YtfE est un nouveau type d'enzyme nitrite réductase qui produit NON.
Ils ont montré que YtfE n'éliminait pas efficacement le NO des amas de fer-soufre endommagés, et qu'il n'était pas non plus un donneur efficace de fer pour l'assemblage des amas.
La production catalysée par YtfE de NO toxique à partir de nitrite (NO2 - ) peut sembler étrange, mais YtfE est co-régulé avec une autre enzyme, appelée Hcp, qui a pour fonction de détoxifier le NO (via sa réduction en protoxyde d'azote, N2 O).
La voie de détoxification couplée YtfE/Hcp représente un moyen efficace par lequel la cellule traite les niveaux toxiques de nitrite qui peuvent se produire dans des conditions anaérobies.
L'équipe a utilisé une gamme d'approches, y compris la spectroscopie génétique in vivo et in vitro des protéines et la spectrométrie de masse, pour obtenir des informations détaillées sur le mécanisme par lequel YtfE génère du NO à partir de nitrite.
Les travaux clarifient le rôle d'une protéine qui a longtemps intrigué les chercheurs, démontrant que YtfE est un acteur clé pour la gestion du stress dans des conditions de respiration anaérobie, telles que celles trouvées dans l'intestin humain ou lors de l'infection d'un hôte humain.
Les travaux sont publiés cette semaine dans le Journal of the American Chemical Society . Les chercheurs suggèrent que les clusters [FeS] agissent comme des fusibles biologiques dans de nombreuses protéines impliquées dans la réponse immunitaire