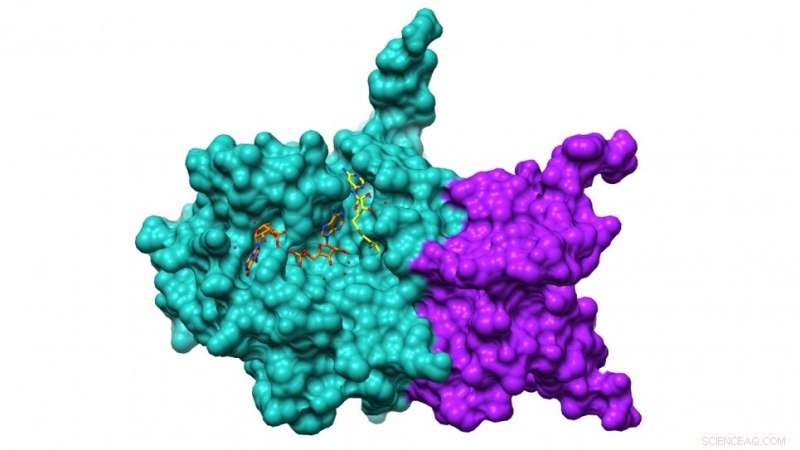
Surface Nsp10/16 avec des ligands. Les chercheurs ont développé un pipeline pour connecter les superordinateurs ALCF aux expériences APS afin de permettre l'analyse en temps réel des protéines COVID-19, ouvrant la voie à l'élucidation d'importantes dynamiques structurelles des protéines du coronavirus. Crédit :Mateusz Wilamowski, Université de Chicago, Centre de génomique structurelle des maladies infectieuses ; George Minasov, Université du nord-ouest, Centre de génomique structurale des maladies infectieuses
Les chercheurs d'Argonne ont développé un pipeline entre les superordinateurs ALCF et les expériences Advanced Photon Source pour permettre une analyse à la demande de la structure cristalline des protéines COVID-19.
Comme le coronavirus SARS-CoV-2 et sa maladie associée, COVID-19 [FEMININE, développé et répandu à travers le pays et la planète, le laboratoire national d'Argonne du département américain de l'Énergie (DOE) s'est joint à la lutte mondiale en commençant à travailler pour mieux comprendre et traiter la pandémie. Plusieurs de ces axes de recherche ont été lancés à l'Argonne Leadership Computing Facility, une installation utilisateur du DOE Office of Science, tirer parti de ses ressources scientifiques considérables; l'une de ces lignées a analysé la structure cristalline d'un complexe protéique associé au coronavirus.
La clé pour comprendre le coronavirus est de démêler sa structure. À cette fin, Les chercheurs d'Argonne ont utilisé le supercalculateur Theta de l'ALCF pour analyser des images cristallographiques d'un complexe protéique associé au SARS-CoV-2. Les images proviennent de l'Advanced Photon Source (APS) d'Argonne, une installation utilisateur du DOE Office of Science, suite à des expériences utilisant une technique connue sous le nom de cristallographie synchrotron en série conçue pour élucider la chimie complexe des protéines virales.
Des expériences de cristallographie synchrotron en série utilisent des rayons X à haute intensité pour révéler les structures de grosses molécules en utilisant uniquement des doses de rayonnement fractionnées par rapport aux exigences des techniques cristallographiques traditionnelles. Par conséquent, la cristallographie synchrotron en série permet aux chercheurs d'imager des dizaines de milliers de cristaux microscopiques, avec des durées d'exposition très courtes pour chaque échantillon individuel. La grande vitesse de la technique conduit à la génération d'une vaste gamme de données, dont la complexité et la densité nécessitent des analyses sophistiquées et exigeantes en termes de calcul.
Les systèmes massivement parallèles comme Theta sont uniques dans leur capacité à répondre aux exigences posées par la cristallographie synchrotron en série pour la rapidité, traitement à la volée. L'activation de Theta pour une utilisation dans le traitement à la volée est un pipeline de données construit autour du superordinateur. Ce pipeline automatise l'acquisition de données, une analyse, curation, et la visualisation, transporter les résultats vers un référentiel à partir duquel les métadonnées peuvent être extraites pour publication.
Le pipeline génère de gros lots d'images à un taux élevé, avec des transferts de données atteignant des vitesses de 700 mégaoctets par seconde grâce à Globus, un service de gestion de données géré par l'Université de Chicago.
« Le déploiement de ce pipeline entre l'APS et l'ALCF pour l'analyse à la demande a été un énorme succès, " a déclaré Ryan Chard, un informaticien à Argonne qui dirige les efforts de traitement d'images. "Nous avons atteint un taux de traitement allant jusqu'à 95 images par seconde." Cette vitesse élevée a permis de fournir un retour d'information instantané aux expérimentateurs de l'APS.
Le pipeline commence avec Globus transférant les images de l'APS vers le système Theta. Les images sont ensuite analysées et traitées à l'aide de FuncX, un système de calcul de fonction en tant que service qui organise l'envoi de tâches individuelles aux nœuds de calcul disponibles. FuncX est ensuite également utilisé pour extraire des métadonnées sur les hits, identifier les diffractions cristallines, et générer des visualisations illustrant à la fois l'emplacement de l'échantillon et celui des hits. Après cela, les données brutes, métadonnées, et les visualisations associées sont publiées sur un portail hébergé à l'ALCF, où ils sont indexés et rendus consultables pour une réutilisation.
Dix-neuf échantillons ont été analysés sur près de 1, 500 flux au cours de trois passages de dix heures sur le faisceau APS, au cours de laquelle plus de 700, 000 images ont été traitées sur Theta. Les données résultantes ont été publiées sur le portail de données et utilisées pour affiner davantage le travail expérimental et les configurations. L'orchestration nécessaire pour faciliter la recherche à cette échelle est rendue possible par les services d'automatisation des données de recherche actuellement en cours de développement sur la plateforme Globus, et soutenu par le transfert de fichiers fiable, et des capacités de partage de données sécurisées qui sont déjà largement utilisées sur les lignes de lumière APS. Ces capacités continueront de s'améliorer avec les futures améliorations prévues des lignes de lumière APS, les supercalculateurs ALCF, Globus, et le réseau APS vers ALCF. La prochaine mise à niveau APS, qui permettra aux chercheurs de voir des choses à l'échelle qu'ils n'ont jamais vues auparavant avec des rayons X basés sur des anneaux de stockage, augmentera les débits de données par ordre de grandeur. La combinaison de ces capacités de l'ALCF et de la mise à niveau APS améliorera considérablement la découverte scientifique.
« La pertinence biologique croissante des expériences de cristallographie synchrotron en série incite les chercheurs à préparer un certain nombre d'autres expériences dans les semaines à venir, " a déclaré Darren Sherrell, biophysicien et scientifique en ligne de lumière à la Division des sciences des rayons X de l'APS. "Ce travail ouvre la voie à l'élucidation d'importantes dynamiques structurelles des protéines du coronavirus."