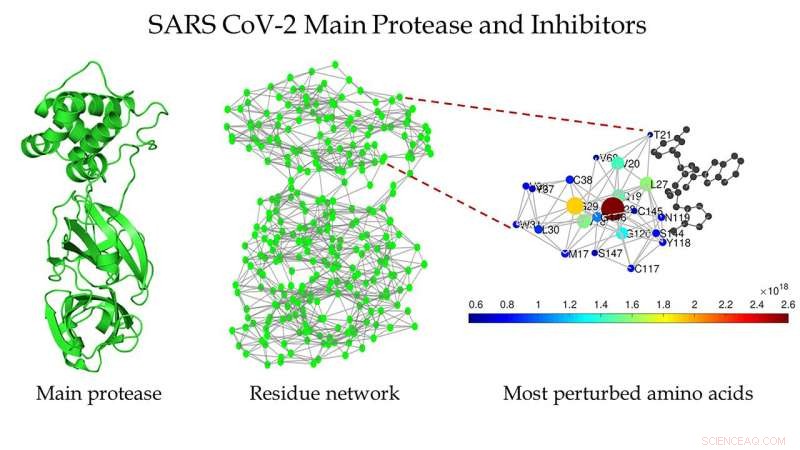
Schéma de la principale protéase du SARS CoV-2 (à gauche), le réseau de résidus protéiques de la principale protéase du SARS CoV-2 (au centre), et une vue agrandie de la région autour du site de liaison telle que détectée par Estrada (à droite). Crédit :Ernesto Estrada
Alors que la pandémie de COVID-19 causée par le coronavirus SARS-CoV-2 continue de se propager dans le monde, de nombreux chercheurs étudient des modèles épidémiologiques pour prédire sa propagation.
Cependant, Ernesto Estrada, mathématicien et expert en systèmes complexes de la Fondation ARAID de l'Université de Saragosse, a décidé de se concentrer sur la recherche de cibles dans le SRAS-CoV-2 pour de nouveaux médicaments à attaquer. De travaux antérieurs, il savait que la principale protéase du virus, une enzyme chargée de la transformation protéolytique des polyprotéines, est une excellente cible.
Dans la revue le chaos , Estrada a déclaré que lorsque lui et ses collègues ont découvert une augmentation spectaculaire de la sensibilité de la principale protéase du SRAS-CoV-2 aux petites perturbations, cela leur a fait soupçonner que les inhibiteurs ont un rôle à jouer dans la destruction du virus.
Les inhibiteurs sont des molécules organiques, médicaments, ou de nouveaux composés chimiques qui se fixent au site de liaison d'une protéase pour inhiber son travail. Un virus mourra sans qu'une enzyme protéolytique ne travaille pour lui.
"J'ai remarqué que les chimistes avaient déjà trouvé de puissants inhibiteurs de la principale protéase du SARS-CoV-2, et qu'ils avaient résolu la structure de cette protéine par cristallographie aux rayons X, " a-t-il déclaré. " C'était choquant de voir que cette protéase est très similaire à celle du coronavirus du SRAS, qui a produit les épidémies de 2003, SRAS-CoV-1."
Lorsque les chercheurs ont superposé les deux structures l'une sur l'autre, ils correspondaient presque parfaitement.
"Si vous alignez les séquences d'acides aminés des deux protéases, il n'y a que 12 résidus sur 306 qui ne coïncident pas, " Dit Estrada. " Y a-t-il quelque chose de caché derrière ces similitudes apparentes entre les deux protéases ? Pouvons-nous apprendre quelque chose d'eux pour améliorer la conception de médicaments contre le virus ?"
Le groupe d'Estrada possède une vaste expérience dans l'analyse des réseaux, comme les réseaux sociaux, l'Internet, ou les chaînes alimentaires entre les espèces au sein d'un environnement - et a décidé de traiter une protéine comme un réseau.
"Ils sont appelés réseaux de résidus de protéines, où nous représentons chaque acide aminé comme un nœud, et l'interaction entre deux acides aminés est représentée par un lien entre les deux, " il expliqua.
Ils ont trouvé plusieurs structures de la protéase principale du SARS CoV-1 et du SARS CoV-2 qui étaient propres, ce qui signifie qu'ils ne contiennent pas de mutations, ligands, ou des solvants au sein de leurs structures. Ils ont transformé leur structure en réseaux de résidus protéiques.
Estrada a déclaré que la plupart des mesures de réseau traditionnelles révélaient que les deux structures étaient très similaires, quelque chose que son équipe savait déjà. "Mais il y a quelques années, nous avons développé une mesure mathématique plus sophistiquée qui nous permet de détecter à quelle distance une perturbation au sein d'un réseau peut se propager. Ce travail était d'une nature très théorique, nature mathématique, mais nous avions supposé qu'il pourrait être utile pour l'étude des protéines."
Alors ils l'ont mis à l'épreuve. Il a révélé que la protéase du SARS-CoV-2 est 1, 900 % plus sensible à la transmission à longue distance des perturbations que la protéase du SARS-CoV-1.
"Cela signifie que lorsqu'une protéine est perturbée, par exemple par l'eau dans l'environnement intracellulaire, de telles perturbations sont transmises à travers un réseau d'intra-résidus qui forment la structure 3-D de la protéine, " Estrada a déclaré. " Si une telle perturbation est produite autour d'un acide aminé donné dans la protéase du SARS-CoV-1, il n'est transmis que par un environnement proche autour de cet acide aminé perturbé."
Mais si cette perturbation se produit sur un acide aminé dans la protéase du SARS-CoV-2, elle est transmise à presque tout le réseau, voire à des acides aminés très éloignés.
"C'est remarquable, car cela signifie qu'avec de minuscules différences structurelles, la protéase du SARS-CoV-2 est beaucoup plus efficace dans les communications intra-résidus, " Estrada a déclaré. "Il devrait être beaucoup plus efficace pour faire son travail en tant qu'enzyme protéolytique du virus. Le diable a fait un travail presque parfait ici, mais il laissa la porte ouverte. Cette grande sensibilité de la protéase SARS-CoV-2 aux perturbations peut être son talon d'Achille par rapport aux inhibiteurs."
L'approche du groupe peut être utilisée pour des protocoles de dépistage massif afin d'identifier de puissants inhibiteurs de la protéase principale du SRAS-CoV-2 et, par conséquent, pour le développement de nouveaux médicaments pour le tuer.