
Dr. Hein Wijma (à gauche) et Prof. Dr. Dick B. Janssen, scientifiques de l'Université de Groningue, qui a utilisé une nouvelle approche informatique pour reconcevoir l'enzyme aspartase et la convertir en un catalyseur pour les réactions d'hydroamination asymétrique. Crédit :Université de Groningue
Les biotechnologues de l'Université de Groningue ont utilisé une méthode informatique pour reconcevoir l'aspartase et la convertir en un catalyseur pour les réactions d'hydroamination asymétrique. Leurs collègues en Chine ont intensifié la production de cette enzyme et ont réussi à produire des kilogrammes de blocs de construction très purs pour les produits pharmaceutiques et autres composés bioactifs. Cette étude de preuve de principe réussie a été publiée dans Nature Chimie Biologie le 21 mai.
Les enzymes sont des catalyseurs naturels qui fonctionnent dans des conditions douces. Ils constituent une alternative intéressante aux réactions chimiques non catalysées qui nécessitent souvent des températures ou des pressions élevées consommatrices d'énergie et peuvent générer des produits secondaires toxiques ou utiliser des solvants. Mais il y a un problème :la gamme des réactions catalysées par les enzymes est limitée. "C'est pourquoi beaucoup d'efforts sont déployés pour modifier les enzymes naturelles, " explique Dick Janssen, Professeur de biotechnologie chimique à l'Institut biomoléculaire et de biotechnologie de Groningen.
La méthode classique pour modifier les enzymes est l'évolution dirigée, une séquence de sélection de mutations en laboratoire qui vise à créer des enzymes aux capacités catalytiques modifiées. Mais il faut beaucoup de travail pour créer et tester des centaines ou des milliers de variantes d'enzymes en plusieurs tours. Il serait beaucoup plus efficace de concevoir de manière rationnelle les changements requis sur la base d'informations sur la structure et les propriétés de l'enzyme.
Mais même cela est compliqué, explique Hein Wijma. Il est un expert en logiciels de conception moléculaire et a effectué la plupart des travaux de calcul de l'étude. "Les protéines sont constituées de 20 acides aminés différents. Donc, si vous voulez changer une enzyme en quatre positions, il y a 20 options pour chacun d'eux. Cela se traduit par une énorme matrice de structures protéiques. » En les testant une par une, même sur l'ordinateur, prend trop de temps. Cependant, un algorithme de recherche Monte Carlo très rapide accélère la découverte du bon résultat en recherchant les tendances de la réactivité de l'enzyme.
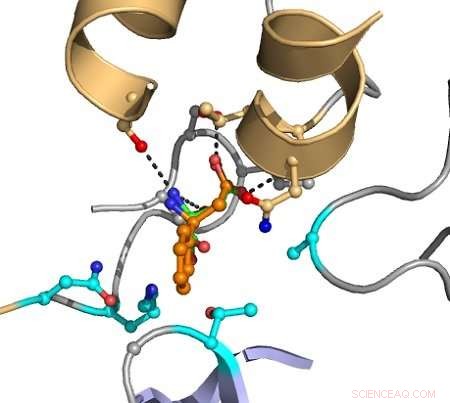
Les lignes brisées indiquent les interactions entre les atomes provoquant la catalyse. Le substrat cible est indiqué en orange, et les groupes indiqués en magenta sont modifiés dans l'ordinateur pour découvrir des mutants qui améliorent la conversion. Crédit :Hein Wijma/Université de Groningue
"À la fin, il a fallu quelques jours pour effectuer cette recherche dans un cluster informatique dédié à l'université de Groningue, " dit Wijma. Mais il a fallu beaucoup de modélisation au préalable. " Il faut modéliser le centre réactif, les poches où se fixe le substrat, et déterminer la distance entre les acides aminés et leurs positions et angles relatifs. » Comme le groupe a utilisé une enzyme (aspartase) comme point de départ pour un certain nombre de réactions différentes, le point de départ était toujours le même. Cela signifiait qu'ils n'avaient qu'à changer la réaction cible. Wijma :« Si on voulait faire une nouvelle modification de l'aspartase, cela ne prendrait probablement que trois mois maintenant."
Le document de recherche décrit quatre conversions, tous les ajouts d'ammonium. L'aspartase est une désaminase, la réaction s'est donc inversée. "La catalyse va dans les deux sens, donc ce n'est pas un problème majeur, " dit Janssen. Pour chaque conversion, la sélection initiale par l'algorithme a produit une centaine de mutants prometteurs. Ceux-ci ont ensuite été vérifiés pour les erreurs évidentes. Janssen :« En fin de compte, quelque cinq à 20 mutants ont été fabriqués en laboratoire et testés pour voir s'ils fonctionnaient comme prévu."
L'étape suivante consistait à tester des enzymes mutantes réussies dans un cadre à grande échelle. "Ce travail a été réalisé par un ancien doctorant et post-doctorant de notre laboratoire, Bian Wu, qui est maintenant professeur assistant en Chine, " dit Janssen. " Il a montré quels candidats pouvaient produire de grandes quantités du produit requis. ce qui signifie que les enzymes prédites par les méthodes de calcul semblent adaptées à une utilisation en milieu industriel. "C'est une vraie preuve de principe que notre méthode de sélection in-silico de mutants fonctionne pour produire des enzymes utiles, " conclut Janssen.