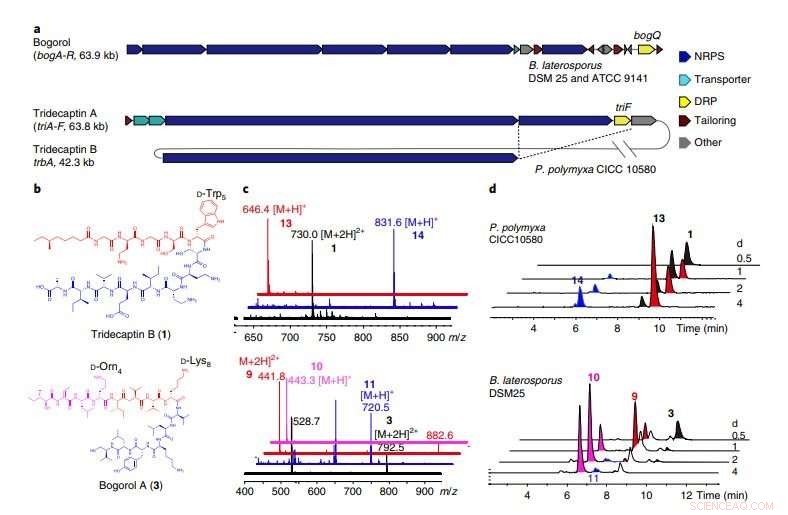
une, Les BGC de bogorol de B. laterosporus DSM 25 et ATCC 9141, ainsi que la tridécaptine A et la tridécaptine B de P. polymyxa CICC 10580. BogQ de la souche DSM 25 et celui de la souche ATCC 9141 partagent 85 pour cent d'identité de séquence d'acides aminés. Les lignes pointillées qui se croisent indiquent des gènes partagés par deux groupes de gènes au sein du même hôte (Fig. 10 supplémentaire). b, Structures des DNRP tridécaptine B et bogorol A, avec des motifs de reconnaissance DRP mis en évidence. c, Superposition empilée des spectres de masse (ionisation électrospray) des composés parents (noir), fragments C-terminaux (bleu), et fragments N-terminaux (rouge). Les données sont représentatives de deux expériences indépendantes. Sommet, tridécaptine B; bas, bogorol A.d, Analyses temporelles des composés correspondants produits par P. polymyxa CICC 10580 (en haut) et B. laterosporus DSM 25 (en bas) à différents temps de fermentation (représentatifs de trois expériences indépendantes). Crédit :Division des sciences de la vie, HKUST
Antibiotiques peptidiques non ribosomiques, dont la polymyxine, vancomycine, et la teixobactine, dont la plupart contiennent des acides aminés D, sont très efficaces contre les bactéries multirésistantes. Cependant, l'abus d'antibiotiques tout en ignorant le risque d'apparition de résistances a inexorablement conduit à l'émergence généralisée de bactéries résistantes. Élucider les mécanismes peu connus de résistance aux antibiotiques peptidiques est essentiel lors de la mise en œuvre d'antibiotiques peptidiques et permettrait d'améliorer efficacement l'efficacité.
Dans une étude récente, un groupe de scientifiques de l'Université des sciences et technologies de Hong Kong révèle à la fois la distribution généralisée et le potentiel de résistance à large spectre des peptidases D-stéréospécifiques, fournissant un indicateur précoce potentiel de la résistance aux antibiotiques aux antibiotiques peptidiques non ribosomiques.
Leurs conclusions ont été publiées dans la revue Nature Chimie Biologie le 26 février, 2018.
"Nous avons appliqué une approche à 5, 585 génomes bactériens complets couvrant tout le domaine des bactéries, " dit Pei-Yuan Qian, professeur titulaire de la Division des sciences de la vie, HKUST, et auteur principal de l'article. "Avec des analyses chimiques et enzymatiques ultérieures, nous avons démontré un mécanisme de résistance aux antibiotiques peptidiques non ribosomiques basé sur le clivage hydrolytique par des peptidases spécifiques D-stéréo."
L'équipe a identifié une famille de peptidases de résistance D-stéréospécifiques (DRP) qui sont phylogénétiquement largement distribuées dans la nature. La découverte des DRP s'est avérée impliquée dans la lutte contre les antibiotiques largement distribués contenant du D-aa pour la survie de leur hôte, qui a été validé expérimentalement par une combinaison d'édition de gènes basée sur CRISPR/Cas9, analyses chimiques et enzymatiques.
« Étant donné le potentiel des DRP pour la résistance à large spectre et leur potentiel à cibler des antibiotiques cliniquement importants contenant du D-aa, ces gènes de résistance largement distribués sont susceptibles d'être particulièrement dangereux s'ils sont transférés à des agents pathogènes opportunistes, " a déclaré le professeur Qian. " La découverte de DRP dans la nature ne constitue que la pointe de l'iceberg, ce qui conduira à d'intenses recherches sur l'utilisation et le développement d'antibiotiques peptidiques pour lutter contre la résistance aux antibiotiques."
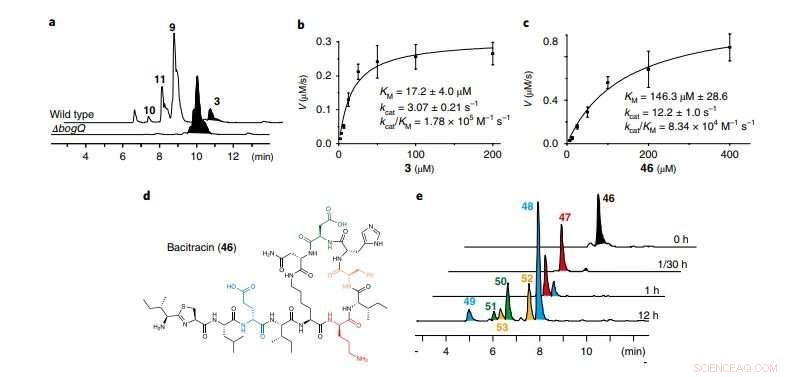
une, Traces LC-MS comparant B. laterosporus ATCC 9141 sauvage et le mutant ΔbogQ (représentatif de trois expériences indépendantes). b, c, Analyses cinétiques de l'hydrolyse catalysée par BogQ du bogorol A (b; 3) et de la bacitracine (c; 46) v, vitesse de réaction. Les données sont moyennes ?± ?s.d. ; n=?3 expériences indépendantes. ré, Structure de l'antibiotique DNRP bacitracine ; les couleurs mettent en évidence les sites de clivage de BogQ. e, Traces LC-MS de dosages in vitro de BogQ (2,0 µM) contre 46 (200 µM; représentatif de deux expériences indépendantes). Les produits de clivage au cours du temps (47-53) de 46 sont étiquetés en utilisant le même code couleur que leurs sites de clivage d-aa dans d. Pour les schémas de clivage enzymatique, voir Fig. 22 supplémentaire. Crédit :Division of Life Science, HKUST