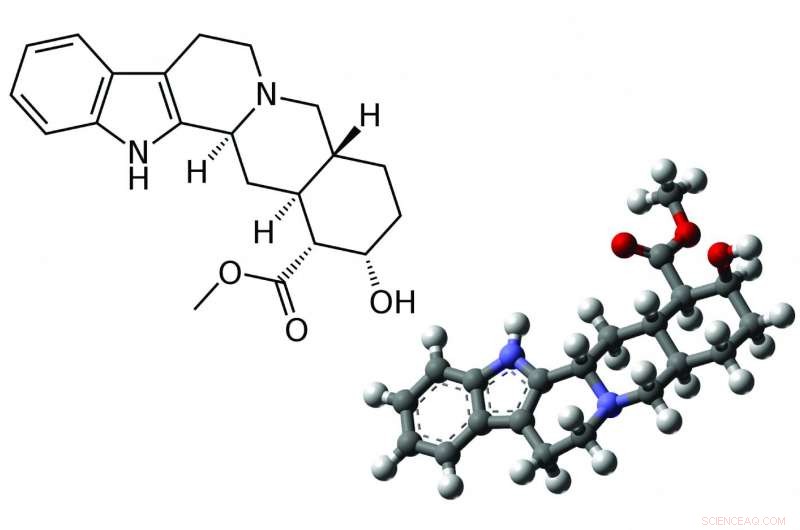
Un programme informatique peut comprendre la structure 3-D (à droite) de molécules comme la yohimbine en appuyant simplement sur un bouton. Les molécules ayant la même structure 2D (à gauche) peuvent avoir des structures 3D différentes. Crédit :Structure 2D :acdx (Wikipédia)Structure 3D :MindZiper (Wikipédia)
Une équipe internationale de chercheurs dirigée par le chimiste de l'Université Carnegie Mellon Roberto R. Gil et le chimiste de l'Université fédérale de Pernambuco Armando Navarro-Vázquez a développé un programme qui automatise le processus de détermination de la structure tridimensionnelle d'une molécule. La technique, décrit dans un article de Angewandte Chemie , compresse un processus qui prend généralement des jours en minutes et pourrait raccourcir le pipeline de découverte de médicaments en réduisant l'erreur humaine.
Comprendre la structure chimique d'une molécule à partir de zéro est une partie essentielle de la recherche de produits chimiques qui proviennent de la nature, ou « produits naturels ». Pour les substances à usage pharmaceutique possible, cette structure peut révéler comment la substance pourrait interagir avec le corps humain.
« Si la molécule doit être un médicament, vous devez connaître la forme de la molécule pour savoir comment elle va interagir avec un récepteur, " dit Gil, professeur au département de chimie du Mellon College of Science de Carnegie Mellon.
La première étape pour déterminer la structure d'une molécule consiste à déterminer ses éléments constitutifs atomiques, suivi de la découverte de sa structure bidimensionnelle, qui montre comment chaque atome est connecté les uns aux autres. Alors que certaines liaisons atomiques sont rigides, d'autres peuvent tourner autour d'un joint, permettant à des molécules ayant les mêmes composants et structures bidimensionnelles (2D) d'avoir des formes tridimensionnelles (3D) différentes.
De petites différences de forme peuvent se traduire par de grands changements dans la façon dont les médicaments agissent dans le corps. Par exemple, la rotation d'un lien dans l'analgésique populaire ibuprofène le rend complètement inactif. De la même manière, l'amidon et la cellulose partagent la même structure 2D mais ont des formes 3D différentes. Cette différence est la raison pour laquelle les humains peuvent digérer les céréales et non le bois.
Gil et Navarro-Vázquez travaillent depuis huit ans pour simplifier le processus de recherche et de tri des formes 3D possibles pour toute structure 2D donnée. Et avec la généralisation et la disponibilité des outils de laboratoire pour collecter des données sur la structure 3D d'une molécule, le moment était venu de développer une méthode pour automatiser et rationaliser le processus.
Les chercheurs ont créé un programme, écrit en langage de programmation Python, qui utilise les informations de couplage dipolaire résiduel (RDC), une mesure de la distance entre les atomes s'étendant des liaisons en rotation. Nourri de données sur une molécule donnée à partir d'expériences RDC, le programme génère des façons possibles que la molécule peut exister en trois dimensions, et choisit l'option la plus probable.
La technique est la plus efficace pour s'attaquer à la structure 3D des molécules organiques de taille petite à moyenne, et relativement rigide, avec des atomes de carbone emballés dans des anneaux plutôt que liés en longs, chaînes pliées. L'équipe a testé son programme sur six de ces molécules, dont la naltrexone, un médicament utilisé pour bloquer les effets des opioïdes, et la strychnine, un pesticide.
D'abord, ils ont déterminé la structure 2D de chaque molécule à l'aide d'un programme d'élucidation de structure assistée par ordinateur (CASE), aidé par une collaboration avec Clemens Anklin, vice-président des applications RMN et de la formation chez Bruker Corporation. Ils ont alimenté cette information, ainsi que les données RDC sur la molécule, dans leur nouveau programme. Dans chaque cas, leur programme a pu sélectionner la structure 3D correcte.
"Vous appuyez sur un bouton, et avec peu ou pas d'intervention humaine, vous passez de la structure 2D à la structure 3D en un seul coup, " dit Gil.
Mais tout aussi important que la vitesse du programme est sa rigueur.
« La quantité de produits naturels qui sont déclarés à tort, où la structure rapportée ne correspond pas à la structure réelle, est vraiment grand, " dit Navarro-Vázquez, professeur de chimie à l'Universidade Federal de Pernambuco au Brésil. Ce programme pourrait être un moyen précieux de vérifier les structures possibles que les chercheurs pourraient autrement manquer, en les aidant à éviter leurs propres préjugés inhérents.
Gil fait remonter sa vision de ce processus à son époque en tant qu'étudiant diplômé et à une conversation avec un mentor il y a 30 ans sur l'idée que les chimistes pourraient un jour mettre une substance dans une machine et voir sa structure en appuyant sur un bouton. Bientôt, il espère, des programmes comme le sien et la création de Navarro-Vázquez intégreront les premières étapes du processus, rapprocher encore plus cette vision de la réalité.
"Nous sommes très proches, " dit Gil. "C'est le rêve de tout chimiste."