L'observation de cellules individuelles au microscope peut révéler une série de phénomènes biologiques cellulaires importants qui jouent fréquemment un rôle dans les maladies humaines, mais le processus de distinction des cellules individuelles les unes des autres et de leurs antécédents prend extrêmement de temps et constitue une tâche bien adaptée. pour l'assistance de l'IA.
Les modèles d'IA apprennent à effectuer de telles tâches en utilisant un ensemble de données annotées par des humains, mais le processus de distinction des cellules de leur arrière-plan, appelé « segmentation unicellulaire », est à la fois long et laborieux. Par conséquent, la quantité de données annotées à utiliser dans les ensembles de formation en IA est limitée. Les chercheurs d'Uc Santa Cruz ont développé une méthode pour résoudre ce problème en construisant un modèle d'IA de génération d'images de microscopie pour créer des images réalistes de cellules uniques, qui sont ensuite utilisées comme « données synthétiques » pour former un modèle d'IA afin de mieux effectuer la segmentation d'une seule cellule.
Le nouveau logiciel est décrit dans un nouvel article publié dans la revue iScience . Le projet a été dirigé par le professeur adjoint de génie biomoléculaire Ali Shariati et son étudiant diplômé Abolfazl Zargari. Le modèle, appelé cGAN-Seg, est disponible gratuitement sur GitHub.
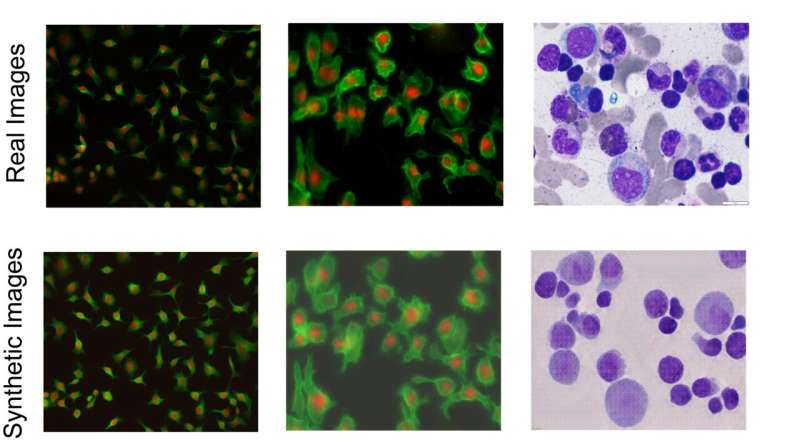
"Les images issues de notre modèle sont prêtes à être utilisées pour former des modèles de segmentation", a déclaré Shariati. "Dans un sens, nous faisons de la microscopie sans microscope, dans la mesure où nous sommes capables de générer des images très proches des images réelles des cellules en termes de détails morphologiques de la cellule unique. La beauté de la chose est que lorsqu'elles sortent, du modèle, elles sont déjà annotées et étiquetées. Les images montrent une tonne de similitudes avec des images réelles, ce qui nous permet ensuite de générer de nouveaux scénarios qui n'ont pas été vus par notre modèle lors de la formation."
Les images de cellules individuelles vues au microscope peuvent aider les scientifiques à en apprendre davantage sur le comportement et la dynamique des cellules au fil du temps, à améliorer la détection des maladies et à trouver de nouveaux médicaments. Les détails subcellulaires tels que la texture peuvent aider les chercheurs à répondre à des questions importantes, comme celle de savoir si une cellule est cancéreuse ou non.
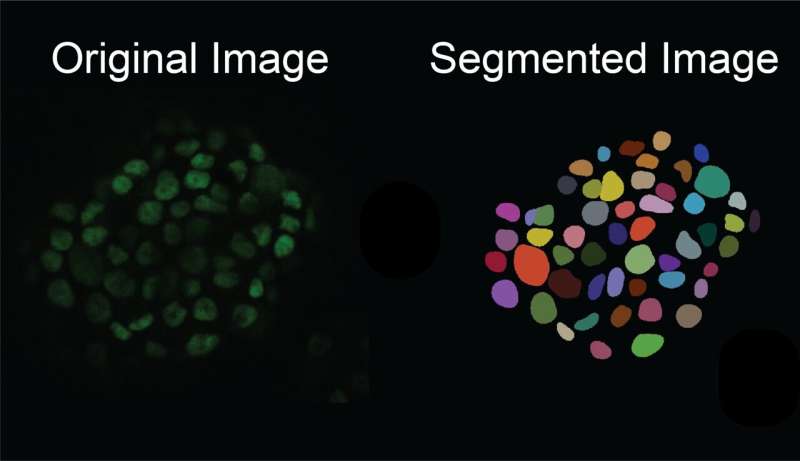
Trouver et marquer manuellement les limites des cellules à partir de leur arrière-plan est cependant extrêmement difficile, en particulier dans les échantillons de tissus où il y a de nombreuses cellules dans une image. Cela pourrait prendre plusieurs jours aux chercheurs pour effectuer manuellement une segmentation cellulaire sur seulement 100 images microscopiques.
L'apprentissage profond peut accélérer ce processus, mais un ensemble initial de données d'images annotées est nécessaire pour entraîner les modèles :au moins des milliers d'images sont nécessaires comme référence pour entraîner un modèle d'apprentissage profond précis. Même si les chercheurs peuvent trouver et annoter 1 000 images, ces images peuvent ne pas contenir les variations de caractéristiques qui apparaissent dans différentes conditions expérimentales.
"Vous souhaitez montrer que votre modèle d'apprentissage profond fonctionne sur différents échantillons avec différents types de cellules et différentes qualités d'image", a déclaré Zargari. "Par exemple, si vous entraînez votre modèle avec des images de haute qualité, il ne pourra pas segmenter les images cellulaires de mauvaise qualité. Nous pouvons rarement trouver un ensemble de données aussi performant dans le domaine de la microscopie."
Pour résoudre ce problème, les chercheurs ont créé un modèle d'IA génératif d'image à image qui prend un ensemble limité d'images cellulaires annotées et étiquetées et en génère davantage, introduisant des caractéristiques et des structures subcellulaires plus complexes et plus variées pour créer un ensemble diversifié d'images « synthétiques ». images. Ils peuvent notamment générer des images annotées avec une densité élevée de cellules, particulièrement difficiles à annoter à la main et particulièrement pertinentes pour l’étude des tissus. Cette technique permet de traiter et de générer des images de différents types de cellules ainsi que différentes modalités d'imagerie, telles que celles prises par fluorescence ou coloration histologique.
Zargari, qui a dirigé le développement du modèle génératif, a utilisé un algorithme d'IA couramment utilisé appelé « réseau contradictoire génératif à cycle » pour créer des images réalistes. Le modèle génératif est amélioré avec ce que l'on appelle des « fonctions d'augmentation » et un « réseau d'injection de style », qui aident le générateur à créer une grande variété d'images synthétiques de haute qualité montrant différentes possibilités quant à l'apparence possible des cellules. À la connaissance des chercheurs, c'est la première fois que des techniques d'injection de style sont utilisées dans ce contexte.
Ensuite, cet ensemble diversifié d'images synthétiques créées par le générateur est utilisé pour entraîner un modèle afin d'effectuer avec précision une segmentation cellulaire sur de nouvelles images réelles prises lors d'expériences.
"En utilisant un ensemble de données limité, nous pouvons former un bon modèle génératif. En utilisant ce modèle génératif, nous sommes en mesure de générer un ensemble plus diversifié et plus large d'images synthétiques annotées. En utilisant les images synthétiques générées, nous pouvons former un bon modèle de segmentation - c'est l'idée principale", a déclaré Zagari.
Les chercheurs ont comparé les résultats de leur modèle utilisant des données d’entraînement synthétiques à des méthodes plus traditionnelles d’entraînement de l’IA pour effectuer la segmentation cellulaire entre différents types de cellules. Ils ont constaté que leur modèle produit une segmentation considérablement améliorée par rapport aux modèles formés avec des données de formation conventionnelles et limitées. Cela confirme aux chercheurs que fournir un ensemble de données plus diversifié lors de la formation du modèle de segmentation améliore les performances.
Grâce à ces capacités de segmentation améliorées, les chercheurs pourront mieux détecter les cellules et étudier la variabilité entre les cellules individuelles, en particulier entre les cellules souches. À l'avenir, les chercheurs espèrent utiliser la technologie qu'ils ont développée pour aller au-delà des images fixes et générer des vidéos, ce qui peut les aider à identifier les facteurs qui influencent le sort d'une cellule au début de sa vie et à prédire son avenir.
"Nous générons des images synthétiques qui peuvent également être transformées en film accéléré, où nous pouvons générer l'avenir invisible des cellules", a déclaré Shariati. "Avec cela, nous voulons voir si nous sommes capables de prédire les états futurs d'une cellule, par exemple si la cellule va croître, migrer, se différencier ou se diviser."
Plus d'informations : Abolfazl Zargari et al, Segmentation cellulaire améliorée avec des ensembles de données de formation limités à l'aide de réseaux contradictoires génératifs de cycle, iScience (2024). DOI :10.1016/j.isci.2024.109740
Informations sur le journal : iScience
Fourni par l'Université de Californie - Santa Cruz