
Extrait du film time-lapse de détection de miARN en 1 heure à l'aide du test miRacles. Crédit :Avancées scientifiques, doi:10.1126/sciadv.aau9443
Les microARN (miARN) sont de courts ARN régulateurs non codants qui peuvent réprimer l'expression des gènes de manière post-transcriptionnelle et sont donc de plus en plus utilisés comme biomarqueurs de la maladie. La détection des miARN peut être ardue et coûteuse car ils nécessitent une amplification, marquage et sondes radioactives. Dans un récent rapport publié le Avancées scientifiques , Arun Richard Chandrasekaran et ses collègues du RNA Institute and Department of Biological Sciences, à l'Université d'Albany, Université d'État de New York, rapporté sur une seule étape, test de détection de microARN non enzymatique utilisant des nanocommutateurs d'ADN conformationnellement insensibles.
Les scientifiques ont nommé l'essai « miRacles, ' pour abréger 'bouclage conditionnel activé par micro-ARN des commutateurs conçus'. L'essai a démontré une spécificité de sous-attomole et de nucléotide unique en utilisant une lecture d'électrophorèse sur gel d'agarose. Dans les expériences, ils ont détecté des microARN cellulaires à partir d'extractions d'ARN obtenues à partir de muscles en différenciation à l'échelle du nanogramme. Les scientifiques ont présenté une configuration expérimentale rentable pour détecter les miARN en quelques heures afin de fournir une alternative convaincante aux méthodes existantes de réaction en chaîne par polymérase quantitative (qPCR) et de Northern blot pour quantifier le matériel génétique réglementaire.
Les miARN peuvent réguler de nombreux processus biologiques au cours du développement physiologique normal et de la maladie en affectant la prolifération cellulaire, différenciation et apoptose in vivo. L'expression des miARN peut être quantifiée dans les tissus, les cellules et les fluides corporels en tant que biomarqueurs stables pour les événements cellulaires et le diagnostic des maladies, soulignant l'importance de leur détection. Néanmoins, La détection des miARN est difficile en raison de leur faible abondance, petite taille et similitudes dans l'ordre. Les biomolécules représentent environ 0,01 pour cent de la teneur totale en ARN et les copies d'ARNm individuelles par plage cellulaire peuvent varier considérablement. En outre, Les miARN d'une famille peuvent différer d'un seul nucléotide, tandis que des miARN spécifiques peuvent être régulés pendant la maladie et la fonction cellulaire régulière. Par conséquent, les stratégies de détection des miARN nécessitent une spécificité élevée et la capacité d'identifier correctement quelques molécules à partir d'un échantillon abondant avec des molécules d'ARN prédominantes.
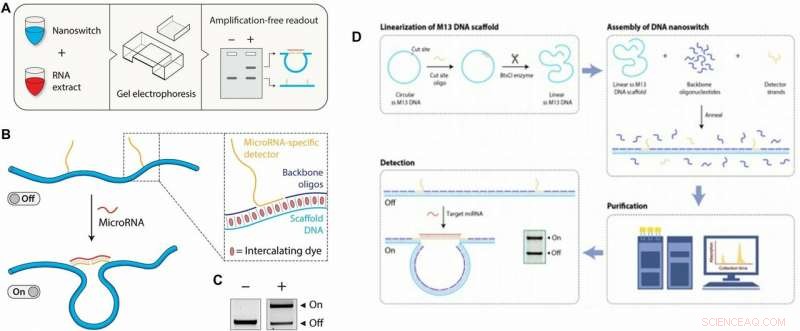
À gauche :Concept et flux de travail du test miRacles. (A) Flux de travail du test miRacles :des nanocommutateurs d'ADN personnalisés sont mélangés avec l'échantillon de miARN cible, incubé, et exécuter sur un gel d'agarose pour la détection. (B) Les nanocommutateurs d'ADN subissent un changement de conformation d'un état linéaire « off » à un état « on » en boucle lorsqu'ils sont liés à un miARN cible. Encart :Le nanoswitch est composé d'un échafaudage monobrin M13, oligonucléotides du squelette, et des extensions simple brin (détecteurs) complémentaires du miARN cible. Les colorants intercalants intrinsèques au processus d'électrophorèse fournissent le signal pour visualiser les nanocommutateurs. (C) Les deux conformations sont résolubles dans un gel d'agarose standard. À droite :examen approfondi du processus de construction des nanocommutateurs d'ADN. (D) Linéarisation :La préparation d'un échafaudage linéaire simple brin M13 procède par annelage d'un oligonucléotide pour permettre le clivage au niveau du site double brin par une enzyme de restriction (BtsCI dans ce cas). Assemblage :l'assemblage du nanoswitch d'ADN est réalisé en mélangeant un excès molaire 10x d'oligonucléotides du squelette et de brins détecteurs (spécifiques à la cible) au M13 simple brin et en appliquant une rampe de refroidissement de 90˚C à 25˚C à 1˚C/min . Purification :Pour éliminer les oligonucléotides en excès, une méthode de chromatographie liquide est utilisée. Détection :Pour utiliser les nanocommutateurs, ils sont mélangés avec une solution contenant des cibles de microARN et exécutés sur un gel pour résoudre les états marche et arrêt comme indiqué dans l'encart. Crédit :Avancées scientifiques, doi:10.1126/sciadv.aau9443
Les méthodes traditionnelles de détection des miARN comprennent le transfert de Northern, amplification en chaîne par polymérase par transcription inverse quantitative (qRT-PCR), séquençage de nouvelle génération et hybridation basée sur des puces à ADN pour séparer le signal miARN du bruit. Parmi les méthodes spécifiées, Le Northern blot permet d'identifier directement les miARN natifs, tandis que d'autres s'appuient sur des méthodes de marquage supplémentaires ou une amplification par étapes, ajouter au coût, complexité et performance de la détection. Par exemple, des nanostructures d'ADN innovantes peuvent être utilisées pour la détection de miARN, où divers groupes de recherche ont précédemment combiné des nanostructures avec des nanoparticules, des réactions en chaîne d'hybridation et des nanofeuillets de dichalcogénure de métal de transition pour permettre le processus.
Dans le travail present, Chandrasekaran et al. développé un dispositif basé sur l'ADN relativement simple pour résoudre un défi biomédical complexe. Dans le test miRacles, les scientifiques ont utilisé un « réactif intelligent » composé de nanocommutateurs d'ADN de conception rationnelle pour une détection simple et économique des miARN natifs sans utiliser d'équipement spécialisé en laboratoire. Les nanocommutateurs d'ADN ont été initialement conçus comme des outils pour les expériences de biophysique à molécule unique et reconnus plus tard pour leur capacité à détecter et quantifier les interactions biomoléculaires à l'aide d'électrophorèse sur gel. Les efforts de recherche collaboratifs précédents de la même équipe de recherche se sont concentrés sur la détection moléculaire pour quantifier les niveaux de protéines et détecter les séquences d'ADN synthétique comme preuve de concept.
Le présent travail s'est étendu sur des études et des concepts préliminaires pour produire une détection et une quantification de miARN multiplexés prêtes à l'emploi. Les scientifiques ont analysé des nanogrammes d'extraits d'ARN cellulaire dans un court laps de temps à l'aide d'une configuration expérimentale construite à l'aide de fournitures de laboratoire courantes. Ils ont conçu le nanocommutateur d'ADN comme un duplex linéaire qui a formé une boucle en présence de la molécule de miARN cible. Pour construire le nanocommutateur, Chandrasekaran et al. ont utilisé l'approche de l'origami d'ADN en hybridant de courts oligonucléotides complémentaires à un échafaudage d'ADN simple brin.
Ils ont conçu deux brins "détecteurs" distants avec des surplombs complémentaires à différents segments du miARN cible. Lorsque le miARN reconnu et lié à la construction, l'interrupteur reconfiguré de l'état linéaire "off" à l'état bouclé "on". Ils ont quantifié les deux états en utilisant une électrophorèse sur gel d'agarose standard pour détecter le signal provenant du nanocommutateur en boucle. Le signal n'a été amplifié que par un seul miARN d'intérêt, les résultats se comparent favorablement avec la technique de transfert d'énergie par résonance de fluorescence (FRET).

Validation du test miRacles. (A) Spécificité des nanocommutateurs d'ADN avec des détecteurs conçus pour let-7b. Une incompatibilité aussi faible que 1 nt entre les détecteurs et le miARN cible élimine le signal. (B) Limite de détection du test. NC, contrôle négatif. UA, unités arbitraires. (C) Déroulement dans le temps du dosage pour une cible à faible concentration. (D) Plage dynamique du dosage à différents temps de réaction. Crédit :Avancées scientifiques, doi:10.1126/sciadv.aau9443.
Pour valider le concept, l'équipe de chercheurs a choisi un miARN cible let-7b en raison de sa famille hautement conservée de plus d'une douzaine de miARN apparentés qui variaient d'un ou plusieurs nucléotides. Ces miARN étaient adaptés en raison de leur rôle essentiel dans les fonctions biologiques et la dérégulation de plusieurs maladies humaines. Pour éliminer la diaphonie et le bruit entre le nanocommutateur et la cible, ce qui a entraîné une réduction de l'intensité du signal par rapport à une correspondance parfaite, les scientifiques ont repensé rationnellement les nanocommutateurs. Pour obtenir une spécificité parfaite, ils ont déstabilisé l'interaction du côté contenant le mésappariement. Les résultats de l'étude ont illustré la haute spécificité du test ainsi développé, apporter une réponse à un défi clé dans la détection des miARN qui a abouti à un rapport signal/bruit élevé.
La faible abondance des miARN nécessitait également une haute sensibilité de détection, que les scientifiques ont obtenu en optimisant le protocole. Ils ont ensuite réalisé des expériences similaires pour deux autres variantes de miARN (miR-15 a et miR-206), résultant en des niveaux de détection de sous-attomole à un seul attomole. Par exemple, pour une faible concentration d'un échantillon cible (6 pM), le signal a augmenté pendant 4 heures, avec peu de changement au-delà de cette période.
Détection de miARN à partir de cellules myoblastiques différenciées. (A) Schéma montrant les cellules de myoblastes, récolté en culture GM et aux jours de différenciation 1 et 4, traités pour donner des fractions d'ARN total et petit. Un marqueur de différenciation myogénique précoce, Myog, et un marqueur de différenciation myogénique tardive, CMH, ont été mesurés par (B) Western blot et (C) par immunocytochimie pour confirmer la différenciation. Myog et MHC étaient tous deux régulés à la hausse dans DM1 et DM4. La GAPDH (glycéraldéhyde-3-phosphate déshydrogénase) a servi de témoin en (B). (D) Chandrasekaran et al. détecté miR-206 dans les échantillons différenciés avec 50 ng de petits ARN et (E) avec 500 ng d'ARN totaux. La quantification des intensités de gel montre une forte régulation à la hausse progressive au cours de la différenciation, similaire dans les échantillons (F) de petit ARN et (G) d'ARN total. A partir d'échantillons DM4, nous notons la détection à partir d'aussi peu que (H) 200 pg de petits ARN et (I) 500 pg d'ARN totaux. Crédit :Avancées scientifiques, doi:10.1126/sciadv.aau9443. 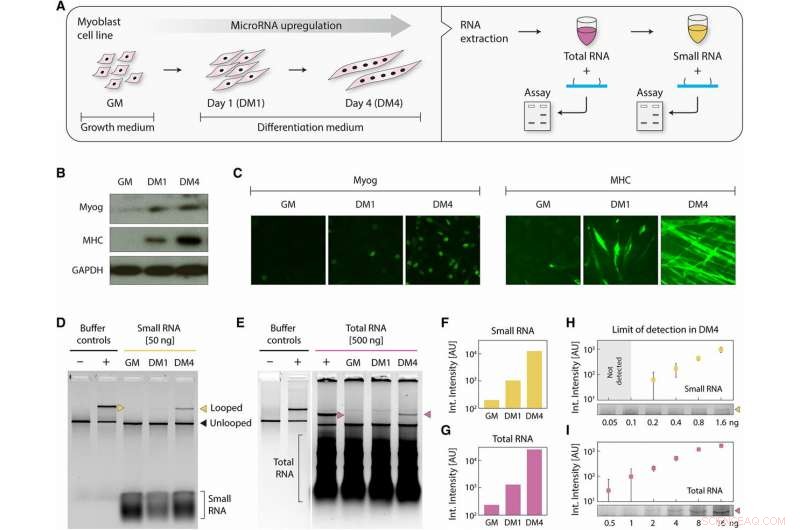
Étant donné que plusieurs ARNm peuvent également modifier leur expression au cours de différents stades cellulaires ou de la maladie, le phénomène nécessitait des capacités de détection supplémentaires. Pour ce faire dans le cadre expérimental, Chandrasekaran et al. utilisé la programmabilité des nanocommutateurs et développé un système de multiplexage capable de détecter plusieurs miARN à partir du même échantillon. Ils ont placé les brins du détecteur aux emplacements souhaités de l'échafaudage d'ADN, résultant en des boucles de différentes tailles lorsqu'elles sont liées au miARN cible. La taille de boucle du nanocommutateur ainsi déterminé la migration du gel, résultant en une bande unique sur le gel pour une détection précise. Pour l'expérimentation, les scientifiques ont choisi quatre miARN présents dans les cellules musculaires; miR-206, miR-125b, miR-24 et miR-133-3p et un miARN témoin négatif; miR-39 spécifique à l'espèce Caenorhabditis elegans.
Dans les 50 ng de petits ARN, les scientifiques ont détecté les quatre miARN à différents niveaux d'expression, tout en confirmant l'absence de détection avec le contrôle négatif. La stratégie de multiplexage a permis aux scientifiques de comparer directement les niveaux de miARN dans un seul échantillon, sans marquage ni amplification. Au total, le travail a fourni une étape supplémentaire dans le sens de l'expansion du débit du test miRacles. La capacité peut également être étendue pour accueillir plus de miARN par commutateur.
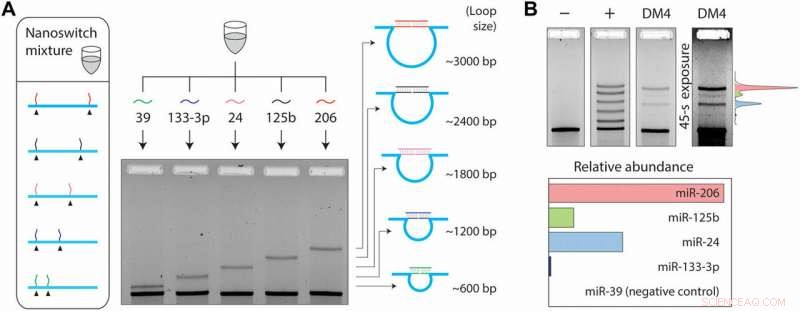
Configuration de multiplexage à cinq canaux. (A) Le multiplexage permet la détection de différents miARN avec différentes tailles de boucles. (B) Un mélange de nanoswitch multiplexé montre cinq bandes avec une intensité similaire dans un contrôle positif composé des cinq miARN cibles. Dans 50 ng de petits ARN DM4, quatre miARN différents sont détectés à différents niveaux d'expression, le miR-39 (un miARN spécifique de C. elegans) n'étant pas détecté.
De cette façon, Chandrasekaran et al. considérablement avancé par rapport à leur détection préliminaire de validation de concept de séquences d'ADN synthétiques ; établir, caractériser et optimiser un test de détection de miARN prêt à l'emploi avec des extraits biologiques. Le travail démontré était un premier exemple d'utilisation de nanocommutateurs d'ADN pour détecter des miARN à partir d'un véritable échantillon biologique. Alors que les performances du test miARN étaient compétitives par rapport à d'autres techniques couramment utilisées, la sélectivité de 1 nucléotide vue dans le présent travail était difficile à accomplir avec les méthodes existantes. La sensibilité de miRacles a également surpassé le transfert de Northern et les puces à ADN. Le test pourrait mesurer les miARN sans avoir besoin d'amplification, avec des protocoles plus simples et sans l'erreur supplémentaire de traitement d'échantillon supplémentaire. Le protocole a simplement mélangé les nanocommutateurs avec l'échantillon liquide pour l'électrophorèse sur gel, pour produire des résultats de haute qualité en laboratoire. Les travaux de recherche sont potentiellement transférables des échantillons biologiques aux échantillons cliniques pour diagnostiquer et surveiller la maladie.
Plus important, le présent travail s'aligne sur le concept plus large de la science frugale; une vision prometteuse de la science rentable qui a déjà produit des solutions peu coûteuses aux techniques de centrifugation du sang et de purification de l'eau en génie biomédical. Chandrasekaran et al. viser à continuer à contribuer à la tendance émergente de la science, en perturbant la relation coût/performance existante pour fournir un accès à grande échelle à des méthodes simples de détection de miARN avec des réactifs intelligents à mélanger et à lire.
© 2019 Réseau Science X