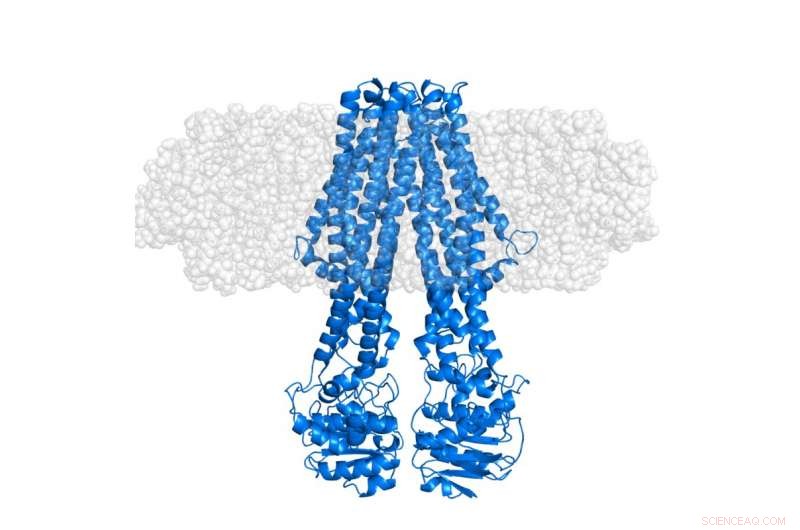
La protéine membranaire intégrale MsbA (bleu) intégrée dans un nanodisque membranaire «semi-transparent» (gris). Le nanodisque lui-même est, par deutération sélective, rendu invisible dans les expériences SANS qui se concentrent sur la conformation de la protéine dans son contexte natif. Crédit :Institut Laue-Langevin
Les membranes qui entourent nos cellules contiennent un grand nombre de protéines. Les protéines membranaires sont donc une classe cruciale de macromolécules dans les systèmes vivants. Ils jouent des rôles clés, tels que la fourniture de passerelles de transport dans et hors de la cellule, faciliter la signalisation entre les cellules, ainsi que d'être impliqué dans la catalyse enzymatique. Ces rôles fonctionnels les rendent particulièrement importants en tant que cibles médicamenteuses, avec la majorité des thérapies actuelles ciblant les protéines membranaires.
Cependant, les études structurelles des protéines membranaires intégrales (IMP) se sont avérées extrêmement difficiles, car la plupart d'entre eux sont difficiles à étudier correctement en l'absence de leur environnement lipidique. Cela les empêche souvent de se cristalliser – une méthode couramment utilisée en biologie structurale classique. Des approches alternatives sont donc nécessaires pour les études structurelles des IMP dans des environnements membranaires. Dans ce but, le Groupe Sciences de la Vie de l'Institut Laue-Langevin (ILL), en collaboration avec l'Université de Copenhague, a lancé avec succès le développement de nanodisques porteurs furtifs. Dans cette approche, une méthode sophistiquée de marquage au deutérium est utilisée pour rendre la membrane efficacement invisible à la diffraction des neutrons à basse résolution tout en mettant en évidence la structure des IMP dans leur environnement lipidique habituel, tel que publié dans Acta Crystallographica D en 2014.
Plus récemment, la première étude structurale d'une protéine membranaire intégrale utilisant cette stratégie de deutération de nanodisques porteurs furtifs vient d'être achevée. Cela a été réalisé à l'aide de la plate-forme Laboratoire de deutération (D-Lab) du Partenariat pour la biologie structurale (PSB) en conjonction avec la diffusion des neutrons aux petits angles (SANS) et la diffusion des rayons X (SAXS) fournies par l'intermédiaire de la plate-forme PSB SANS/SAXS. . Tel que publié dans la revue Structure par Josts et al, l'équipe internationale, dirigé par Henning Tidow, Université de Hambourg, appliqué cette méthode à une protéine de transport de cassette de liaison à l'ATP (ABC), MsbA – qui joue un rôle important dans le transport des lipides chez les bactéries. Les données de diffusion des neutrons résultantes, principalement acquis à l'aide de l'instrument D11 à l'ILL, a permis l'observation directe du signal de la protéine membranaire solubilisée sans contribution du lipide environnant. Les données SAXS ont fourni une référence claire pour la forme externe du nanodisque, compris la bicouche lipidique.
En outre, les changements conformationnels de MsbA ont été étudiés, démontrant la sensibilité de la méthode et son applicabilité générale aux études structurelles des PMI.
Cette approche est susceptible de devenir de plus en plus importante dans les études futures de ces mais d'une importance capitale, macromolécules biologiques, à son tour soutenant une meilleure compréhension pour le développement de médicaments visant les protéines membranaires.