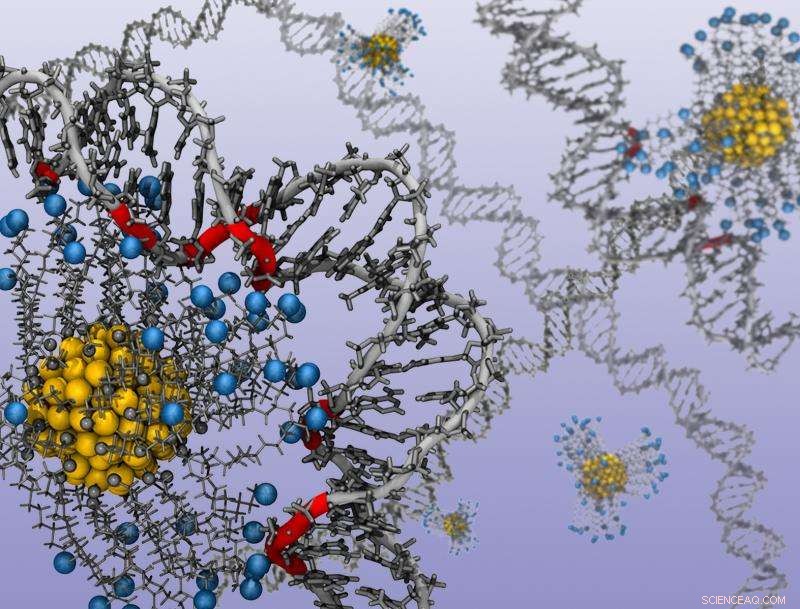
Les chercheurs ont utilisé un modèle informatique de nanoparticules et de ligands d'or pour déterminer comment les acides nucléiques réagissent à diverses charges. En langage technique, l'image montre la liaison de nanoparticules d'or fonctionnalisées avec un ligand alkyle avec des groupes terminaux amine protonés (les sphères bleues) à un ADN double brin. Crédit :Jessica Nash
Des chercheurs de la North Carolina State University ont utilisé la modélisation informatique pour faire la lumière sur l'influence précise des nanoparticules d'or chargées sur la structure de l'ADN et de l'ARN, ce qui pourrait conduire à de nouvelles techniques de manipulation de ce matériel génétique.
Les travaux sont prometteurs pour le développement d'applications capables de stocker et de transporter des informations génétiques, créer des échafaudages personnalisés pour la bioélectronique et créer de nouvelles technologies d'administration de médicaments.
"Dans la nature, mètres d'ADN sont entassés dans chaque cellule vivante, " dit Jessica Nash, un doctorat étudiant à NC State et auteur principal d'un article sur le travail. "C'est possible parce que l'ADN est étroitement enroulé autour d'une protéine chargée positivement appelée histone. Nous aimerions pouvoir façonner l'ADN en utilisant une approche similaire qui remplace l'histone par une nanoparticule d'or chargée. Nous avons donc utilisé des techniques informatiques pour déterminer exactement comment différentes charges influencent la courbure des acides nucléiques - ADN et ARN."
Dans leur modèle, les chercheurs ont manipulé la charge des nanoparticules d'or en ajoutant ou en supprimant des ligands chargés positivement – des molécules organiques attachées à la surface de la nanoparticule. Cela leur a permis de déterminer comment l'acide nucléique a répondu à chaque niveau de charge. Une animation d'une nanoparticule et de ligands façonnant un brin d'ADN est disponible sur www.youtube.com/watch?v=kNpvPy … bmc&feature=youtu.be.
"Cela permettra aux chercheurs de savoir à quoi s'attendre - de la charge dont ils ont besoin pour obtenir la courbure souhaitée dans l'acide nucléique, " dit Yaroslava Yingling, professeur agrégé de science et d'ingénierie des matériaux à NC State et auteur correspondant de l'article.
"Nous avons utilisé des ligands dans le modèle, mais il existe d'autres moyens de manipuler la charge des nanoparticules, " dit Abhishek Singh, chercheur postdoctoral à NC State et co-auteur de l'article. "Par exemple, si les nanoparticules et l'acide nucléique sont en solution, vous pouvez changer la charge en changeant le pH de la solution."
Le travail est également important car il met en évidence le chemin parcouru par la recherche informatique en science des matériaux.
"Nos modèles à grande échelle représentent chaque atome impliqué dans le processus, " dit Nan Li, un doctorat étudiant à NC State et co-auteur de l'article. « Ceci est un exemple de la façon dont nous pouvons utiliser du matériel informatique avancé, tels que les GPU – ou unités de traitement graphique – développés pour une utilisation dans les jeux vidéo, pour effectuer des simulations scientifiques de pointe.
L'équipe de recherche s'appuie maintenant sur ces découvertes pour concevoir de nouvelles nanoparticules avec différentes formes et chimies de surface afin d'obtenir encore plus de contrôle sur la forme et la structure des acides nucléiques.
"Personne n'est parvenu à égaler l'efficacité de la nature lorsqu'il s'agit d'emballer et de déballer les acides nucléiques, ", dit Yingling. "Nous essayons de mieux comprendre comment cela fonctionne."