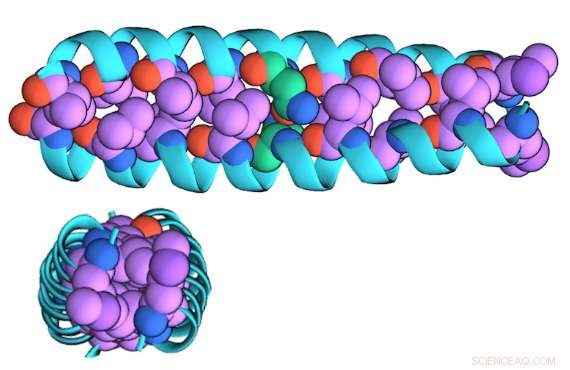
Une illustration de la forme dimère "zippée" de GCN4-p1 vue de côté (en haut) et au-dessus (en bas)
Des chercheurs de l'Université de Pennsylvanie ont fait des progrès vers une nouvelle méthode de séquençage des gènes. Les bases d'un brin d'ADN sont lues lorsqu'elles sont enfilées à travers un trou nanoscopique.
Dans une nouvelle étude, ils ont montré que cette technique peut également être appliquée aux protéines comme moyen d'en savoir plus sur leur structure.
Les méthodes existantes pour ce type d'analyse sont laborieuses, impliquant généralement la collecte de grandes quantités de la protéine. Ils nécessitent aussi souvent de modifier la protéine, limitant l'utilité de ces méthodes pour comprendre le comportement de la protéine à l'état naturel.
La technique de translocation des chercheurs de Penn permet l'étude de protéines individuelles sans les modifier. Des échantillons prélevés sur un seul individu pourraient être analysés de cette façon, ouvrir des applications pour le diagnostic des maladies et la recherche.
L'étude a été dirigée par Marija Drndić, professeur au Département de physique et d'astronomie de l'École des arts et des sciences ; David Niedzwiecki, une chercheuse postdoctorale dans son laboratoire; et Jeffery G. Saven, professeur au département de chimie de Penn Arts &Sciences.
Il a été publié dans la revue ACS Nano .
La technique de l'équipe Penn est issue des travaux de Drndić sur le séquençage des gènes nanopores, qui vise à distinguer les bases d'un brin d'ADN par le pourcentage différent de l'ouverture qu'elles bloquent chacune lorsqu'elles traversent un pore nanoscopique. Différentes silhouettes laissent passer différentes quantités d'un liquide ionique. Le changement de flux d'ions est mesuré par l'électronique entourant le pore; les pics et les creux de ce signal peuvent être corrélés à chaque base.
Alors que les chercheurs s'efforcent d'augmenter la précision de ces lectures à des niveaux utiles, Drndić et ses collègues ont expérimenté l'application de la technique à d'autres molécules biologiques et structures à l'échelle nanométrique.
Collaborer avec le groupe de Saven, ils ont entrepris de tester leurs pores sur des molécules biologiques encore plus délicates.
"Il existe de nombreuses protéines qui sont beaucoup plus petites et plus difficiles à manipuler qu'un brin d'ADN que nous aimerions étudier, " a déclaré Saven. "Nous sommes intéressés à en savoir plus sur la structure d'une protéine donnée, comme s'il existe en tant que monomère, ou combiné avec une autre copie dans un dimère, ou un agrégat de copies multiples connu sous le nom d'oligomère."
La détection est aussi souvent une limitation.
"Il n'y a aucun moyen d'amplifier les peptides et les protéines comme c'est le cas pour l'ADN, " a déclaré Drndić. " Si vous voulez étudier les protéines d'une source particulière, vous êtes coincé avec de très petits échantillons. Avec cette méthode, cependant, vous pouvez simplement collecter la quantité de données dont vous avez besoin et le nombre de protéines que vous souhaitez faire passer à travers le pore, puis les étudier une par une telles qu'elles existent naturellement dans le corps."
En utilisant les nanopores de nitrure de silicium du groupe Drndić, qui peut être percé à des diamètres personnalisés, l'équipe de recherche a entrepris de tester leur technique sur GCN4-p1, une protéine sélectionnée parce qu'elle contient un motif structurel commun trouvé dans les facteurs de transcription et les récepteurs intracellulaires.
"La version dimère est 'zippée' ensemble, " Niedzwiecki a dit, "C'est une" bobine enroulée "d'hélices entrelacées qui est à peu près cylindrique. La version monomère est décompressée et n'est probablement pas hélicoïdale; c'est probablement plus comme une ficelle."
Les chercheurs ont mis différents ratios de versions zippées et décompressées de la protéine dans un fluide ionique et les ont passés à travers les pores. Bien qu'incapable de faire la différence entre les protéines individuelles, les chercheurs pourraient effectuer cette analyse sur des populations de la molécule.
"La forme dimère et monomère de la protéine bloque un nombre différent d'ions, nous voyons donc une baisse de courant différente lorsqu'ils traversent le pore, " a déclaré Niedzwiecki. "Mais nous obtenons une plage de valeurs pour les deux, car tous les événements de translocation moléculaire ne sont pas identiques."
Déterminer si un échantillon spécifique de ces types de protéines s'agrège ou non pourrait être utilisé pour mieux comprendre la progression de la maladie.
« De nombreux chercheurs, " Saven a dit, "ont observé ces longs enchevêtrements de peptides et de protéines agrégés dans des maladies comme la maladie d'Alzheimer et la maladie de Parkinson, mais il y a de plus en plus de preuves qui suggèrent que ces enchevêtrements se produisent après le fait, que ce qui cause vraiment le problème, ce sont des assemblages de protéines plus petits. Déterminer ce que sont ces assemblages et leur taille est actuellement très difficile à faire, donc c'est peut-être une façon de résoudre ce problème."