
SINGLE utilise l'imagerie TEM in situ de nanocristaux de platine en rotation libre dans une cellule liquide de graphène pour déterminer les structures 3-D de nanoparticules colloïdales individuelles. Crédit :Berkeley Lab
Tout comme les protéines sont l'un des éléments de base de la biologie, les nanoparticules peuvent servir de blocs de construction de base pour les matériaux de la prochaine génération. Dans la continuité de ce parallèle entre biologie et nanotechnologie, une technique éprouvée pour déterminer les structures tridimensionnelles de protéines individuelles a été adaptée pour déterminer les structures 3D de nanoparticules individuelles en solution.
Une équipe multi-institutionnelle de chercheurs dirigée par le Lawrence Berkeley National Laboratory (Berkeley Lab) du département américain de l'Énergie (DOE), a développé une nouvelle technique appelée « SINGLE » qui fournit les premières images à l'échelle atomique de nanoparticules colloïdales. CÉLIBATAIRE, qui signifie 3D Structure Identification of Nanoparticles by Graphene Liquid Cell Electron Microscopy, a été utilisé pour reconstruire séparément les structures 3D de deux nanoparticules de platine individuelles en solution.
"La compréhension des détails structurels des nanoparticules colloïdales est nécessaire pour relier nos connaissances sur leur synthèse, mécanismes de croissance, et physiques pour faciliter leur application aux énergies renouvelables, la catalyse et bien d'autres domaines, " déclare Paul Alivisatos, directeur du Berkeley Lab et autorité renommée en nanosciences, qui a mené cette recherche. « Alors que la plupart des études structurelles des nanoparticules colloïdales sont réalisées sous vide une fois la croissance cristalline terminée, notre méthode SINGLE nous permet de déterminer leur structure 3D dans une solution, une étape importante vers l'amélioration de la conception des nanoparticules pour les applications de catalyse et de recherche énergétique."
Alivisatos, qui est également titulaire de la Samsung Distinguished Chair in Nanoscience and Nanotechnology à l'Université de Californie à Berkeley, et dirige le Kavli Energy NanoSciences Institute à Berkeley (Kavli ENSI), est l'auteur correspondant d'un article détaillant cette recherche dans la revue Science . L'article s'intitule "Structure 3D de nanocristaux individuels en solution par microscopie électronique". Les co-auteurs principaux sont Jungwon Park de l'Université Harvard, Hans Elmlund de l'Université Monash en Australie, et et Peter Ercius de Berkeley Lab. Les autres co-auteurs sont Jong Min Yuk, David Limmer, Qian Chen, Kwanpyo Kim, Sang Hoon Han, David Weitz et Alex Zettl.
Les nanoparticules colloïdales sont des amas de centaines à des milliers d'atomes en suspension dans une solution dont les propriétés chimiques et physiques collectives sont déterminées par la taille et la forme des nanoparticules individuelles. Les techniques d'imagerie couramment utilisées pour analyser la structure 3D de cristaux individuels dans un matériau ne peuvent pas être appliquées aux nanomatériaux en suspension, car les particules individuelles dans une solution ne sont pas statiques. La fonctionnalité des protéines est également déterminée par leur taille et leur forme, et les scientifiques qui voulaient imager des structures protéiques en 3D ont été confrontés à un problème similaire. Le problème de l'imagerie des protéines a été résolu par une technique appelée « microscopie cryoélectronique à particule unique, " dans lequel des dizaines de milliers d'images au microscope électronique à transmission (MET) 2D de copies identiques d'une protéine individuelle ou d'un complexe protéique congelé dans des orientations aléatoires sont enregistrées puis combinées par calcul en reconstructions 3D haute résolution. Alivisatos et ses collègues ont utilisé ce concept pour créer leur technique UNIQUE.
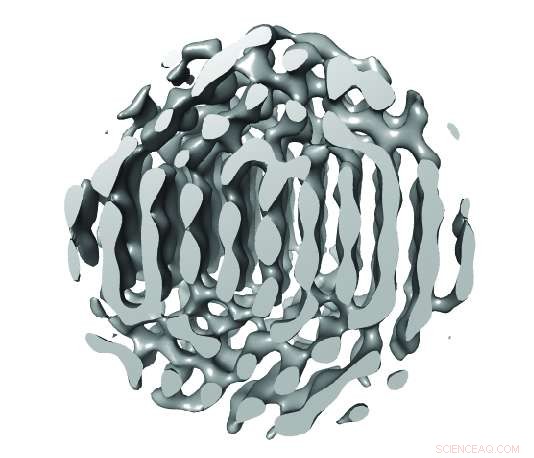
Une dalle à travers la reconstruction 3-D de la particule 1 le long du plan vertical avec des positions atomiques provisoires indiquées. Les répétitions ABC des plans {111} sont visibles. Crédit :Centre d'excellence ARC en imagerie moléculaire avancée
« En science des matériaux, nous ne pouvons pas supposer que les nanoparticules dans une solution sont toutes identiques, nous avons donc dû développer une approche hybride pour reconstruire les structures 3D de nanoparticules individuelles, " déclare le co-auteur principal du Science papier Peter Ercius, membre du personnel scientifique du Centre national de microscopie électronique (NCEM) à la Fonderie moléculaire, une installation utilisateur du DOE Office of Science.
"SINGLE représente une combinaison de trois avancées technologiques de l'imagerie TEM en biologie et science des matériaux, " dit Ercius. " Ces trois avancées sont le développement d'une cellule liquide de graphène qui permet l'imagerie TEM de nanoparticules tournant librement en solution, des détecteurs d'électrons directs capables de produire des films avec une résolution temporelle d'une milliseconde à l'autre des nanocristaux en rotation, et une théorie pour la reconstruction 3D d'une seule particule ab initio."
La cellule liquide au graphène (GLC) qui a permis de rendre cette étude possible a également été développée au Berkeley Lab sous la direction d'Alivisatos. L'imagerie TEM utilise un faisceau d'électrons plutôt que de la lumière pour l'éclairage et le grossissement, mais ne peut être utilisée que dans un vide poussé car les molécules dans l'air perturbent le faisceau d'électrons. Étant donné que les liquides s'évaporent sous vide poussé, les échantillons dans les solutions doivent être hermétiquement scellés dans des conteneurs solides spéciaux - appelés cellules - avec une fenêtre de visualisation très mince avant d'être imagés avec MET. Autrefois, les cellules liquides comportaient des fenêtres de visualisation à base de silicium dont l'épaisseur limitait la résolution et perturbait l'état naturel des matériaux de l'échantillon. Le GLC développé au laboratoire de Berkeley dispose d'une fenêtre de visualisation constituée d'une feuille de graphène qui n'a qu'un seul atome d'épaisseur.
"Le GLC nous fournit un revêtement ultra-mince de nos nanoparticules tout en maintenant des conditions liquides dans le vide MET, " dit Ercius. " Puisque la surface de graphène du GLC est inerte, il n'adsorbe pas ou ne perturbe pas l'état naturel de nos nanoparticules."
Travaillant au sein de l'ÉQUIPE I du NCEM, le microscope électronique le plus puissant au monde, Ercius, Alivisatos et leurs collègues ont pu imager in situ les mouvements de translation et de rotation de nanoparticules individuelles de platine d'un diamètre inférieur à deux nanomètres. Les nanoparticules de platine ont été choisies en raison de leur force de diffusion élevée des électrons et parce que leur structure atomique détaillée est importante pour la catalyse.
"Nos précédentes études GLC sur les nanocristaux de platine ont montré qu'ils se développent par agrégation, résultant en des structures complexes qu'il n'est possible de déterminer par aucune méthode développée précédemment, " dit Ercius. " Puisque SINGLE tire ses structures 3D d'images de nanoparticules individuelles tournant librement en solution, il permet l'analyse de populations hétérogènes de nanoparticules potentiellement non ordonnées qui sont synthétisées en solution, fournissant ainsi un moyen de comprendre la structure et la stabilité des défauts à l'échelle nanométrique."
La prochaine étape pour SINGLE consiste à récupérer une carte de densité de résolution atomique 3D complète des nanoparticules colloïdales à l'aide d'une caméra plus avancée installée sur TEAM I qui peut fournir 400 images par seconde et une meilleure qualité d'image.
"Nous prévoyons d'imager des défauts dans des nanoparticules faites de différents matériaux, particules de coquille de noyau, et aussi des alliages constitués de deux espèces atomiques différentes, " dit Ercius.