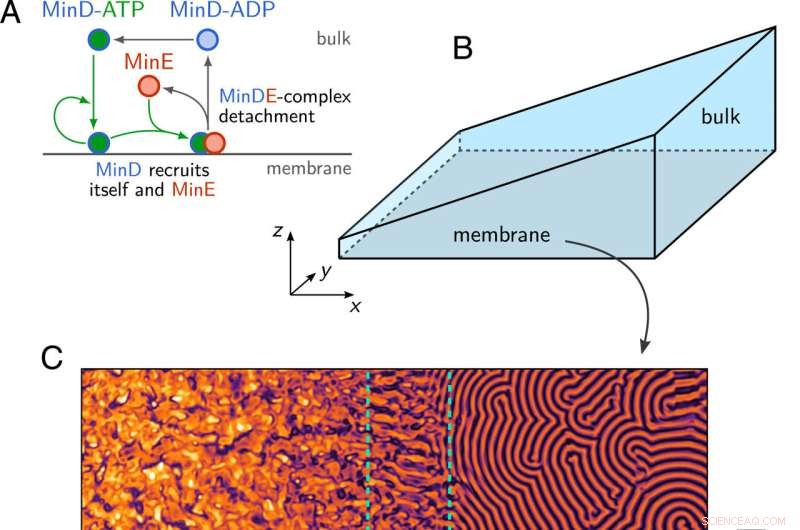
(A) Illustration schématique du réseau de réaction protéique Min. (B) Géométrie de coin avec une surface de membrane au plan inférieur (z =0) et une hauteur de masse H(x) augmentant linéairement le long de la direction x. (C) Instantané de la densité membranaire de MinD, obtenu en simulant numériquement la dynamique Min Eqs. 1–3 dans la géométrie illustrée en B. On observe des régions avec des motifs chaotiques, des ondes stationnaires (SW, contour vert en pointillés) et des ondes progressives (TW) le long de la membrane et à différentes hauteurs de volume. Crédit :Actes de l'Académie nationale des sciences (2022). DOI :10.1073/pnas.2206888119
Pour de nombreux processus importants pour la vie tels que la division cellulaire, la migration cellulaire et le développement des organes, la formation spatialement et temporellement correcte de modèles biologiques est essentielle. Pour comprendre ces processus, la tâche principale ne consiste pas à expliquer comment des motifs se forment à partir d'une condition initiale homogène, mais à expliquer comment des motifs simples se transforment en motifs de plus en plus complexes. Éclairer les mécanismes de cette auto-organisation complexe à différentes échelles spatiales et temporelles est un enjeu clé pour la science.
Les techniques dites de "gros grain" permettent de simplifier de tels systèmes multi-échelles, de sorte qu'ils peuvent être décrits avec un modèle réduit à de grandes échelles de longueur et de temps. « Le prix que vous payez pour le grain grossier, cependant, est que des informations importantes sur les modèles à petite échelle – comme le type de modèle – sont perdues. Mais le fait est que ces modèles jouent un rôle décisif dans les systèmes biologiques. Pour donner un exemple , ils contrôlent d'importants processus cellulaires », explique Laeschkir Würthner, membre de l'équipe dirigée par le professeur Erwin Frey, physicien du LMU, et auteur principal d'une nouvelle étude publiée dans les Proceedings of the National Academy of Sciences qui surmonte ce problème.
En collaboration avec le groupe de recherche du professeur Cees Dekker (TU Delft), l'équipe de Frey a développé une nouvelle approche à gros grains pour les systèmes de réaction-diffusion dits à conservation de masse, dans lesquels l'analyse à grande échelle des densités totales de les particules impliquées permettent de prédire des motifs à petite échelle.
Les scientifiques ont illustré le potentiel de leur approche avec le système protéique Min, un modèle paradigmatique pour la formation de modèles biologiques. La bactérie E. coli utilise diverses protéines Min circulant dans une cellule pour déterminer à quel endroit la division cellulaire a lieu. Un facteur décisif ici est que les protéines impliquées se produisent à des fréquences différentes en fonction de leur emplacement dans la cellule et de l'état chimique, c'est-à-dire qu'elles ont une variété de densités différentes.
"Nous avons maintenant réussi à réduire la complexité de ce système en développant une théorie basée uniquement sur les densités totales des protéines, de sorte que nous pouvons refléter complètement la dynamique de formation des motifs", explique Frey. "C'est une énorme réduction. Les calculs numériques sont maintenant accomplis en quelques minutes au lieu de plusieurs mois."
Les chercheurs ont pu confirmer expérimentalement les prédictions théoriques du modèle, selon lesquelles la distribution des protéines dépend de la géométrie de l'environnement. Pour ce faire, ils ont reconstruit le système protéique Min dans une cellule à flux in vitro, les résultats montrant les mêmes modèles de protéines que ceux révélés dans la simulation.
"Une telle reconstruction d'informations à petite échelle à partir d'une dynamique réduite au niveau macroscopique ouvre de nouvelles voies pour une meilleure compréhension des systèmes multi-échelles complexes, qui se produisent dans un large éventail de systèmes physiques", explique Frey. Les systèmes de formation de motifs biologiques sont mieux caractérisés par la géométrie que par les simulations