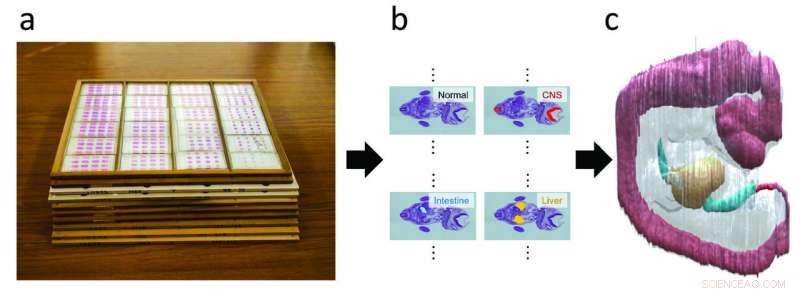
Un ensemble de coupes en série histologiques d'un embryon humain (a) avec des annotations d'organes (b) et une reconstruction 3D (c). Crédit :Kajihara et al. 2019
Malgré les avancées de l'imagerie 3D telles que l'IRM et la tomodensitométrie, les scientifiques comptent toujours sur le découpage d'un spécimen en sections 2D pour acquérir les informations les plus détaillées. En utilisant ces informations, ils essaient ensuite de reconstruire une image 3D de l'échantillon. Des chercheurs de l'Institut des sciences et technologies de Nara rapportent un nouvel algorithme qui peut effectuer cette tâche à moindre coût et avec une plus grande robustesse que les méthodes standard.
Des scientifiques japonais rapportent en La reconnaissance de formes une nouvelle méthode pour construire des modèles 3D à partir d'images 2D. L'approche, qui implique un recalage non rigide avec un mélange de transformées rigides, surmonte plusieurs des limitations des méthodes actuelles. Les chercheurs valident leur méthode en l'appliquant à la collection Kyoto d'embryons et de fœtus humains, la plus grande collection d'embryons humains au monde, avec plus de 45 ans, 000 exemplaires.
L'IRM et la tomodensitométrie sont des techniques standard pour acquérir des images 3D du corps. Ces modalités peuvent tracer avec une précision sans précédent l'emplacement d'une blessure ou d'un accident vasculaire cérébral. Ils peuvent même révéler les dépôts de protéines microscopiques observés dans des pathologies cérébrales comme la maladie d'Alzheimer. Cependant, pour la meilleure résolution, les scientifiques dépendent toujours de tranches du spécimen, c'est pourquoi le cancer et d'autres biopsies sont prises. Une fois les informations souhaitées acquises, les scientifiques utilisent des algorithmes qui peuvent assembler les tranches 2D pour recréer une image 3D simulée. De cette façon, ils peuvent reconstruire un organe entier ou même un organisme.
Empiler des tranches pour créer une image 3D revient à assembler un gâteau après l'avoir coupé. Oui, la forme générale est là, mais le couteau cassera certaines tranches de sorte que le gâteau reconstitué ne sera jamais aussi beau que l'original. Bien que cela puisse ne pas déranger la fête des enfants de cinq ans qui veulent se faire plaisir, le parti des chirurgiens à la recherche de la localisation précise d'une tumeur est plus difficile à apaiser.
En réalité, le spécimen peut subir une série de changements lorsqu'il est préparé pour la coupe. "Le processus de sectionnement s'étire, plie et déchire le tissu. Le processus de coloration varie selon les échantillons. Et le processus de fixation provoque la destruction des tissus, " explique l'Institut des sciences et technologies de Nara (NAIST), Nara, Japon, Professeur agrégé Takuya Funatomi, qui a mené le projet.
Fondamentalement, il y a trois défis qui émergent avec la reconstruction 3-D. La première est la déformation non rigide, dans laquelle la position et l'orientation de divers points de l'échantillon d'origine ont changé. Deuxièmement, la discontinuité tissulaire, où des lacunes peuvent apparaître dans la reconstruction si l'enregistrement échoue. Finalement, il y a un changement d'échelle, où des portions de la reconstruction sont disproportionnées par rapport à leur taille réelle en raison d'un enregistrement non rigide.
Pour chacun de ces problèmes, Funatomi et son équipe de recherche ont proposé une solution qui, une fois combinée, a entraîné une reconstruction qui minimise les trois facteurs en utilisant moins de coûts de calcul que les méthodes standard.
"D'abord, nous représentons une déformation non rigide en utilisant un petit nombre de points de contrôle en mélangeant des transformées rigides, " dit Funatomi. Le petit nombre de points de contrôle peut être estimé de manière robuste par rapport à la variation de coloration.
"Ensuite, nous sélectionnons les images cibles en fonction des résultats d'enregistrement non rigides et appliquons un ajustement d'échelle, " il continue.
La nouvelle méthode se concentre principalement sur un certain nombre d'images de coupes en série d'embryons humains de la collection d'embryons et de fœtus humains de Kyoto et pourrait reconstruire des embryons en 3D avec un succès extraordinaire.
Notamment, il n'y a pas d'IRM ou de tomodensitométrie des échantillons, ce qui signifie qu'aucun modèle 3-D ne peut être utilisé comme référence pour la reconstruction 3-D. Plus loin, la grande variabilité des lésions tissulaires et de la coloration a compliqué la reconstruction.
"Notre méthode pouvait décrire une déformation complexe avec un plus petit nombre de points de contrôle et était robuste à une variation de coloration, " dit Funatomi.