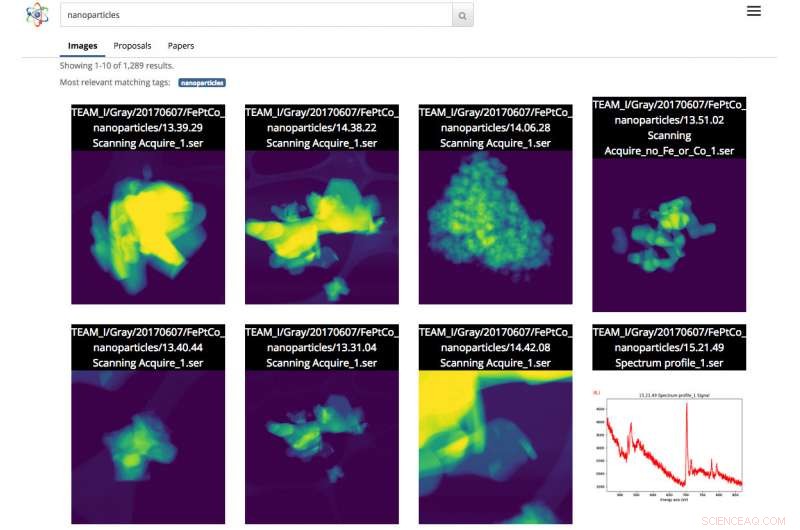
Capture d'écran de l'interface de recherche scientifique. Dans ce cas, l'utilisateur a fait une recherche d'images de nanoparticules. Crédit :Gonzalo Rodrigo, Laboratoire de Berkeley
À mesure que les ensembles de données scientifiques augmentent à la fois en taille et en complexité, la capacité d'étiqueter, filtrer et rechercher ce déluge d'informations est devenu une tâche laborieuse, tâche chronophage et parfois impossible, sans l'aide d'outils automatisés.
Avec ça en tête, une équipe de chercheurs du Lawrence Berkeley National Laboratory (Berkeley Lab) et de l'UC Berkeley développent des outils d'apprentissage automatique innovants pour extraire des informations contextuelles d'ensembles de données scientifiques et générer automatiquement des balises de métadonnées pour chaque fichier. Les scientifiques peuvent ensuite rechercher ces fichiers via un moteur de recherche Web pour les données scientifiques, appelé Recherche scientifique, que l'équipe de Berkeley est en train de construire.
Comme preuve de concept, l'équipe travaille avec le personnel de la fonderie moléculaire du ministère de l'Énergie (DOE), situé au Berkeley Lab, pour démontrer les concepts de Science Search sur les images capturées par les instruments de l'installation. Une version bêta de la plateforme a été mise à la disposition des chercheurs de Foundry.
"Un outil comme Science Search a le potentiel de révolutionner notre recherche, " dit Colin Ophus, un chercheur scientifique en fonderie moléculaire au sein du Centre national de microscopie électronique (NCEM) et un collaborateur de recherche scientifique. « Nous sommes une facilité nationale d'utilisation financée par les contribuables, et nous souhaitons rendre toutes les données largement disponibles, plutôt que le petit nombre d'images choisies pour la publication. Cependant, aujourd'hui, la plupart des données collectées ici ne sont vraiment examinées que par une poignée de personnes - les producteurs de données, y compris le PI (chercheur principal), leurs post-doctorants ou étudiants diplômés, car il n'existe actuellement aucun moyen simple de passer au crible et de partager les données. En rendant ces données brutes facilement consultables et partageables, par Internet, Science Search pourrait ouvrir ce réservoir de « données obscures » à tous les scientifiques et maximiser l'impact scientifique de notre installation. »
Les défis de la recherche de données scientifiques
Aujourd'hui, Les moteurs de recherche sont omniprésents pour trouver des informations sur Internet, mais la recherche de données scientifiques présente un autre ensemble de défis. Par exemple, L'algorithme de Google s'appuie sur plus de 200 indices pour réaliser une recherche efficace. Ces indices peuvent se présenter sous forme de mots clés sur une page web, des métadonnées dans des images ou des commentaires d'audience de milliards de personnes lorsqu'elles cliquent sur les informations qu'elles recherchent. En revanche, les données scientifiques se présentent sous de nombreuses formes qui sont radicalement différentes d'une page Web moyenne, nécessite un contexte spécifique à la science et manque souvent de métadonnées pour fournir le contexte requis pour des recherches efficaces.
Dans les installations nationales des utilisateurs comme la fonderie moléculaire, des chercheurs du monde entier demandent du temps et se rendent ensuite à Berkeley pour utiliser gratuitement des instruments extrêmement spécialisés. Ophus note que les caméras actuelles des microscopes de la fonderie peuvent collecter jusqu'à un téraoctet de données en moins de 10 minutes. Les utilisateurs doivent ensuite passer au crible manuellement ces données pour trouver des images de qualité avec une "bonne résolution" et enregistrer ces informations sur un système de fichiers partagé sécurisé, comme Dropbox, ou sur un disque dur externe qu'ils emportent finalement avec eux pour analyser.
Souvent, les chercheurs qui viennent à la Molecular Foundry n'ont que quelques jours pour collecter leurs données. Parce qu'il est très fastidieux et chronophage d'ajouter manuellement des notes à des téraoctets de données scientifiques et qu'il n'y a pas de norme pour le faire, la plupart des chercheurs tapent simplement des descriptions abrégées dans le nom de fichier. Cela peut avoir du sens pour la personne qui enregistre le fichier, mais n'a souvent pas beaucoup de sens pour quelqu'un d'autre.
"L'absence d'étiquettes de métadonnées réelles finit par poser des problèmes lorsque le scientifique essaie de trouver les données plus tard ou tente de les partager avec d'autres, " dit Lavanya Ramakrishnan, membre du personnel scientifique de la division de recherche informatique (CRD) du Berkeley Lab et co-chercheur principal du projet Science Search. "Mais avec les techniques d'apprentissage automatique, on peut se faire aider par les ordinateurs pour ce qui est laborieux pour les utilisateurs, y compris l'ajout de balises aux données. Ensuite, nous pouvons utiliser ces balises pour rechercher efficacement les données."
Pour résoudre le problème des métadonnées, l'équipe du Berkeley Lab utilise des techniques d'apprentissage automatique pour exploiter « l'écosystème scientifique », y compris les horodatages des instruments, journaux des utilisateurs de l'installation, propositions scientifiques, publications et structures de système de fichiers—pour des informations contextuelles. Les informations collectives provenant de ces sources, y compris l'horodatage de l'expérience, des notes sur la résolution et le filtre utilisés et la demande de temps de l'utilisateur, tous fournissent des informations contextuelles critiques. L'équipe du laboratoire de Berkeley a mis en place une pile logicielle innovante qui utilise des techniques d'apprentissage automatique, notamment le traitement du langage naturel, extrait des mots-clés contextuels sur l'expérience scientifique et crée automatiquement des balises de métadonnées pour les données.
Pour la preuve de concept, Ophus a partagé les données du microscope électronique TEAM 1 de la fonderie moléculaire au NCEM qui ont été récemment collectées par le personnel de l'installation, avec l'équipe de recherche scientifique. Il s'est également porté volontaire pour étiqueter quelques milliers d'images pour donner aux outils d'apprentissage automatique des étiquettes à partir desquelles commencer à apprendre. Bien que ce soit un bon début, Le co-chercheur principal de Science Search, Gunther Weber, note que la plupart des applications d'apprentissage automatique réussies nécessitent généralement beaucoup plus de données et de commentaires pour obtenir de meilleurs résultats. Par exemple, dans le cas de moteurs de recherche comme Google, Weber note que des ensembles de données de formation sont créés et que les techniques d'apprentissage automatique sont validées lorsque des milliards de personnes dans le monde vérifient leur identité en cliquant sur toutes les images avec des panneaux de signalisation ou des vitrines après avoir tapé leurs mots de passe, ou sur Facebook lorsqu'ils taguent leurs amis dans une image.
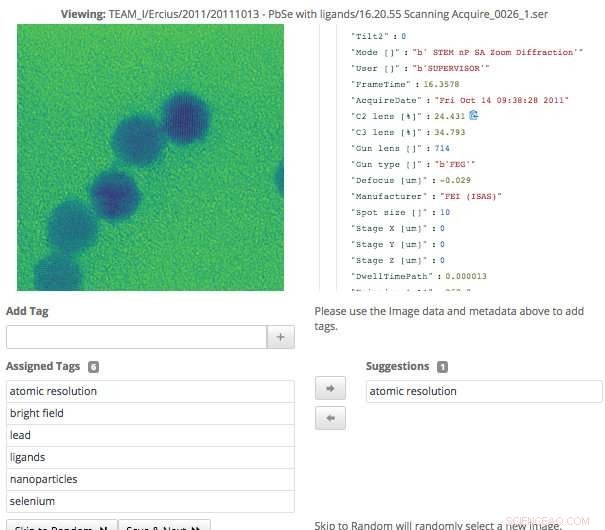
Cette capture d'écran de l'interface Science Search montre comment les utilisateurs peuvent facilement valider les balises de métadonnées qui ont été générées via l'apprentissage automatique, ou ajouter des informations qui n'ont pas encore été capturées. Crédit :Gonzalo Rodrigo, Laboratoire de Berkeley
"Dans le cas des données scientifiques, seule une poignée d'experts du domaine peuvent créer des ensembles de formation et valider des techniques d'apprentissage automatique, donc l'un des gros problèmes actuels auxquels nous sommes confrontés est un nombre extrêmement petit d'ensembles d'entraînement, " dit Weber, qui est également membre du personnel scientifique du CRD de Berkeley Lab.
Pour surmonter ce défi, les chercheurs du Berkeley Lab ont utilisé l'apprentissage par transfert pour limiter les degrés de liberté, ou nombre de paramètres, sur leurs réseaux de neurones convolutifs (CNN). L'apprentissage par transfert est une méthode d'apprentissage automatique dans laquelle un modèle développé pour une tâche est réutilisé comme point de départ d'un modèle sur une deuxième tâche, ce qui permet à l'utilisateur d'obtenir des résultats plus précis à partir d'un ensemble d'entraînement plus petit. Dans le cas du microscope TEAM I, les données produites contiennent des informations sur le mode de fonctionnement dans lequel se trouvait l'instrument au moment de la collecte. Avec ces informations, Weber a pu former le réseau de neurones sur cette classification afin qu'il puisse générer automatiquement cette étiquette de mode de fonctionnement. Il a ensuite gelé cette couche convolutive du réseau, ce qui signifiait qu'il n'aurait qu'à recycler les couches densément connectées. Cette approche réduit efficacement le nombre de paramètres sur le CNN, permettant à l'équipe d'obtenir des résultats significatifs à partir de leurs données d'entraînement limitées.
L'apprentissage automatique pour exploiter l'écosystème scientifique
En plus de générer des balises de métadonnées via des ensembles de données d'entraînement, l'équipe du Berkeley Lab a également développé des outils qui utilisent des techniques d'apprentissage automatique pour extraire l'écosystème scientifique du contexte des données. Par exemple, le module d'ingestion de données peut examiner une multitude de sources d'informations de l'écosystème scientifique, y compris les horodatages des instruments, journaux d'utilisateurs, propositions et publications—et identifier les points communs. Les outils développés au Berkeley Lab qui utilisent des méthodes de traitement du langage naturel peuvent ensuite identifier et classer les mots qui donnent un contexte aux données et facilitent des résultats significatifs pour les utilisateurs plus tard. L'utilisateur verra quelque chose de similaire à la page de résultats d'une recherche sur Internet, où le contenu avec le plus de texte correspondant aux mots de recherche de l'utilisateur apparaîtra plus haut sur la page. Le système apprend également des requêtes des utilisateurs et des résultats de recherche sur lesquels ils cliquent.
Parce que les instruments scientifiques génèrent un corpus de données sans cesse croissant, tous les aspects du moteur de recherche scientifique de l'équipe de Berkeley devaient être évolutifs pour suivre le rythme et l'échelle des volumes de données produits. L'équipe y est parvenue en installant son système dans une instance Spin sur le supercalculateur Cori du National Energy Research Scientific Computing Center (NERSC). Spin est une technologie de services de périphérie basée sur Docker et développée au NERSC qui peut accéder aux systèmes informatiques et au stockage haute performance de l'installation sur le back-end.
"L'une des raisons pour lesquelles il nous est possible de créer un outil comme Science Search est notre accès aux ressources du NERSC, " dit Gonzalo Rodrigo, un chercheur postdoctoral du Berkeley Lab qui travaille sur les défis du traitement du langage naturel et de l'infrastructure dans la recherche scientifique. "Nous devons stocker, analyser et récupérer des ensembles de données très volumineux, et il est utile d'avoir accès à une installation de supercalcul pour faire le gros du travail pour ces tâches. Spin de NERSC est une excellente plate-forme pour exécuter notre moteur de recherche qui est une application destinée à l'utilisateur qui nécessite l'accès à de grands ensembles de données et à des données analytiques qui ne peuvent être stockées que sur de grands systèmes de stockage de supercalcul.
Une interface de validation et de recherche de données
Lorsque l'équipe du Berkeley Lab a développé l'interface permettant aux utilisateurs d'interagir avec leur système, ils savaient qu'il devrait accomplir quelques objectifs, y compris une recherche efficace et permettant une entrée humaine dans les modèles d'apprentissage automatique. Étant donné que le système s'appuie sur des experts du domaine pour aider à générer les données d'entraînement et valider la sortie du modèle d'apprentissage automatique, l'interface nécessaire pour faciliter cela.
"L'interface de marquage que nous avons développée affiche les données originales et les métadonnées disponibles, ainsi que toutes les balises générées par machine que nous avons jusqu'à présent. Les utilisateurs experts peuvent ensuite parcourir les données et créer de nouvelles balises et vérifier la précision de toutes les balises générées par la machine, " dit Matt Henderson, qui est ingénieur en systèmes informatiques au CRD et dirige l'effort de développement de l'interface utilisateur.
Pour faciliter une recherche efficace des utilisateurs sur la base des informations disponibles, l'interface de recherche de l'équipe fournit un mécanisme de requête pour les fichiers disponibles, propositions et articles dont les outils d'apprentissage automatique développés par Berkeley ont analysé et extrait les balises. Chaque élément de résultat de recherche répertorié représente un résumé de ces données, avec une vue secondaire plus détaillée disponible, y compris des informations sur les balises qui correspondent à cet élément. L'équipe étudie actuellement la meilleure façon d'intégrer les commentaires des utilisateurs pour améliorer les modèles et les balises.
« Avoir la capacité d'explorer des ensembles de données est important pour les percées scientifiques, et c'est la première fois que quelque chose comme Science Search a été tenté, ", déclare Ramakrishnan. "Notre vision ultime est de jeter les bases qui soutiendront à terme un 'Google' pour les données scientifiques, où les chercheurs peuvent même rechercher des ensembles de données distribués. Notre travail actuel fournit les bases nécessaires pour parvenir à cette vision ambitieuse. »
"Berkeley Lab est vraiment un endroit idéal pour créer un outil comme Science Search parce que nous avons un certain nombre d'installations pour les utilisateurs, comme la Fonderie Moléculaire, qui ont des décennies de données qui fourniraient encore plus de valeur à la communauté scientifique si les données pouvaient être recherchées et partagées, " ajoute Katie Antypas, qui est le chercheur principal de Science Search et chef du département des données du NERSC. « De plus, nous avons un excellent accès à l'expertise en apprentissage automatique dans le domaine des sciences informatiques du Berkeley Lab ainsi qu'aux ressources HPC du NERSC afin de développer ces capacités. »