Une équipe de recherche du département de chimie de l'Université de Hambourg a réussi pour la première fois à identifier au niveau moléculaire le mécanisme dynamique utilisé par l'enzyme RNase R pour dégrader la sous-unité ribosomale 30S. Les résultats de l'étude ont été publiés dans la revue scientifique Nature .
La synthèse des protéines est un processus vital et gourmand en énergie dans la cellule dans lequel les ribosomes jouent un rôle crucial. Ces molécules relativement grosses se trouvent dans tous les organismes vivants et agissent comme des « usines à protéines » des cellules.
Pour ce faire, les ribosomes lisent le modèle d’une protéine spécifique sur une molécule messagère – l’ARN messager (ARNm) – puis convertissent cette information en une nouvelle protéine. Les ribosomes sont constitués de deux sous-unités. La petite sous-unité est responsable de la lecture et de la vérification des erreurs dans l'ARNm, tandis que la grande sous-unité est responsable de la polymérisation des acides aminés pour former des protéines.
Une production contrôlée et un renouvellement régulé des ribosomes sont nécessaires à la synthèse des protéines. Bien que l'assemblage des ribosomes ait été de mieux en mieux compris ces dernières années, il n'existe aucun aperçu structurel de la dégradation des ribosomes.
Ceci est important car dans des situations de stress comme le manque de nourriture ou à la fin de leur cycle de croissance, les cellules réduisent leur métabolisme afin de survivre plus longtemps. Cet état est appelé phase stationnaire. Durant cette phase, la synthèse des protéines énergivores est réduite, et certains ribosomes sont dégradés afin de libérer l'énergie investie en eux pour assurer la survie cellulaire.
Pour leurs investigations, les chercheurs ont étudié Bacillus subtilis, une bactérie du sol en forme de bâtonnet que l'on trouve dans l'air, la poussière et l'eau ainsi que dans les intestins des humains et des animaux. "Contrairement aux études précédentes, nous avons pris des cellules qui étaient encore en croissance et qui n'étaient pas en phase stationnaire. Nous voulions savoir quels processus se déroulent lors du passage à la phase stationnaire", explique le Dr Helge Paternoga du département de chimie de l'Université de Vienne. Universität Hamburg, dernier auteur de l'étude.
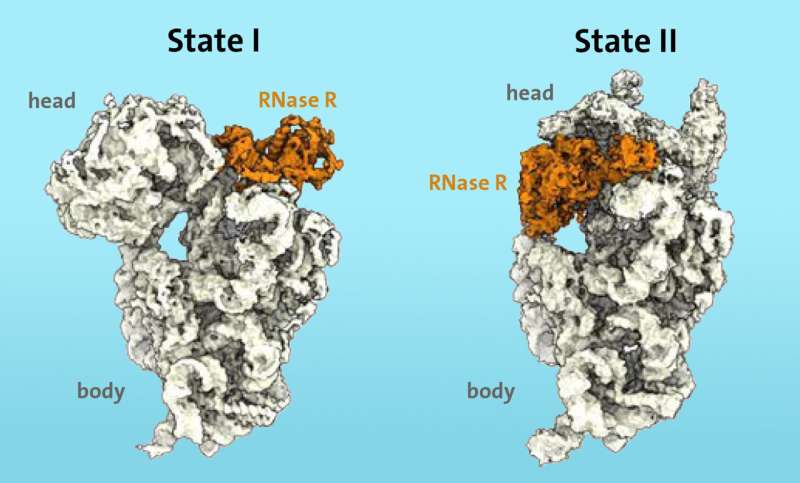
Les chercheurs savaient grâce à des travaux antérieurs que certaines enzymes, comme la ribonucléase R (RNase R), sont impliquées dans le processus de dégradation des ribosomes en situation de stress. Grâce à la cryomicroscopie électronique, ils ont pu montrer pour la première fois que l'enzyme RNase R se lie à la petite sous-unité 30S du ribosome. Le "S" signifie "unités Svedberg" et fait référence à la masse de la sous-unité ribosomale.
La RNase R ne coupe pas arbitrairement la sous-unité 30S, mais s'attache plutôt à une zone libre, que les chercheurs appellent le « cou », puis détache la « tête », la zone supérieure de la sous-unité, en deux étapes consécutives. /P>
"Dans la première étape, l'enzyme RNase R rencontre un obstacle au niveau du 'cou' et déstabilise la zone du cou, la rendant plus flexible. Dans la deuxième étape, la 'tête' est tournée, ce qui supprime l'obstacle et permet à l'enzyme de poursuivre le processus de dégradation de la sous-unité 30S sans entrave", explique Paternoga.
"Nos expériences de dégradation in vitro indiquent que l'interrupteur de tête constitue une barrière cinétique importante pour la RNase R. De plus, nous avons pu montrer que l'enzyme seule est suffisante pour accomplir le processus complet de dégradation du 30S", explique le professeur Daniel. Wilson, chef du groupe de recherche au Département de chimie de l'Université de Hambourg et co-auteur de l'étude.
Plus d'informations : Lyudmila Dimitrova-Paternoga et al, Base structurelle de la dégradation de la sous-unité ribosomale 30S par la RNase R, Nature (2024). DOI :10.1038/s41586-024-07027-6
Fourni par l'Université de Hambourg