Une nouvelle étude de l’Université de Tel Aviv a découvert qu’une pratique connue des technologies de l’information peut également être appliquée à la chimie. Les chercheurs ont découvert que pour améliorer l'échantillonnage dans les simulations chimiques, il suffit d'arrêter et de redémarrer.
La recherche a été dirigée par un doctorat. étudiant Ofir Blumer, en collaboration avec le professeur Shlomi Reuveni et le Dr Barak Hirshberg de l'École de chimie Sackler de l'Université de Tel Aviv. L'étude a été publiée dans Nature Communications .
Les chercheurs expliquent que les simulations de dynamique moléculaire sont comme un microscope virtuel. Ils suivent le mouvement de tous les atomes dans les systèmes chimiques, physiques et biologiques, tels que les protéines, les liquides et les cristaux. Ils donnent un aperçu de divers processus et ont différentes applications technologiques, y compris la conception de médicaments.
Cependant, ces simulations sont limitées à des processus plus lents qu’un millionième de seconde et ne peuvent donc pas décrire des processus plus lents tels que le repliement des protéines et la nucléation des cristaux. Cette limitation, connue sous le nom de problème d'échelle de temps, constitue un défi de taille dans le domaine.
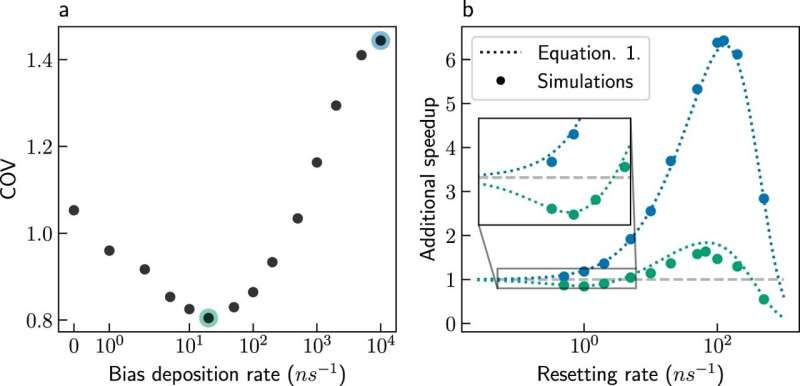
doctorat L'étudiant Ofir Blumer déclare :« Dans notre nouvelle étude, nous montrons que le problème d'échelle de temps peut être surmonté par une réinitialisation stochastique des simulations. Cela semble contre-intuitif à première vue :comment les simulations peuvent-elles se terminer plus rapidement lorsqu'elles sont redémarrées ? Pourtant, il s'avère que cette réaction les temps varient considérablement entre les simulations. Dans certaines simulations, les réactions se produisent rapidement, mais d'autres simulations se perdent dans des états intermédiaires pendant de longues périodes. "
Les chercheurs ont également combiné la réinitialisation stochastique avec la métadynamique, une méthode populaire pour accélérer les simulations de processus chimiques lents. La combinaison permet une plus grande accélération que l’une ou l’autre méthode séparément. De plus, la métadynamique s'appuie sur des connaissances préalables :les coordonnées de la réaction doivent être connues pour accélérer la simulation.
La combinaison de la métadynamique avec la réinitialisation réduit considérablement la dépendance aux connaissances préalables, ce qui fait gagner du temps aux praticiens de la méthode. Enfin, les chercheurs ont montré que cette combinaison permettait de prédire plus précisément la vitesse des processus lents. La méthode combinée a été utilisée pour améliorer avec succès les simulations de repliement de protéines dans l'eau, et elle devrait être appliquée à davantage de systèmes à l'avenir.
Plus d'informations : Ofir Blumer et al, Combiner la réinitialisation stochastique avec la métadynamique pour accélérer les simulations de dynamique moléculaire, Nature Communications (2024). DOI : 10.1038/s41467-023-44528-w
Informations sur le journal : Communications naturelles
Fourni par l'Université de Tel-Aviv