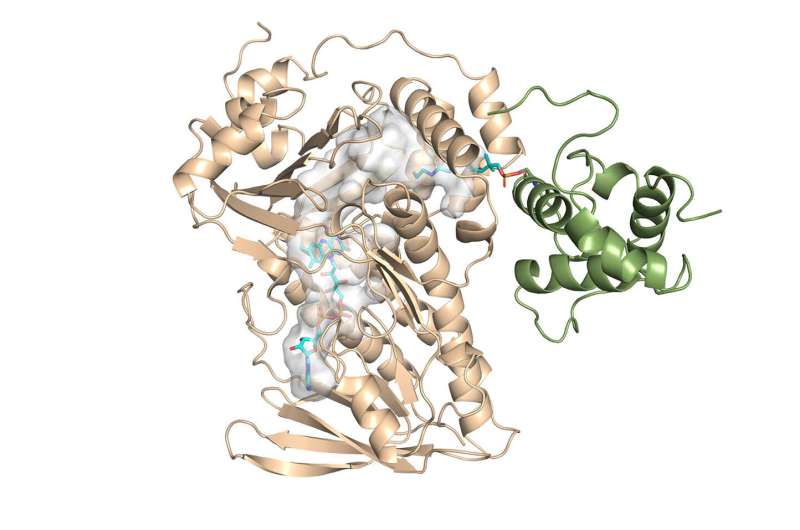
Les chimistes ont déterminé pour la première fois la structure cristalline d'un composant clé de l'enzyme monensine et ont déverrouillé le mécanisme de son activité réactionnelle.
"La principale découverte a été la première structure cristalline de cette famille d'enzymes", a déclaré Chu-Young Kim, professeur de biochimie à l'Université de l'Illinois à Urbana-Champaign (UIUC), qui a dirigé le volet expérimental de l'étude. Lui et ses collègues ont résolu la structure cristalline du MonCI, une enzyme clé dans les bactéries du sol qui synthétise naturellement le monensin.
Lela Vukovic, professeure agrégée à l'Université du Texas à El Paso (UTEP), a réalisé les études informatiques sur la recherche sur le monensin, publiées dans Nature Communications .
L'initiative de cyberinfrastructure de recherche de l'Université du Texas (UTRC) a attribué des allocations de supercalculateurs Vukovic sur le système Lonestar6 du Texas Advanced Computing Center (TACC) pour relever ces défis. L'UTRC fournit des capacités informatiques avancées aux chercheurs des 14 institutions du système UT.
Lonestar6 du TACC a contribué à révéler la séquence de réactions qui produit le monensin. Cette recherche ouvre la porte à la conception future d'antibiotiques plus sûrs et plus efficaces.
"Nous avons découvert que MonCI est utilisé pour effectuer trois réactions d'époxydation cruciales", a déclaré Kim. "C'est très inhabituel et cela a des implications sur la façon dont nous pouvons concevoir la bactérie pour produire de nouveaux antibiotiques."
Kim, qui a récemment quitté l'UTEP pour rejoindre l'UIUC, a consulté le laboratoire de Vukovic avec les résultats de la structure et plein de nouvelles questions. Ce qu’il a découvert était une réaction séquentielle intéressante à l’intérieur de l’enzyme. Cependant, il était encore expérimentalement impossible d'obtenir la structure cristalline de l'enzyme avec le substrat à l'intérieur en mode actif.
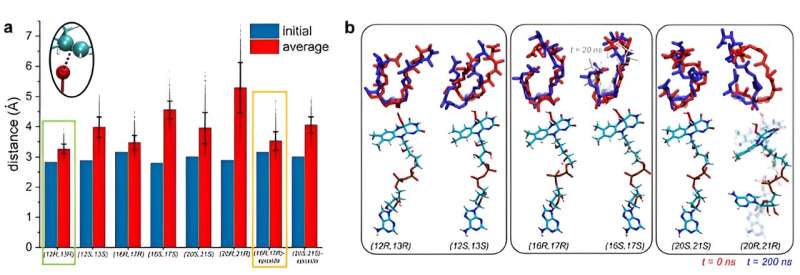
C'est alors que Kim et Vukovic ont décidé de modéliser l'enzyme et le sous-état dans des simulations lorsque le substrat est stable. Si un substrat est stable dans une certaine position, alors une réaction peut se produire pour cette position.
Vukovic et ses étudiants Tara Nitka et Anju Yadav ont développé des modèles complets du système d'environ 78 000 atomes basés sur les structures cristallines déterminées par les expérimentateurs de l'étude.
"Les défis informatiques sont nés de l'examen de plusieurs systèmes pour déterminer la position dans laquelle la prémonensine A et ses versions époxydées sont les plus stables et les plus susceptibles de subir des première, deuxième et troisième réactions d'époxydation", a déclaré Vukovic.
"Les superordinateurs ont été très utiles pour caractériser ces molécules biologiques qui produisent naturellement des antibiotiques", a ajouté Vukovic. "Nous ne serions pas en mesure de réaliser des études informatiques sans eux. Les études informatiques nous aident à découvrir et à comprendre ces réactions séquentielles complexes importantes pour la société."
Vukovic a effectué ses recherches postdoctorales à l'UIUC sous la direction de feu Klaus Schulten, dont l'héritage perdure dans le logiciel NAMD développé par son groupe et utilisé dans cette étude et dans d'innombrables autres.
"L'UIUC fait beaucoup de travail pour optimiser le NAMD afin qu'il fonctionne sur les superordinateurs du pays, tels que Lonertar6 et Stampede2 du TACC", a-t-elle déclaré. "NAMD nous a permis de zoomer sur cette enzyme et de voir quelles réactions se produisent en premier, en deuxième et en troisième pour générer du monensine."
"La biosynthèse du monensine nécessite au moins 14 enzymes différentes, dont MonCI", a ajouté Kim. "Nous devons également étudier toutes les autres enzymes. À l'avenir, nous espérons générer des versions améliorées du monensin pour mieux prendre soin du bétail et de la volaille. De plus, le monensin est toxique pour les chevaux et les chiens, de sorte que ces animaux de ferme sont parfois accidentellement empoisonnés. et tué. Par conséquent, un monensin non toxique est nécessaire."
Les auteurs sont Qian Wang, Tara A. Nitka, Anju Yadav et Lela Vukovic de l'Université du Texas à El Paso; Ning Liu, Hongli Xiao, Hui Yang et Xi Chen de l'Université du Nord-Ouest, Chine ; Irimpan I. Mathews du Laboratoire national des accélérateurs du SLAC ; et Chu-Young Kim de l'Université de l'Illinois à Urbana-Champaign.
Plus d'informations : Qian Wang et al, Formation de triépoxyde par une monooxygénase dépendante de la flavine dans la biosynthèse du monensine, Nature Communications (2023). DOI :10.1038/s41467-023-41889-0
Fourni par l'Université du Texas à Austin