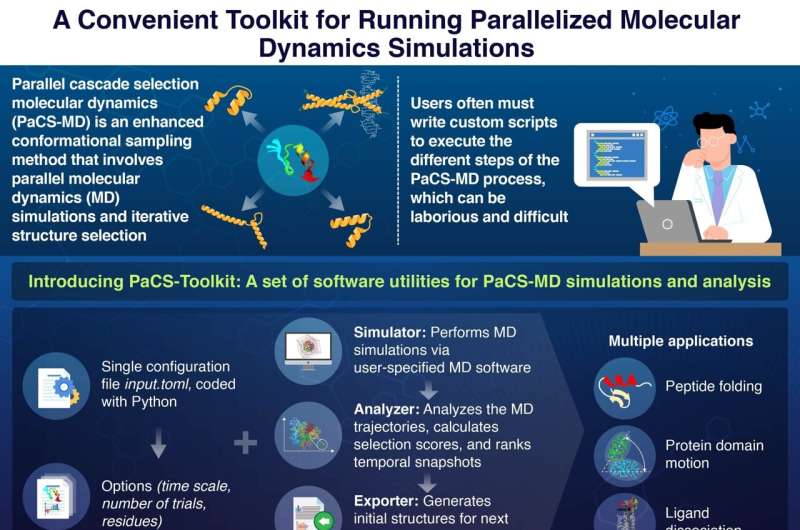
Les simulations de dynamique moléculaire (MD) sont devenues un outil puissant dans les domaines en constante croissance de la biologie moléculaire et du développement de médicaments. Bien qu'il existe de nombreuses techniques de simulation MD, la sélection en cascade parallèle MD (PaCS-MD) est particulièrement utile lors de l'étude du repliement (ou « conformation ») des protéines ou des interactions entre protéines et ligands.
Le nœud de cette méthode consiste à exécuter plusieurs simulations MD en parallèle, explorant ainsi simultanément différentes conformations possibles. Grâce à des critères de sélection soigneusement élaborés, les conformations prometteuses peuvent être détectées automatiquement dans des « instantanés temporels » et étudiées plus en détail. Cette stratégie accélère considérablement la découverte d'interactions moléculaires clés et de processus dynamiques qui aident les scientifiques à comprendre les mouvements fonctionnels des protéines.
Cependant, l'un des obstacles de PaCS-MD est que les utilisateurs doivent écrire des scripts personnalisés pour exécuter les simulations MD souhaitées. Dans ces scripts, ils doivent spécifier les conditions initiales et les caractéristiques cibles, sélectionner le logiciel MD à utiliser, mettre en œuvre une procédure de classement instantané et préparer les structures initiales pour le prochain cycle de simulation. Ce processus, qui peut être assez complexe et sujet aux erreurs, crée une barrière à l'entrée considérable pour les scientifiques intéressés par l'utilisation de PaCS-MD.
Heureusement, une équipe de chercheurs de l’École des sciences et technologies de la vie de l’Institut de technologie de Tokyo, au Japon, a récemment entrepris de résoudre ce problème. Dans leur dernière étude, publiée dans The Journal of Physical Chemistry B et dirigée par le professeur Akio Kitao, l'équipe a développé un progiciel appelé PaCS-Toolkit pour rendre PaCS-MD plus accessible et plus pratique à utiliser.
Un avantage notable de PaCS-Toolkit est que l'ensemble du processus de simulation est configuré via un seul fichier de configuration. Dans ce fichier, les utilisateurs spécifient des paramètres importants pour les simulations, notamment le type de PaCS-MD, le nombre de simulations MD à exécuter en parallèle et les résidus protéiques ou atomes à suivre comme critères de sélection pour les branches parallèles.
PaCS-Toolkit prend ce fichier de configuration, ainsi que les fichiers d'entrée MD standard, et exécute des simulations PaCS-MD selon le logiciel MD spécifié. Il convient de noter que puisque le package est un logiciel ouvert écrit en Python, un langage de programmation populaire, les utilisateurs peuvent contribuer à améliorer PaCS-Toolkit et à étendre ses fonctionnalités.
"Notre boîte à outils maintient la flexibilité afin que de nouvelles fonctionnalités, bibliothèques et logiciels MD puissent être ajoutés en introduisant des classes responsables en Python. Les utilisateurs capables de programmer dans ce langage devraient être capables de modifier le code de PaCS-Toolkit et d'implémenter de nouvelles méthodes si nécessaire, " dit le professeur Kitao.
Un autre avantage crucial de PaCS-Toolkit réside dans son optimisation et sa compatibilité avec différents environnements informatiques, qu'il s'agisse d'un ensemble de superordinateurs dotés d'une interface de transmission de messages (MPI), de serveurs équipés de plusieurs cartes graphiques (GPU) ou d'ordinateurs personnels tels que des ordinateurs portables.
"PaCS-Toolkit intègre fortement la parallélisation utilisant MPI, GPU et le package multitraitement de Python, permettant d'optimiser le temps de calcul en fonction des ressources de calcul disponibles", explique le professeur Kitao.
Pour présenter le potentiel de leur boîte à outils, les chercheurs ont mené des simulations PaCS-MD adaptées à trois applications différentes. Ceux-ci comprenaient le repliement de la mini-protéine chignoline, le mouvement des domaines protéiques dans une enzyme du SRAS-CoV-2 et la dissociation du ligand d'un important récepteur de l'adénosine.
"Pris ensemble, nos résultats indiquent que PaCS-Toolkit peut être facilement utilisé pour simuler une grande variété de dynamiques pour différents types de systèmes moléculaires", conclut le professeur Kitao.
Cette boîte à outils pourrait aider à libérer le véritable potentiel des simulations PaCS-MD dans divers domaines, permettant aux chercheurs intéressés de faire la lumière sur des processus moléculaires complexes et d'accélérer la découverte de médicaments.
Plus d'informations : Shinji Ikizawa et al, PaCS-Toolkit :utilitaires logiciels optimisés pour les simulations de dynamique moléculaire de sélection en cascade parallèle (PaCS-MD) et les analyses ultérieures, The Journal of Physical Chemistry B (2024). DOI :10.1021/acs.jpcb.4c01271
Informations sur le journal : Journal de Chimie Physique B
Fourni par l'Institut de technologie de Tokyo