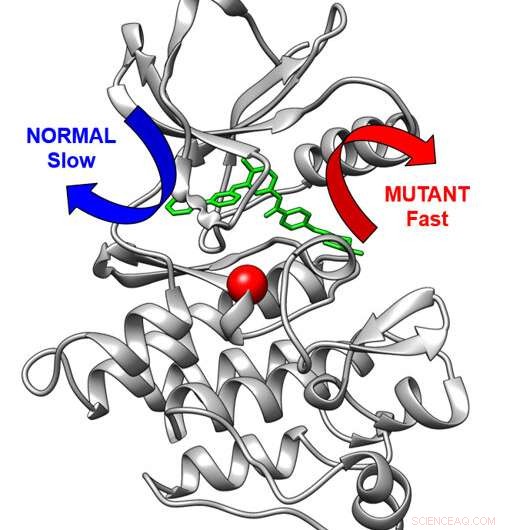
Structure tridimensionnelle de la cible médicamenteuse anticancéreuse Abl kinase (gris) liée à l'imatinib, un médicament anticancéreux. Normalement, le médicament sort en ralentissant via la flèche bleue. Une modification de la kinase (sphère rouge) fait sortir le médicament par une voie rapide (flèche rouge). Crédit :Aziz M. Rangwala
Comprendre pourquoi et comment la résistance à la chimiothérapie se produit est une étape majeure vers l'optimisation des traitements contre le cancer. Une équipe de scientifiques, dont Markus Seeliger, Ph.D., du Stony Brook Cancer Center et de la Renaissance School of Medicine de l'Université de Stony Brook, pense avoir découvert un nouveau processus par lequel la résistance aux médicaments se produit. Ils utilisent un modèle de simulation informatique qui les aide à comprendre exactement comment les molécules interagissent avec le médicament anticancéreux Imatinib (connu sous le nom de Gleevec) dans le processus de résistance à la chimiothérapie. L'imatinib traite la leucémie myéloïde chronique (LMC) de manière très efficace, mais de nombreux patients à un stade avancé présentent une résistance aux médicaments, ce qui rend le médicament peu efficace à ce stade.
La recherche est mise en évidence dans un article publié dans Angewandte Chemie et s'appuie sur des recherches antérieures détaillées en 2021 dans PNAS .
L'imatinib inhibe la protéine kinase BCR-Abl, une machinerie de signalisation cellulaire trop active dans la LMC. Dans le PNAS étude, les chercheurs ont montré que des variations dans le plan de construction de la kinase peuvent rendre plus difficile la liaison de l'imatinib à la kinase et également accélérer la libération du médicament par la kinase. Dans la Angewandte Chemie article, l'équipe de recherche a utilisé la méthodologie informatique, développée par le co-auteur Pratyush Tiwary de l'Université du Maryland, qui leur a permis d'étudier la libération très lente d'imatinib à partir de la kinase.
« Cette méthode en elle-même est une réalisation technique majeure qui étend les capacités de calcul pour la recherche sur la résistance aux médicaments et nous a surtout permis de prédire la rapidité avec laquelle des protéines saines et mutantes libéreraient ce médicament », déclare Seeliger, professeur agrégé au Département de pharmacologie. Les sciences. "Pour la première fois, nous avons pu voir la libération d'un médicament à partir d'une protéine avec autant de détails et de précision. De plus, nous avons pu montrer que la mutation change fondamentalement dans la voie de sortie du médicament de la protéine.
"Ceci est important car la vitesse de libération du médicament peut être tout aussi importante pour l'effet thérapeutique d'un médicament que la force avec laquelle un médicament se lie à la protéine."
Seeliger explique en outre que la méthode pourrait fournir une base pour comprendre les mécanismes moléculaires derrière la résistance à la chimiothérapie.
Plus largement, les implications de ce qu'ils ont découvert sont que si les scientifiques peuvent comprendre comment les médicaments sont libérés de leurs protéines, ils pourraient être en mesure de concevoir des médicaments avec une libération plus lente et un impact thérapeutique plus élevé. De plus, si la libération rapide du médicament peut entraîner une résistance aux médicaments et que les cliniciens peuvent montrer que cela se produit, ils peuvent réactiver l'efficacité du médicament en demandant au patient de prendre le médicament plus fréquemment.
Le travail de base pour le test de mutation via la méthode de calcul a été décrit dans le PNAS papier. Seeliger et ses collègues ont testé comment l'imatinib se lie aux mutations chez les patients atteints de LMC résistante à l'imatinib. Ils ont découvert que la majorité des mutations se lient facilement à l'imatinib, ce qui pose la question de savoir comment ces mutations provoquent une résistance chez les patients ? Les chercheurs ont ensuite identifié plusieurs mutants qui se lient facilement à l'imatinib mais libèrent le médicament beaucoup plus rapidement.
Après avoir identifié ces mutants avec une libération plus rapide du médicament, l'équipe a utilisé la résonance magnétique nucléaire (RMN) et la dynamique moléculaire pour lier la protéine à la dissociation du médicament, soulignant l'importance de la cinétique de dissociation du médicament pour l'efficacité du médicament. Cela leur a permis d'identifier un nouveau mécanisme de résistance à l'imatinib.
Le travail résultant de l'article publié dans PNAS a impliqué les efforts de collaboration de Seeliger et de ses collègues de Stony Brook, et des chercheurs du Memorial Sloan Kettering Cancer Center et de l'Université Goethe de Francfort, en Allemagne.
La recherche qui a abouti à l'article le plus récent a été menée par Tiwary et ses collègues de l'Université du Maryland, en collaboration avec Seeliger et des scientifiques du Broad Institute du MIT et de l'Université de Harvard. Une nouvelle simulation informatique perce le mystère de la résistance aux médicaments anticancéreux