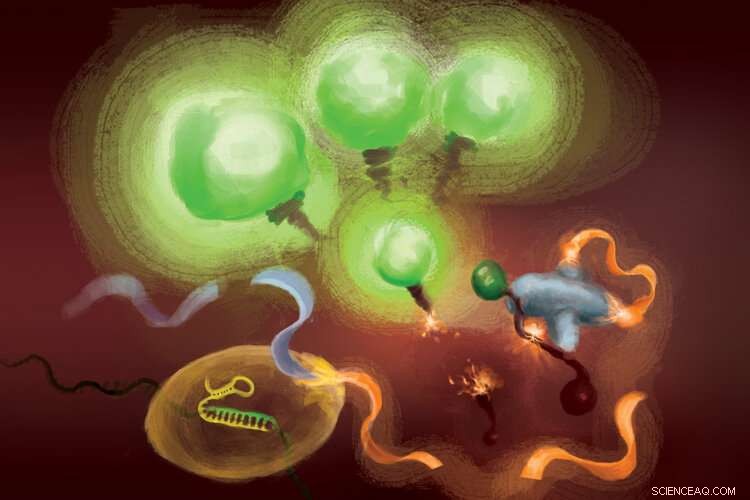
Les réactions impliquées dans le test FIND-IT pour détecter l'infection par le virus SARS-CoV-2. Lorsque l'enzyme Cas13 (en bas à gauche) se lie à son ARN cible, il coupe une molécule (ruban orange et bleu clair) pour libérer un activateur (orange) qui suralimente la nucléase Csm6 (à droite) pour cliver et libérer des molécules fluorescentes qui s'allument (vert) et signalent la présence d'ARN viral. Crédit :Margaret L. Liu, École de médecine Pritzker de l'Université de Chicago.
Fréquent, les tests rapides pour COVID-19 sont essentiels pour contrôler la propagation des épidémies, surtout comme neuf, des variantes plus transmissibles émergent.
Alors que le test de diagnostic COVID-19 de référence d'aujourd'hui, qui utilise qRT-PCR (réaction en chaîne de la transcriptase inverse-polymérase quantitative (PCR)) est extrêmement sensible, détecter jusqu'à une copie d'ARN par microlitre, il nécessite un équipement spécialisé, une autonomie de plusieurs heures et un laboratoire centralisé. Par conséquent, les tests prennent généralement au moins un à deux jours.
Une équipe de recherche dirigée par des scientifiques des laboratoires de Jennifer Doudna, David Savage et Patrick Hsu à l'Université de Californie, Berkeley, vise à développer un test de diagnostic beaucoup plus rapide et plus facile à déployer que la qRT-PCR. Il a maintenant combiné deux types différents d'enzymes CRISPR pour créer un test capable de détecter de petites quantités d'ARN viral en moins d'une heure. Doudna a partagé le prix Nobel de chimie 2020 pour l'invention de l'édition du génome CRISPR-Cas9.
Bien que la nouvelle technique n'en soit pas encore au stade où elle rivalise avec la sensibilité de la qRT-PCR, qui ne peut détecter que quelques copies du virus par microlitre de liquide, il est déjà capable de capter des niveaux d'ARN viral - environ 30 copies par microlitre - suffisants pour être utilisés pour surveiller la population et limiter la propagation des infections.
"Vous n'avez pas besoin de la sensibilité de la PCR pour détecter et diagnostiquer le COVID-19 dans la communauté, si le test est assez pratique et assez rapide, " a déclaré le co-auteur David Savage, professeur de biologie moléculaire et cellulaire. "Notre espoir était de pousser la biochimie le plus loin possible au point où vous pourriez imaginer un format très pratique dans un cadre où vous pouvez vous faire tester tous les jours, dire, à l'entrée du travail."
Les chercheurs publieront leurs résultats en ligne le 5 août dans la revue Nature Chimie Biologie .
Plusieurs tests basés sur CRISPR ont été autorisés pour une utilisation d'urgence par la Food and Drug Administration, mais tous nécessitent une étape initiale dans laquelle l'ARN viral est amplifié de sorte que le signal de détection, qui implique la libération d'une molécule fluorescente qui brille sous la lumière bleue, soit suffisamment brillant pour être vu. Bien que cette amplification initiale augmente la sensibilité du test à un niveau similaire à la qRT-PCR, il introduit également des étapes qui rendent le test plus difficile à réaliser en dehors d'un laboratoire.
L'équipe dirigée par l'UC Berkeley a cherché à atteindre une sensibilité et une vitesse utiles sans sacrifier la simplicité du test.
« Pour les applications au point de service, vous voulez avoir une réponse rapide pour que les gens sachent rapidement s'ils sont infectés ou non, avant de prendre l'avion, par exemple, ou aller rendre visite à des proches, " a déclaré la chef d'équipe Tina Liu, chercheur au laboratoire de Doudna à l'Institut de génomique innovante (IGI), un centre axé sur CRISPR impliquant des scientifiques de l'UC Berkeley et de l'UC San Francisco.
En plus d'avoir une étape supplémentaire, un autre inconvénient de l'amplification initiale est que, parce qu'il fait des milliards de copies d'ARN viral, il y a un plus grand risque de contamination croisée entre les échantillons de patients. La nouvelle technique développée par l'équipe inverse cela et amplifie à la place le signal fluorescent, éliminer une source majeure de contamination croisée.
La technique sans amplification, qu'ils appellent Fast Integrated Nuclease Detection In Tandem (FIND-IT), pourrait permettre des tests de diagnostic rapides et peu coûteux pour de nombreuses autres maladies infectieuses.
« Bien que nous ayons lancé ce projet dans le but exprès d’avoir un impact sur COVID-19, Je pense que cette technique particulière pourrait être applicable à plus que cette pandémie parce que, finalement, CRISPR est programmable, " dit Liu. " Alors, vous pourriez charger l'enzyme CRISPR avec une séquence ciblant le virus de la grippe ou le virus VIH ou tout type de virus à ARN, et le système a le potentiel de fonctionner de la même manière. Cet article établit vraiment que cette biochimie est un moyen plus simple de détecter l'ARN et a la capacité de détecter cet ARN dans un délai sensible et rapide qui pourrait se prêter à de futures applications dans les diagnostics sur le lieu de soins. »
Les chercheurs sont actuellement en train de construire un tel diagnostic à l'aide de FIND-IT, qui comprendrait des étapes pour collecter et traiter les échantillons et pour exécuter le test sur un dispositif microfluidique compact.
Utilisation de protéines Cas en tandem
Pour supprimer l'amplification cible de l'équation, l'équipe a utilisé une enzyme CRISPR - Cas13 - pour détecter d'abord l'ARN viral, et un autre type de protéine Cas, appelé Csm6, pour amplifier le signal de fluorescence.
Cas13 est un ciseau à usage général pour couper l'ARN; une fois qu'il se lie à sa séquence cible, spécifié par un ARN guide, il est amorcé pour couper une large gamme d'autres molécules d'ARN. Cette activité de coupe déclenchée par une cible peut être exploitée pour coupler la détection d'une séquence d'ARN spécifique à la libération d'une molécule reporter fluorescente. Cependant, tout seul, Cas13 peut nécessiter des heures pour générer un signal détectable lorsque de très faibles quantités d'ARN cible sont présentes.
L'idée de Liu était d'utiliser Csm6 pour amplifier l'effet de Cas13. Csm6 est une enzyme CRISPR qui détecte la présence de petits anneaux d'ARN et s'active pour couper une large gamme de molécules d'ARN dans les cellules.
Pour booster la détection de Cas13, elle et ses collègues ont conçu une molécule activatrice spécialement conçue qui est coupée lorsque Cas13 détecte l'ARN viral. Un fragment de cette molécule peut se lier à et déclencher le Csm6 pour couper et libérer une molécule fluorescente brillante à partir d'un morceau d'ARN. Normalement, la molécule activatrice est rapidement décomposée par le Csm6, limitant ainsi la quantité de signal fluorescent qu'il peut générer. Liu et ses collègues ont mis au point un moyen de modifier chimiquement l'activateur afin qu'il soit protégé de la dégradation et puisse surcharger le Csm6 pour couper et libérer à plusieurs reprises des molécules fluorescentes liées à l'ARN. Il en résulte une sensibilité 100 fois supérieure à celle de l'activateur d'origine.
"Lorsque Cas13 est activé, il clive ce petit activateur, supprimer un segment qui le protège, " dit Liu. " Maintenant qu'il est libéré, il peut activer de nombreuses molécules différentes de cette deuxième enzyme, Csm6. Et donc, une cible reconnue par Cas13 ne conduit pas seulement à l'activation de sa propre capacité de coupure d'ARN; cela conduit à la génération de beaucoup plus d'enzymes actives qui peuvent chacune cliver encore plus de reporters fluorescents."
L'équipe de chercheurs a également incorporé une combinaison optimisée d'ARN guides qui permet une reconnaissance plus sensible de l'ARN viral par Cas13. Lorsque cela a été combiné avec Csm6 et son activateur, l'équipe a pu détecter jusqu'à 31 copies par microlitre d'ARN du SRAS-CoV-2 en aussi peu que 20 minutes.
Les chercheurs ont également ajouté de l'ARN extrait d'échantillons de patients au test FIND-IT dans une cartouche microfluidique, pour voir si ce test pourrait être adapté pour fonctionner sur un appareil portable. À l'aide d'un petit appareil avec un appareil photo, ils pourraient détecter l'ARN du SRAS-CoV-2 extrait d'échantillons de patients à une sensibilité qui capturerait les infections COVID-19 à leur apogée.
"Cette approche de nucléase en tandem—Cas13 plus Csm6—combine tout en une seule réaction à une seule température, 37 degrés Celsius, il ne nécessite donc pas de chauffage à haute température ou plusieurs étapes, comme cela est nécessaire pour d'autres techniques de diagnostic, " a déclaré Liu. " Je pense que cela ouvre des opportunités pour plus rapidement, des tests plus simples qui peuvent atteindre une sensibilité comparable à d'autres techniques actuelles et pourraient potentiellement atteindre des sensibilités encore plus élevées à l'avenir."
Le développement de cette méthode sans amplification pour la détection de l'ARN est le résultat d'une réorientation de la recherche au sein de l'IGI lorsque la pandémie a commencé vers des problèmes de diagnostic et de traitement du COVID-19. Finalement, cinq laboratoires de l'UC Berkeley et deux laboratoires de l'UCSF se sont impliqués dans ce projet de recherche, l'un des nombreux au sein de l'IGI.
"Quand nous avons commencé cela, nous avions l'espoir de créer quelque chose qui atteindrait la parité avec la PCR, mais ne nécessitait pas d'amplification - ce serait le rêve, " dit Sauvage, qui était le chercheur principal du projet. "Et du point de vue de la sensibilité, nous avions un écart d'environ dix mille fois à sauter. Nous l'avons fait environ mille fois; nous l'avons fait baisser d'environ trois ordres de grandeur. Donc, nous y sommes presque. Avril dernier, quand nous commencions vraiment à le cartographier, cela semblait presque impossible."