Des chercheurs utilisent des outils linguistiques d'intelligence artificielle pour décoder les mouvements moléculaires
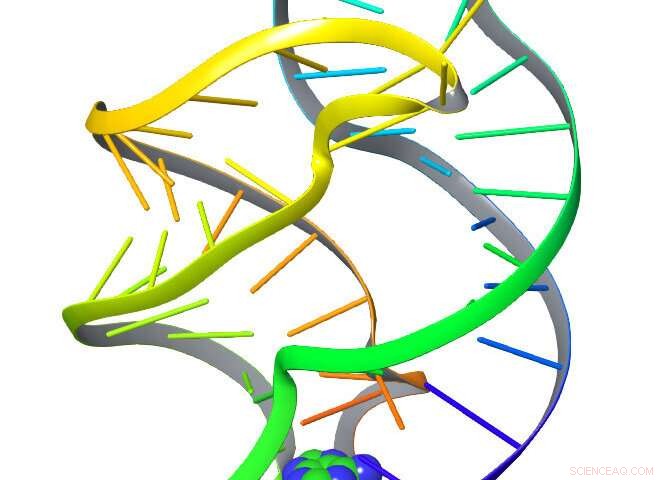
Des scientifiques de l'Université du Maryland ont appliqué un système de traitement du langage aux mouvements d'une molécule riboswitch illustrée ici, comprendre comment et quand la molécule prend différentes formes. Crédit :Zachary Smith/UMD
En appliquant des outils de traitement du langage naturel aux mouvements des molécules de protéines, Des scientifiques de l'Université du Maryland ont créé un langage abstrait qui décrit les multiples formes qu'une molécule de protéine peut prendre et comment et quand elle passe d'une forme à une autre.
La fonction d'une molécule de protéine est souvent déterminée par sa forme et sa structure, ainsi, comprendre la dynamique qui contrôle la forme et la structure peut ouvrir la porte à tout comprendre, du fonctionnement d'une protéine aux causes de la maladie et à la meilleure façon de concevoir des thérapies médicamenteuses ciblées. C'est la première fois qu'un algorithme d'apprentissage automatique est appliqué à la dynamique biomoléculaire de cette manière, et le succès de la méthode fournit des informations qui peuvent également aider à faire progresser l'intelligence artificielle (IA). Un document de recherche sur ce travail a été publié le 9 octobre. 2020, dans la revue Communication Nature .
"Ici, nous montrons les mêmes architectures d'IA utilisées pour compléter des phrases lors de la rédaction d'e-mails peuvent être utilisées pour découvrir une langue parlée par les molécules de la vie, " a déclaré l'auteur principal de l'article, Pratyush Tiwary, professeur adjoint au Département de chimie et de biochimie de l'UMD et à l'Institut des sciences physiques et technologiques. "Nous montrons que le mouvement de ces molécules peut être cartographié dans un langage abstrait, et que les techniques d'IA peuvent être utilisées pour générer des histoires biologiquement véridiques à partir des mots abstraits qui en résultent."
Les molécules biologiques sont constamment en mouvement, se trémousser dans leur environnement. Leur forme est déterminée par la façon dont ils sont pliés et tordus. Ils peuvent rester dans une forme donnée pendant des secondes ou des jours avant de s'ouvrir soudainement et de se replier dans une forme ou une structure différente. La transition d'une forme à une autre se produit un peu comme l'étirement d'une bobine enchevêtrée qui s'ouvre par étapes. Au fur et à mesure que différentes parties de la bobine se libèrent et se déplient, la molécule prend différentes conformations intermédiaires.
Mais le passage d'une forme à une autre se fait en picosecondes (millièmes de seconde) ou plus vite, ce qui rend difficile pour les méthodes expérimentales telles que les microscopes à haute puissance et la spectroscopie de capturer exactement comment le déroulement se produit, quels paramètres affectent le déroulement et quelles différentes formes sont possibles. Les réponses à ces questions forment l'histoire biologique que la nouvelle méthode de Tiwary peut révéler.
Tiwary et son équipe ont appliqué les lois du mouvement de Newton - qui peuvent prédire le mouvement des atomes dans une molécule - avec de puissants superordinateurs, y compris Deepthought2 d'UMD, développer des modèles de physique statistique qui simulent la forme, mouvement et trajectoire de molécules individuelles.
