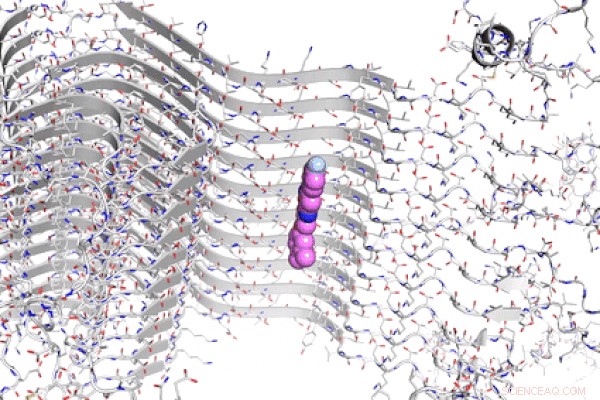
La structure chimique d'une fibrille d'alpha-synucléine avec une molécule « exemplaire », représenté par des sphères colorées, lié à un site de liaison préalablement identifié. Des chercheurs de Penn ont récemment démontré une nouvelle méthode de criblage et d'identification informatique de molécules qui peuvent ensuite être développées en sondes d'imagerie pour étudier les protéines associées à la maladie de Parkinson. Crédit :E. James Petersson
Pour la plupart des 200, 000 patients diagnostiqués avec la maladie de Parkinson aux États-Unis chaque année, le diagnostic n'intervient souvent qu'après l'apparition de symptômes sévères tels que des tremblements ou des difficultés d'élocution. Dans le but de reconnaître et de traiter plus tôt les maladies neurologiques, les chercheurs recherchent de nouvelles façons d'imager des molécules biologiques qui indiquent la progression de la maladie avant l'apparition des symptômes. L'un de ces candidats, et une caractéristique connue de la maladie de Parkinson, est la formation d'amas de protéine alpha-synucléine, et, alors que cette protéine a été identifiée il y a plus de 20 ans, un moyen fiable de suivre les agrégats d'alpha-synucléine dans le cerveau n'a pas encore été développé.
Maintenant, une nouvelle étude publiée dans Sciences chimiques décrit une approche innovante pour identifier des molécules qui peuvent aider à suivre la progression de la maladie de Parkinson. Mené par des chercheurs dans les laboratoires d'E. James Petersson, Robert Mach, et Virginie Lee, cette étude de validation de principe pourrait changer le paradigme de la façon dont les chercheurs sélectionnent et testent de nouvelles molécules pour étudier un large éventail de maladies neurodégénératives.
L'étude de ces types d'agrégats de protéines nécessite de nouveaux traceurs, les molécules radioactives que les cliniciens utilisent pour imager les tissus et les organes, pour la tomographie par émission de positons (TEP). En tant que chercheur senior dans le domaine du développement des traceurs PET, Mach et son groupe ont travaillé pendant plusieurs années avec la Fondation Michael J. Fox pour développer un traceur d'alpha-synucléine, mais sans données sur la structure de la protéine, ils n'ont pas pu trouver de candidats suffisamment sélectifs pour être utilisés comme outil de diagnostic.
Puis, avec la première publication de la structure de l'alpha-synucléine et une augmentation des outils disponibles dans le domaine de la chimie computationnelle, Mach et Petersson ont commencé à collaborer au développement d'un traceur TEP alpha-synucléine. En conjuguant leurs expertises respectives en radiochimie et en ingénierie des protéines, ils ont pu confirmer expérimentalement où sur le potentiel de la protéine alpha-synucléine les molécules traceuses étaient capables de se lier, des informations cruciales pour les aider à découvrir et concevoir des molécules qui seraient spécifiques à l'alpha-synucléine.
Dans leur dernière étude, les chercheurs ont développé une méthode de calcul à haut débit, leur permettant de cribler des millions de molécules candidates, pour voir lesquels se lieront aux sites de liaison connus sur l'alpha-synucléine. En s'appuyant sur une méthode précédemment publiée, leur approche identifie d'abord un « exemple, " une pseudo-molécule qui s'intègre parfaitement dans le site de liaison de l'alpha-synucléine. Ensuite, cet exemple est comparé à des molécules réelles disponibles dans le commerce pour voir lesquelles ont une structure similaire. Les chercheurs utilisent ensuite d'autres programmes informatiques pour affiner la liste des candidats aux tests en laboratoire.
Pour évaluer la performance de leur méthode de dépistage, les scientifiques ont identifié un petit sous-ensemble de 20 candidats prometteurs parmi les 7 millions de composés sélectionnés et ont découvert que deux avaient une affinité de liaison extrêmement élevée avec l'alpha-synucléine. Les chercheurs ont également utilisé des tissus cérébraux de souris fournis par le groupe Lee pour valider davantage cette nouvelle méthode. Les chercheurs ont été impressionnés, et agréablement surpris, par leur taux de réussite, qu'ils attribuent à la spécificité de leur méthode de recherche. "Il y a certainement un peu de chance aussi, " Petersson ajoute, "La plus grande surprise est probablement à quel point cela a bien fonctionné."
L'idée d'utiliser la méthode exemplaire pour résoudre ce problème est venue au premier auteur et au doctorat. diplômé John "Jack" Ferrie alors qu'il apprenait les méthodes de chimie computationnelle à l'Institute for Protein Design de l'Université de Washington dans le cadre d'une bourse d'été de la Fondation Parkinson. "La bourse d'été est conçue pour former les étudiants à de nouvelles méthodes pouvant être appliquées à la recherche sur la maladie de Parkinson, et c'est exactement ce qui s'est passé ici, " dit Petersson. " Les idées avec lesquelles Jack est revenu ont formé la base d'un gros effort à la fois dans mon laboratoire et dans celui de Bob Mach pour identifier les traceurs TEP par ordinateur. "
Maintenant, dans le cadre d'une importante subvention multi-institutionnelle, Petersson, Mach, Lee, et de nombreux autres collaborateurs sont prêts à tirer les leçons de cette découverte pour développer des traceurs TEP pour la maladie de Parkinson et d'autres maladies neurodégénératives. « Je vois vraiment cela comme un changement dans la façon dont nous développons les sondes PET, " dit Mach. " L'importance est que nous sommes en mesure de cribler des millions de composés dans un très court laps de temps, et nous sommes en mesure d'identifier un grand nombre de composés qui se lieront probablement avec une affinité élevée à l'alpha-synucléine. Nous allons également appliquer cette même méthode au développement d'autres sondes importantes mais qui ont présenté des défis sur le terrain."
En développant des outils fiables à haut débit qui utilisent une connaissance détaillée de la structure des protéines, l'objectif des efforts futurs est de trouver de nouveaux candidats traceurs et de les amener à la clinique dès qu'ils sont prêts à être testés. "C'est certainement accéléré par rapport à ce qui est typique, " dit Petersson au sujet des délais de la subvention. " Cela peut prendre 10 à 15 ans dans l'industrie, et nous essayons de le faire dans environ cinq. »
Mach ajoute que cet effort est un parfait exemple de "comment les choses fonctionnent ici à Penn, " avec un succès rendu possible par des collaborations entre des chercheurs aux compétences diverses et uniques. " Penn est un endroit formidable parce que vous avez beaucoup de gens talentueux qui ont un véritable esprit de collaboration, et c'est ce qu'il faut pour faire de la science de nos jours, " il dit.