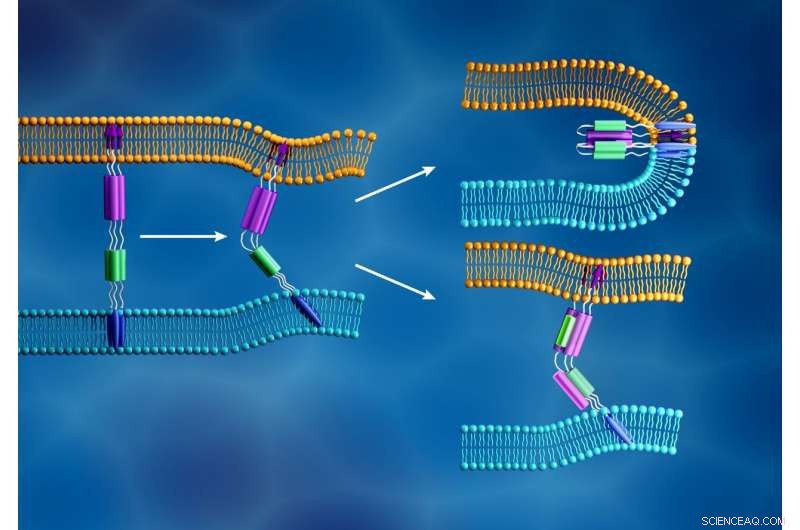
La nouvelle membrane du coronavirus (bleu clair) et la membrane cellulaire humaine (orange) fusionnent lorsque le peptide de fusion de la sous-unité virale S2 (flèches violettes) s'insère dans la membrane cellulaire et qu'un composant différent de la sous-unité S2 (violet et vert) se replie pour former une structure serrée, comme indiqué en haut à droite. En revanche, comme illustré en bas à droite, les inhibiteurs de fusion sont conçus pour prévenir l'infection virale en perturbant ce processus. Crédit :ORNL/Jill Hemman
SRAS-CoV-2, le coronavirus responsable de la maladie COVID-19, infecte le monde à un rythme rapide. Comprendre comment cette infection fonctionne au niveau moléculaire pourrait aider les experts à découvrir des moyens de modérer ou d'arrêter la propagation.
Une équipe de scientifiques du laboratoire national d'Oak Ridge (ORNL) du ministère de l'Énergie (DOE) utilise la réflectométrie neutronique pour faire exactement cela.
Les neutrons sont capables de sonder des matériaux biologiques dans des conditions physiologiques sans les endommager. En exploitant ces propriétés, les chercheurs peuvent mesurer la dynamique de l'infection du virus au fur et à mesure qu'il se produit.
Leur mission est d'obtenir un aperçu détaillé de certaines des premières étapes de l'infection se produisant au niveau de la membrane cellulaire. Ces résultats aideront l'équipe à tester des candidats médicaments antiviraux qui pourraient perturber ce processus. Les données obtenues à partir de ces expériences pourraient également éclairer d'autres études axées sur le développement de thérapies et de vaccins.
Les chercheurs concentrent leur analyse sur les protéines de pointe du SRAS-CoV-2, des protéines structurelles de type barbe qui recouvrent la surface du virus et déclenchent le processus d'infection. La protéine de pointe se lie à un récepteur sur la couche externe de la cellule hôte et facilite la fusion entre les membranes virales et cellulaires, permettant au virus d'entrer dans la cellule et de libérer son matériel génétique. La machinerie de fabrication de protéines de la cellule utilise ensuite cette information génétique pour faire de nouvelles copies du virus.
Lorsque le SARS-CoV-2 détourne une cellule hôte, sa protéine de pointe se divise en deux sous-unités, appelés S1 et S2. Les deux parties sont toutes deux essentielles à l'infection. La sous-unité S1 contient un domaine de liaison au récepteur qui reconnaît et s'accroche à un récepteur cellulaire. Les récepteurs cellulaires sont des protéines intégrées dans la membrane cellulaire qui peuvent se lier à des molécules spécifiques à l'extérieur de la cellule. Cette connexion peut entraîner un changement de forme des composants, ce qui à son tour pourrait induire des changements en cascade dans la cellule. Pour la protéine de pointe SARS-CoV-2, cette connexion active le sous-ensemble S2, qui aide le virus à fusionner sa membrane avec celle de la cellule. Par conséquent, la fonction de la protéine de pointe est similaire à l'ouverture d'une porte verrouillée, avec S1 comme clé qui déverrouille la porte et S2 comme force qui pousse la porte à s'ouvrir.
Apprendre des épidémies passées
La structure globale de la protéine de pointe SARS-CoV-2 est très similaire à celle du SARS-CoV, un ancien coronavirus qui a causé le syndrome respiratoire aigu sévère (SRAS), et cette ressemblance a aidé l'équipe à développer leur stratégie de recherche.
La sous-unité S1 est au centre de nombreuses études de développement de médicaments, car il a été démontré que cette partie de la protéine de pointe provoque une réponse immunitaire dans le corps humain. Cependant, des études précédentes sur le SRAS-CoV ont révélé que la sous-unité S1 présentait des taux de mutation élevés, permettant au virus d'échapper aux traitements à base d'anticorps tout en maintenant sa capacité à infecter les cellules. "C'est la leçon que nous avons tirée de l'épidémie initiale de SRAS, " dit Minh Phan, associé de recherche postdoctoral à l'ORNL et chercheur principal de ce projet.
Phan et ses collègues étudient la sous-unité S2 car ce composant de la protéine de pointe ne mute pas aussi rapidement. Les traitements qui réussissent à inhiber la fonction S2 peuvent rester efficaces plus longtemps.
Une vue nanométrique du coronavirus
Pour mieux comprendre la dynamique des sous-unités S2 virales et des membranes cellulaires hôtes, les chercheurs utilisent le réflectomètre pour liquides (LIQREF) à la source de neutrons de spallation (SNS) de l'ORNL. En mesurant la réflexion des neutrons sous différents angles lorsqu'ils traversent différents types de matière, l'instrument peut aider à faire la lumière sur la structure des matériaux biologiques à l'échelle moléculaire.
L'équipe a d'abord synthétisé une membrane lipidique qui imite la membrane externe des cellules qui tapissent les surfaces à l'intérieur des poumons humains, où cette infection virale peut avoir lieu. Ils ont identifié comment les lipides étaient organisés au sein de la membrane et comment cet arrangement change lorsque les membranes sont exposées à différentes conditions, comme la température, pression, et l'acidité.
A l'instrument LIQREF, les chercheurs ont étendu la membrane lipidique au-dessus d'une fine couche d'eau dans un appareil appelé auge de Langmuir. Ils introduisent ensuite la sous-unité S2 dans ces membranes pour observer en détail comment les membranes S2 et lipidiques changent de forme lorsqu'elles interagissent.
Les neutrons sont également idéaux pour cette étude car ils sont sensibles à l'élément hydrogène, commun à toutes les molécules biologiques, et ses isotopes. En remplaçant certains atomes d'hydrogène par des atomes de deutérium, les scientifiques peuvent créer un contraste dans leurs échantillons et cibler sélectivement différentes caractéristiques structurelles. Cette technique est utile pour étudier des échantillons qui impliquent plusieurs composants avec des densités similaires, comme les membranes lipidiques.
"Généralement, ces membranes ne sont pas des membranes monolipidiques, " said John Ankner, an instrument scientist involved with this study. "They consist of lipids of a certain structure, lipids of another structure, cholestérol, protéines, and things that come in contact with them."
To capture this complexity, the research team is investigating multiple versions of the membrane, changing the contrast of the sample with deuterium each time.

Researchers at ORNL are using neutron scattering at the Spallation Neutron Source to better understand how spike proteins help the COVID-19 virus infect human cells and what drugs could be effective in stopping them. This research team includes John Ankner (left) and Minh Phan (right). Crédit :ORNL/Geneviève Martin
"By taking multiple measurements and assembling all of this information together, you can create a single picture of how these different components go together, " said Ankner.
The information derived from these experiments will then help steer the team's efforts in selecting and testing drug candidates that could block this interaction, such as fusion inhibitors that successfully blocked original SARS-CoV infections. If these inhibitors can stop the new coronavirus from invading healthy cells, existing drugs could potentially be repurposed to treat COVID-19 patients. The results may also help guide the design of new fusion inhibitors.
Capturing infection
While other studies have used protein crystallography to better understand the atomic structure of the coronavirus S2 subunit alone, this project is analyzing how S2 changes shape when interacting with a lipid membrane. A shape change could be important for inducing actions within a cell after the spike S1 subunit binds to the cell receptor. Phan also notes that the LIQREF instrument allows the team to measure these dynamics under physiological conditions, whereas protein crystallography only allows researchers to capture what the S2 subunit looks like in a crystallized form.
"At ORNL, we have the right tools to study the dynamics of the interaction under physiological conditions. This allows us to better understand how the S2 subunit moves and changes shape naturally in a wet environment, " said Phan. "Such information could complement what experts already know about the protein from crystallography. If we can help verify what this mechanism looks like, then we may have a clearer understanding to guide the development of drugs that block the fusion process.
Collaboration is key
Bien sûr, learning more about the S2 subunit and its certain behaviors depends on the ability to grow quality samples, which involves synthesizing S2 subunit proteins, purifying them, and preparing them for experimentation.
Phan and Ankner note that this part of their research has been made possible only through collaboration with labs across ORNL and at outside institutions.
The S2 subunit protein was synthesized in mammalian cell cultures by Steve Foster, a biomedical researcher at the University of Tennessee Medical Center in Knoxville, Tennessee. Through this method, he can develop S2 proteins for research that retain several aspects of its natural structure and function.
"In our lab we routinely use mammalian cell cultures for protein production, so we hope we've produced an S2 protein best suited for this research analysis. Our proximity to ORNL also works well in that the sample doesn't have to travel far, meaning less risk of damaging the protein or distorting its original structure, which is critical for this work, " said Foster.
Following its synthesis, the sample was purified by Jessy Labbé and Michael Melesse Vergara from ORNL's Biosciences Division. Scientists from the ORNL Neutron Sciences Directorate then performed a series of tests to confirm the structure of the sample protein and check its purity. This effort was implemented by Yichong Fan and Wellington Leite from the Bio-Labs team, and Jacob Kinnun and Mary Odom from the SNS team.
"We put an enormous effort into making sure the protein has the right properties going into the experiment. If it does not, we could get spurious results and misinterpret what we're doing, " said Hugh O'Neill, director of ORNL's Center for Structural and Molecular Biology and lead researcher for the Bio-Labs team.
"This virus is extremely delicate in its components, and it's a big challenge to get these materials to the neutron instrument, " said Ankner. "That's why involving various ORNL labs and the University of Tennessee is so crucial. Each step that eventually gets the sample onto our instrument requires the expertise of lots of people."
This project also relied on efforts from the LIQREF instrument staff, who were instrumental in developing the systems, protocols, and modeling frameworks necessary to run the experiments and interpret the data.
"Experts across the division, across ORNL, and from partner institutes have come together for this project, " said Phan. "We couldn't have done this without their support, and it's greater motivation to fulfill our mission."