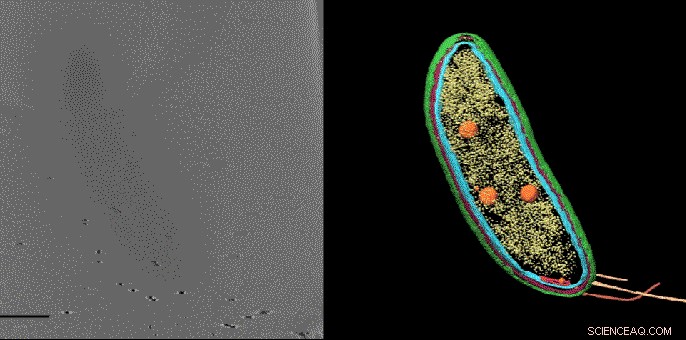
Les scientifiques de l'Université de Stanford pensaient qu'une zone apparemment vide à une extrémité de la bactérie Caulobacter pourrait contenir deux protéines impliquées dans la division cellulaire. En marquant les protéines avec des marqueurs fluorescents, puis en imageant ces mêmes échantillons avec une tomographie cryoélectronique, ils ont pu confirmer cet emplacement et montrer exactement comment les protéines étaient disposées. Crédit :Peter Dahlberg et al., PNAS , 8 juin 2020
Les scientifiques ont marié deux des techniques de microscopie les plus puissantes d'aujourd'hui pour faire des images qui identifient, pour la première fois, les identités et les emplacements précis des protéines individuelles dans le contexte détaillé des cellules bactériennes. Cette information est cruciale pour apprendre comment les molécules de protéines travaillent ensemble pour organiser la division cellulaire et effectuer d'autres tâches importantes, comme permettre aux microbes de flairer la nourriture et le danger.
La nouvelle méthode a déjà mis au jour de nouvelles informations sur les protéines bactériennes et leurs voisinages cellulaires proches. Les chercheurs disent qu'il a également le potentiel de répondre à des questions fondamentales sur la machinerie moléculaire des virus, parasites, et des processus comme la photosynthèse.
"C'est un grand pas pour la biologie, et je pense qu'il y en a beaucoup, de nombreux systèmes qui bénéficieront de ce type d'imagerie, " a déclaré le professeur de Stanford Lucy Shapiro, dont le groupe de recherche a participé à l'étude.
La nouvelle méthode hybride, appelée imagerie corrélée par annotation avec des molécules uniques, ou CIASM (prononcé « gouffre »), a été développé par Peter Dahlberg, chercheur postdoctoral dans le laboratoire du professeur W. E. Moerner à l'Université de Stanford.
C'est une variante d'une technique appelée microscopie monomoléculaire à basse température, inventé par Moerner il y a trois décennies, qui attache des étiquettes lumineuses aux molécules afin qu'elles puissent être identifiées individuellement. Cette méthode sous-tend la microscopie à fluorescence à super-résolution, le sujet du prix Nobel de chimie de Moerner en 2014.
Dahlberg a trouvé un moyen de faire fonctionner ce type d'imagerie par fluorescence à des températures inférieures au point de congélation afin que les mêmes échantillons puissent également être examinés par tomographie électronique cryogénique (CET). Le CET utilise des flux d'électrons pour créer des images 3D de cellules congelées par flash et de leurs composants à une résolution proche de l'atome. La combinaison du CET avec l'imagerie fluorescente permet aux scientifiques de voir les molécules marquées dans le contexte de la cellule environnante, une perspective cruciale pour comprendre leur rôle dans la machinerie cellulaire.
"Nous pouvons étiqueter des molécules d'intérêt spécifiques afin que la lumière que nous voyons ne provienne que de ces molécules, et puis nous trouvons où ils sont à environ 10 nanomètres, ou des milliardièmes de mètre. Cela nous donne une image beaucoup plus précise de ce qui se passe, " a déclaré Dahlberg. "Nous avons pris les instantanés ultra-précis fournis par CET et ajouté un peu de couleur."
Il ajouta, « C'est passionnant de développer de nouvelles méthodes d'imagerie. Lorsque vous avez terminé, vous pouvez prendre du recul et examiner toutes les nouvelles questions que vous pouvez attaquer."
Avec le CIASM, l'équipe de recherche a pu localiser les emplacements de trois types de protéines dans des images CET haute résolution de bactéries prises au laboratoire d'accélérateur national SLAC du ministère de l'Énergie. Les résultats ont été rapportés dans le Actes de l'Académie nationale des sciences aujourd'hui.
"Chaque méthode a ses avantages et ses inconvénients, " Moerner a dit, "Et c'est une belle situation où nous pouvons combiner deux méthodes pour en savoir plus."
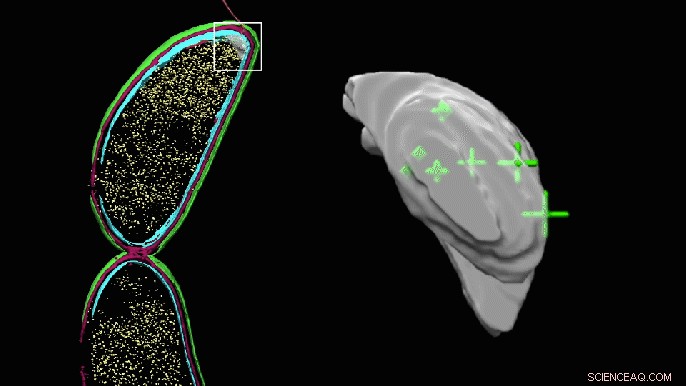
Une image 3D en rotation de la poche apparemment vide à une extrémité d'une cellule Caulobacter montre maintenant les emplacements précis des molécules PopZ. La poche semble grumeleuse car elle a été colorée manuellement pour mettre en évidence la zone où les chercheurs pensaient que les molécules pourraient se trouver, mais n'a pas pu identifier directement en tomographie cryoélectronique. Crédit :Peter Dahlberg et al., PNAS , 8 juin 2020
Trouver de l'ordre dans une soupe cellulaire
Même dans des cellules bactériennes relativement simples, l'emplacement est tout, dit Saumya Saurabh, un chercheur postdoctoral dans le laboratoire de Shapiro qui a joué un rôle de premier plan dans la recherche.
"Les gens ont tendance à considérer les bactéries comme des sacs de protéines sans organisation, " dit-il. " Mais il s'avère que ce n'est pas vrai, et en fait, de nombreuses molécules des bactéries sont précisément situées à la fois dans l'espace et dans le temps. S'ils ne sont pas dans la bonne position, la cellule meurt. Ce que le travail de Pete nous permet enfin de faire, c'est de regarder à l'intérieur avec une résolution moléculaire et de découvrir quand et où ces molécules sont situées les unes par rapport aux autres."
Caulobacter crescentus, par exemple, une espèce bien étudiée de bactéries d'eau douce, est connu pour se diviser en deux types de cellules filles très différents :l'une nage librement, tandis que l'autre forme une tige et s'attache à une surface. Comment chaque cellule fille obtient ce dont elle a besoin pour suivre son chemin unique est un mystère de longue date.
Les scientifiques avaient précédemment identifié de petites zones à chaque extrémité de la cellule en division qui pourraient contenir des protéines jouant un rôle clé dans cette division cellulaire déséquilibrée. L'une des protéines, PopZ, se trouve aux deux extrémités de la cellule de division, tandis que l'autre, SpmX ("Spam-X") ne se trouve que dans la moitié qui développera une tige.
Pour cette étude, Saurabh et l'étudiant diplômé Jiarui Wang ont marqué des protéines chez Caulobacter avec des marqueurs fluorescents. Puis Dahlberg a gelé ces échantillons, réalisé une imagerie de fluorescence à molécule unique sur eux avec l'aide de l'étudiante diplômée Annina Sartor, et les a emmenés aux installations Cryo-EM de Stanford-SLAC pour l'imagerie CET dirigée par Wah Chiu, professeur à Stanford et au SLAC.
Cartographier un hangout protéiné
Les images combinées ont non seulement confirmé que les deux protéines se trouvaient dans les zones suspectées par les scientifiques, mais a également révélé exactement comment ils étaient disposés :SpmX était incrusté dans la membrane interne de la cellule et faisait saillie à l'intérieur de la cellule, où il est entré en contact direct avec PopZ.
"L'orientation exacte de ce complexe protéique a été débattue au cours des 12 dernières années, " Saurabh a déclaré. "Nous avons pu observer les partenaires protéiques avec une résolution exquise. Nous avons maintenant une image très précise de la façon dont ces protéines communiquent entre elles dans la cellule."
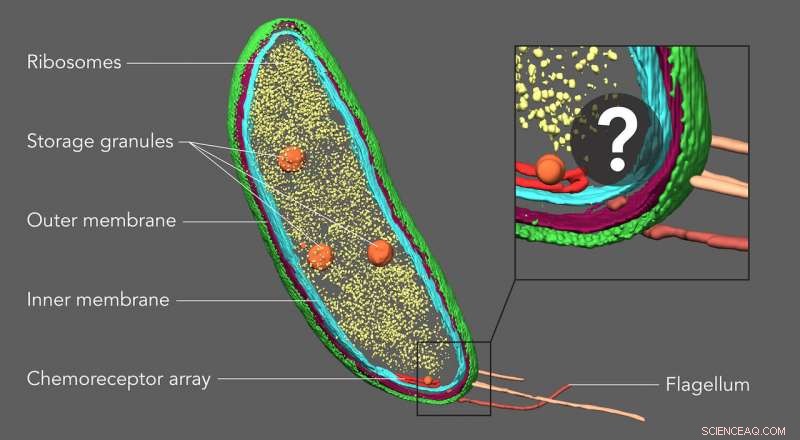
Avec une technique appelée tomographie cryoélectronique, les scientifiques peuvent créer des images 3D détaillées des cellules, comme cette bactérie Caulobacter, et mettre en évidence leurs composants - dans ce cas, les membranes cellulaires (rouge et bleu), coque protéinée (verte), usines de protéines appelées ribosomes (jaune) et granules de stockage (orange). Mais jusqu'à maintenant, des structures plus petites et des molécules individuelles n'ont pas pu être identifiées et localisées avec précision dans ces images. Une nouvelle technique d'imagerie développée à Stanford comble cette lacune, révélant de petites molécules qui ne sont pas visibles ici. Crédit :Peter Dahlberg et al., PNAS , 8 juin 2020
L'équipe a testé l'exactitude du CIASM en l'utilisant pour confirmer l'emplacement d'une protéine appelée McpA qui était connue pour faire partie d'un réseau de chimiorécepteurs dans la bactérie. "Les protéines extrêmement sensibles de cette matrice servent de nez à Caulobacter, " Saurabh a dit, "détecter la chimie de l'environnement environnant afin qu'ils puissent s'éloigner des choses désagréables et se diriger vers le glucose qu'ils mangent."
Le réseau apparaît sous forme de lignes noires parallèles dans les images CET, et le marquage fluorescent des mêmes images a localisé les emplacements des protéines McpA individuelles dans un rayon d'environ 10 nanomètres.
Un aperçu détaillé des points quantiques
Dans un séparé, étude parallèle, publié le 24 avril dans Angewandte Chemie , les chercheurs ont utilisé une technique similaire pour examiner des points quantiques uniques, avec des résultats surprenants.
Les points quantiques sont des cristaux nanométriques de matériau semi-conducteur qui émettent naturellement une fluorescence dans des couleurs déterminées par leur taille, forme et composition. Ces points sont utilisés dans la recherche pour marquer et suivre les protéines et autres matériaux biologiques, et ont des applications potentielles dans l'électronique future, éclairage, l'informatique quantique, l'imagerie médicale et d'autres domaines.
Dans cette étude, l'objectif était de voir comment les détails structurels les plus fins des points individuels étaient liés à des détails spécifiques de leurs propriétés optiques, dit Davis Perez, un doctorat étudiant dans le laboratoire de Moerner.
« Nous avons pu observer des comportements surprenants des points quantiques individuels, par exemple, dans leur réponse à l'excitation avec la lumière laser, " dit-il. " Mais l'aspect le plus excitant pour moi est que la méthode que nous avons développée pour étudier les points quantiques peut également être utilisée pour étudier des systèmes biologiques tels que les protéines photosynthétiques, où l'énergie est transférée entre des groupes de protéines, et voyez comment fonctionne la machinerie photosynthétique."
Moerner a déclaré que son laboratoire travaillait avec Chiu pour relever ces défis.
"C'est le début de la combinaison des deux méthodes, et nous sommes ravis d'explorer d'autres collaborations liant la lumière et les électrons, " a déclaré Chiu. " Cette approche d'imagerie hybride a le potentiel de découvrir des structures de composants moléculaires impliqués dans des processus biologiques clés dans des cellules couvrant tous les domaines de la vie. "