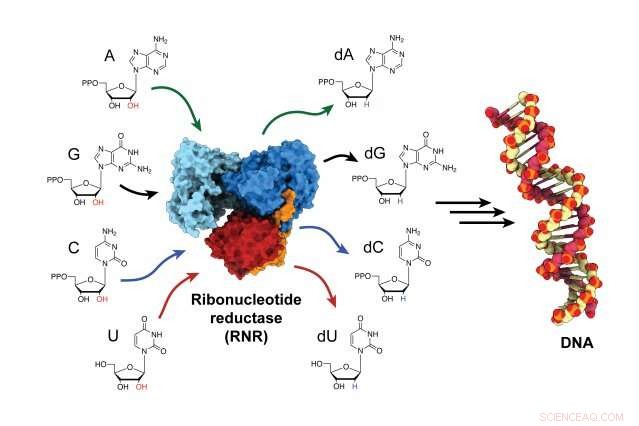
L'enzyme ribonucléotide réductase (RNR) est responsable de la conversion des éléments constitutifs de l'ARN en éléments constitutifs de l'ADN, et est un acteur essentiel à la fois dans la synthèse et la réparation de l'ADN dans tous les organismes. Crédit :Gyunghoon "Kenny" Kang
Comment capturer un processus cellulaire qui se déroule en un clin d'œil ? Les biochimistes du MIT ont mis au point un moyen de piéger et de visualiser une enzyme vitale au moment où elle devient active, informant ainsi le développement de médicaments et révélant comment les systèmes biologiques stockent et transfèrent l'énergie.
L'enzyme, la ribonucléotide réductase (RNR), est responsable de la conversion des blocs de construction d'ARN en blocs de construction d'ADN, afin de construire de nouveaux brins d'ADN et de réparer les anciens. RNR est une cible pour les thérapies anticancéreuses, ainsi que des médicaments qui traitent les maladies virales comme le VIH/SIDA. Mais pendant des décennies, les scientifiques ont eu du mal à déterminer comment l'enzyme est activée parce que cela se produit si rapidement. Maintenant, pour la première fois, les chercheurs ont piégé l'enzyme dans son état actif et observé comment l'enzyme change de forme, rapprocher ses deux sous-unités et transférer l'énergie nécessaire pour produire les éléments constitutifs de l'assemblage de l'ADN.
Avant cette étude, beaucoup pensaient que les deux sous-unités de RNR s'assemblaient et s'accordaient avec une symétrie parfaite, comme une clé dans une serrure. « Depuis 30 ans, c'est ce que nous pensions, " dit Catherine Drennan, un professeur de chimie et de biologie au MIT et un chercheur du Howard Hughes Medical Institute. "Mais maintenant, on voit que le mouvement est beaucoup plus élégant. L'enzyme exécute en fait une "danse carrée moléculaire, ' où différentes parties de la protéine s'accrochent et se balancent autour d'autres parties. C'est vraiment très beau."
Drennan et JoAnne Stubbe, professeur émérite de chimie et de biologie au MIT, sont les auteurs principaux de l'étude, paru dans le journal Science le 26 mars. L'ancien étudiant diplômé Gyunghoon "Kenny" Kang Ph.D. '19 est l'auteur principal.
Toutes les protéines, dont RNR, sont composés d'unités fondamentales appelées acides aminés. Depuis plus d'une décennie, Le laboratoire de Stubbe a expérimenté la substitution des acides aminés naturels de RNR par des acides aminés synthétiques. Ce faisant, le laboratoire s'est rendu compte qu'ils pouvaient piéger l'enzyme dans son état actif et ralentir son retour à la normale. Cependant, ce n'est que lorsque le laboratoire Drennan a eu accès à une avancée technologique clé - la microscopie cryoélectronique - qu'il a pu prendre des images haute résolution de ces enzymes "piégées" du laboratoire Stubbe et les regarder de plus près.
« Nous n'avions vraiment pas fait de cryomicroscopie électronique au moment où nous avons activement commencé à essayer de faire l'impossible :obtenir la structure de RNR dans son état actif, " Drennan dit. " Je ne peux pas croire que cela a fonctionné; Je me pince encore."
La combinaison de ces techniques a permis à l'équipe de visualiser la danse moléculaire complexe qui permet à l'enzyme de transporter la « puissance de feu » catalytique d'une sous-unité à l'autre, afin de générer des blocs de construction d'ADN. Cette puissance de feu est dérivée d'un électron non apparié hautement réactif (un radical), qui doit être soigneusement contrôlé pour éviter d'endommager l'enzyme.
Selon Drennan, l'équipe "voulait voir comment RNR fait l'équivalent de jouer avec le feu sans se brûler".
Le premier auteur Kang dit que le ralentissement du transfert radical leur a permis d'observer des parties de l'enzyme que personne n'avait pu voir en entier auparavant. « Avant cette étude, nous savions que cette danse moléculaire se produisait, mais nous n'avions jamais vu la danse en action, " dit-il. " Mais maintenant que nous avons une structure pour RNR dans son état actif, nous avons une bien meilleure idée de la façon dont les différents composants de l'enzyme se déplacent et interagissent afin de transférer le radical sur de longues distances."
Bien que cette danse moléculaire rassemble les sous-unités, il y a encore une distance considérable entre eux :le radical doit parcourir 35-40 angströms de la première sous-unité à la seconde. Ce voyage est environ 10 fois plus loin que le transfert radical moyen, selon Drennan. Le radical doit ensuite retourner à son point de départ et être stocké en toute sécurité, le tout en une fraction de seconde avant que l'enzyme ne revienne à sa conformation normale.
Parce que le RNR est une cible pour les médicaments traitant le cancer et certains virus, connaître sa structure à l'état actif pourrait aider les chercheurs à concevoir des traitements plus efficaces. Comprendre l'état actif de l'enzyme pourrait également donner un aperçu du transport biologique des électrons pour des applications telles que les biocarburants. Drennan et Kang espèrent que leur étude encouragera d'autres personnes à capturer des événements cellulaires éphémères qui ont été difficiles à observer dans le passé.
"Nous devrons peut-être réévaluer des décennies de résultats passés, " Drennan dit. " Cette étude pourrait ouvrir plus de questions qu'elle n'apporte de réponses; c'est plus un début qu'une fin."
Cette histoire est republiée avec l'aimable autorisation de MIT News (web.mit.edu/newsoffice/), un site populaire qui couvre l'actualité de la recherche du MIT, innovation et enseignement.