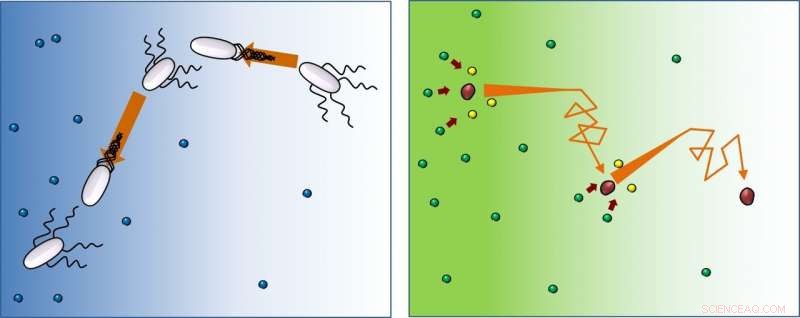
En présence de nourriture (bleu), les bactéries (blanches) nagent droit, puis changer d'orientation (tumble), et suivez ce modèle encore et encore. Cette étude montre que les enzymes (rouge) se déplacent de la même manière, mais après réaction avec les substrats (du vert au jaune), ils continuent à courir et à dégringoler loin de la concentration plus élevée de substrats. Crédit :IBS
Bien qu'associer les mots « aléatoire » et « partial » puisse sembler être un conflit, ce sont les attributs qui décrivent comment les bactéries naviguent et vont chercher leur nourriture. Ils suivent un chemin aléatoire, mais biaisé vers la direction de la source des nutriments. Scientifiques du Centre de la Matière Douce et Vivante, au sein de l'Institut des sciences fondamentales (IBS, Corée du Sud), observé que les enzymes, les molécules qui aident les réactions biologiques à se produire plus rapidement, déplacer avec un modèle similaire, mais dans le sens inverse, en se déplaçant au hasard vers la zone avec moins de substrats. Ces résultats, obtenu avec les nouvelles technologies optiques et publié comme article inaugural dans Actes de l'Académie nationale des sciences (PNAS) , sont particulièrement intrigantes car elles s'écartent de la vision traditionnelle.
Des milliers d'enzymes favorisent une multitude de réactions dans notre corps, dont chacun soutient nos vies. Les enzymes agissent sur d'autres produits chimiques appelés substrats et accélèrent les réactions chimiques qui convertissent les substrats en produits. Pour faire ça, enzymes et substrats doivent se rencontrer. On croyait à l'origine que les enzymes et leurs substrats correspondants se heurtaient accidentellement:"Les manuels standard mentionnent que les enzymes et les substrats sont censés se rapprocher" d'une certaine manière, " interagir, et c'est ainsi que les réactions se produisent. Nous avons essayé de creuser la partie « en quelque sorte » de ce processus. Après avoir collecté des milliers d'événements, nous avons conclu que la marche des enzymes est biaisée aléatoirement, ce qui signifie qu'ils sont microscopiquement directionnels, comme les bactéries, " explique Ah-Young Jee, premier auteur de cette étude.
Le mouvement des bactéries nageuses est connu sous le nom de « courir et culbuter » :tourner et répéter. Ils vont dans un sens, puis changer de direction au hasard, et répétez. Cependant, tandis que les bactéries s'orientent vers l'approvisionnement alimentaire, les enzymes se déplacent dans le sens d'une concentration moindre en substrat. "Les molécules manquent de capacités de prise de décision, mais se déplacent étonnamment vers des zones avec moins de substrat. En effet, le substrat introduit la polarisation de direction, et plus la concentration de substrat que nous avons utilisée dans l'expérience est élevée, plus cette tendance est forte. Nous émettons l'hypothèse que cela pourrait être un moyen d'uniformiser la concentration du produit autour du milieu, même lorsque la concentration du substrat n'est pas, " dit le professeur Tsvi Tlusty, qui a offert la compréhension théorique de cette recherche.
Comme chaque foulée d'enzyme ne mesure que 50 nanomètres de longueur, des appareils extrêmement précis sont nécessaires pour mesurer leur vitesse et leur orientation. L'équipe de recherche, dirigé par Steve Granick, regardé le mouvement enzymatique avec une technologie de microscopie à super-résolution, connue sous le nom de spectroscopie de corrélation de fluorescence à émission-appauvrissement stimulée, ou STED-FCS en abrégé.

A) Enzyme-substrat-buffer (E+S+B) entre dans l'entrée gauche, et le tampon enzymatique (E+B) est injecté à l'entrée droite. De cette façon, la concentration en enzyme est uniforme sur toute la puce, tandis que la concentration en substrat est plus élevée du côté gauche (bleu) diminuant progressivement vers la droite (blanc). Le rectangle en pointillés indique la zone où le mouvement de l'enzyme est étudié en détail à l'aide de la technologie de microscopie STED-FCS. B) La concentration de l'enzyme uréase (noir) et la concentration de l'urée (son substrat, gris) sont tracés en fonction de la position dans la zone FCS, entre 0 et 2,5 millimètres du faisceau laser. L'échelle à chaque point est une barre d'erreur, montrant l'écart type sur cinq mesures répétées. C) Le coefficient de diffusion (Da), qui indique à quelle vitesse l'enzyme se déplace, ne change pas en l'absence de substrat, mais il augmente lorsque le substrat est présent, ce qui signifie que l'enzyme se déplace plus rapidement vers la droite. Crédit :IBS, publie dans PNAS
Dans la méthode FCS, les enzymes sont décorées de molécules fluorescentes et leur passage à travers un petit point est détecté par un faisceau laser. Des informations statistiques sont recueillies, tels que le nombre d'enzymes traversant le point et leur vitesse (taux de diffusion). L'équipe a complété FCS avec la technologie STED, l'une des forces de ce centre de recherche. STED concentre le faisceau laser dans une zone extrêmement petite, permettant une mesure plus fine de la position des enzymes. Le faisceau laser FCS couvre une zone d'environ 250 nanomètres de diamètre, qui est environ 25 fois le diamètre de l'enzyme (10 nanomètres). STED-FCS est plus précis car il réduit le diamètre du faisceau à 50 nanomètres, qui est comparable à la distance de marche d'une enzyme.
Les chercheurs ont également conçu une nouvelle puce microfluidique, formé par des canaux d'une épaisseur micrométrique par lesquels les liquides peuvent passer. Études précédentes, en utilisant FCS sans STED et une puce microfluidique différente, suggèrent que les enzymes se déplacent vers la zone avec la plus forte concentration de substrat. Cependant, cette nouvelle technologie permet des observations plus approfondies et a montré le contraire. Les auteurs pensent que les résultats diffèrent parce que les informations détaillées sur le mouvement de l'enzyme ont été annulées à l'aide d'un faisceau laser plus large. Cela reviendrait à localiser quelqu'un dans un rayon de 50 kilomètres carrés ou de 2 kilomètres carrés, ce dernier est plus précis. Ils ont créé des conditions où l'enzyme est uniformément distribuée à l'intérieur de la puce, mais la concentration du substrat varie de gauche à droite :à partir d'une concentration minimale du côté droit de la puce, à une concentration maximale sur la gauche. Puis, ils ont utilisé FCS pour observer la concentration et la vitesse de l'enzyme à l'intérieur de la puce, et a constaté que l'enzyme a tendance à accélérer vers la zone avec une concentration de substrat moindre (c'est-à-dire vers le côté droit de la puce). Au lieu, en l'absence de substrat, les enzymes ne changent pas leur vitesse. De plus, les capacités de super-résolution de STED-FCS ont montré une dynamique d'exécution et de chute.
L'équipe a observé le même comportement avec deux couples enzyme-substrat séparément :uréase-urée et acétylcholinestérase (AChE)-acétylcholine. La première enzyme est utilisée par certains micro-organismes pour transformer l'urée en ammoniac et en acide carbonique, ce dernier joue un rôle essentiel dans les jonctions neuromusculaires.
À l'avenir, l'équipe de recherche vise à étendre la complexité de l'étude et à tester des conditions plus proches de la vie réelle :« Cette étude a examiné le comportement d'une enzyme en réponse à un substrat. Ensuite, nous prévoyons d'examiner plusieurs enzymes en même temps. Notre corps fonctionne avec une cascade de réactions, ce qui signifie qu'une réaction déclenche la suivante :l'enzyme A déclenche l'enzyme B, alors B déclenche C, etc. L'analyse STED-FCS pourrait être un outil clé pour faire avancer la recherche métabolique en révélant comment ces multiples systèmes enzymatiques sont organiquement liés, " conclut Jee.