Une nouvelle étude de l’Université hébraïque révèle des informations sur la régulation de l’ARNm au cours du développement embryonnaire. L'étude met en lumière le processus complexe de régulation de l'ARNm au cours du développement embryonnaire, fournissant ainsi de nouvelles informations sur la façon dont les cellules pluripotentes adoptent des identités spécialisées grâce à l'expression des gènes.
Les travaux sont publiés dans la revue Nature Communications .La recherche a été dirigée par un doctorat. L'étudiant Lior Fishman et son équipe sous la direction du chercheur Dr Michal Rabani de l'Institut Alexander Silberman des sciences de la vie de l'Université hébraïque, et en collaboration avec des chercheurs des National Institutes of Health des États-Unis.
Le développement embryonnaire implique des cellules pluripotentes assumant des identités spécialisées en adoptant des profils d'expression génique particuliers. Cependant, comprendre les contributions relatives de la transcription et de la dégradation de l'ARNm à la formation de ces profils s'est avéré difficile, en particulier au sein d'embryons aux identités cellulaires diverses.
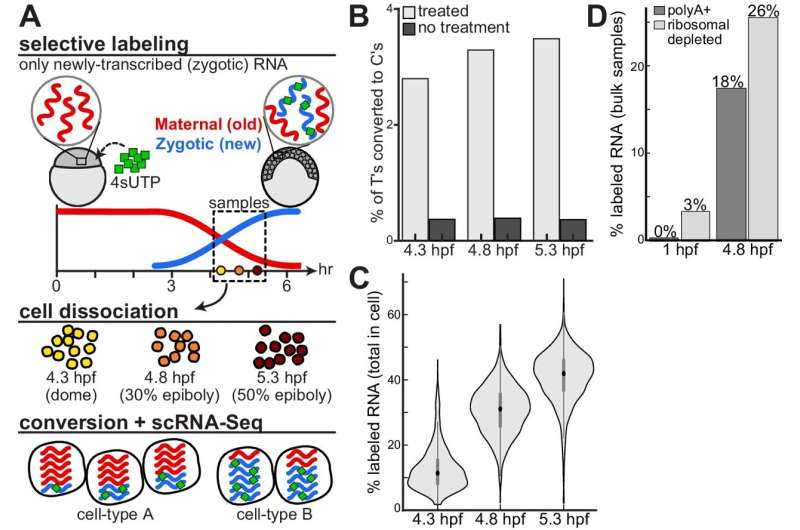
Dans l’étude, les chercheurs ont utilisé une technique appelée séquençage de l’ARN unicellulaire ainsi que le marquage métabolique pour suivre la manière dont les gènes sont activés et désactivés au fil du temps dans les embryons de poisson zèbre. Ils pouvaient distinguer l’ARNm qui avait été créé (à partir de l’embryon lui-même) et l’ARNm qui était déjà là (à partir de la mère). À l'aide de modèles mathématiques, ils ont mesuré la rapidité avec laquelle les gènes étaient activés et désactivés dans différents types de cellules au fur et à mesure de leur développement.
Les résultats de l’étude révèlent des taux de régulation très variés sur des milliers de gènes. Les chercheurs ont observé des taux de transcription et de destruction coordonnés pour de nombreux transcrits et ont lié les différences de dégradation à des éléments de séquence spécifiques. Plus important encore, ils ont identifié des différences de dégradation spécifiques à chaque type de cellule, notamment la rétention sélective des transcrits maternels dans les cellules germinales primordiales et les cellules de la couche enveloppante, deux des premiers types de cellules spécifiés.
Le Dr Rabani, auteur principal de l'étude, a commenté :« Notre étude propose une approche quantitative pour étudier la régulation de l'ARNm au cours d'une réponse spatio-temporelle dynamique. Ce travail ouvre de nouvelles voies pour comprendre les mécanismes moléculaires sous-jacents à la détermination du destin cellulaire au cours du développement embryonnaire. "
Les résultats de cette étude contribuent à une compréhension plus approfondie de la régulation de l’ARNm et de son rôle dans la formation des identités cellulaires au cours du développement embryonnaire. L'équipe de recherche espère que leurs travaux ouvriront la voie à de futures études visant à élucider les complexités de la régulation de l'expression génique dans divers contextes biologiques.
Plus d'informations : Lior Fishman et al, Transcription et cinétique de dégradation de l'ARNm spécifique à un type de cellule dans l'embryogenèse du poisson zèbre à partir d'un séquençage d'ARN unicellulaire marqué métaboliquement, Nature Communications (2024). DOI :10.1038/s41467-024-47290-9
Informations sur le journal : Communications naturelles
Fourni par l'Université hébraïque de Jérusalem