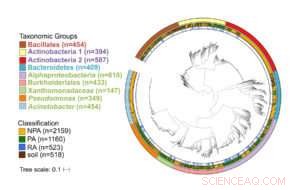
Arbre phylogénétique de plus de 3, 800 génomes bactériens de haute qualité et non redondants. L'anneau extérieur désigne le groupe taxonomique, l'anneau central désigne la source d'isolement, et l'anneau interne désigne les génomes associés aux racines dans les génomes associés aux plantes. Les noms des taxons sont codés par couleur en fonction du phylum :vert – Protéobactéries, rouge – Firmicutes, bleu –Bacteroidetes, violet – Actinobactéries. Crédit :Asaf Levy
Alors que la population mondiale augmente, estimé à près de 10 milliards d'ici 2050, il en va de même de la nécessité d'augmenter les rendements des cultures et de produire suffisamment de matériel végétal pour l'alimentation et les carburants alternatifs durables. Pour aider à améliorer les stratégies de sélection des cultures et surmonter les défis tels que rendre les plantes plus tolérantes aux terres marginales, et les stress tels que la sécheresse et la faible disponibilité des nutriments, les chercheurs se concentrent sur la compréhension et la promotion des relations bénéfiques entre les plantes et les microbes.
Publié le 18 décembre Numéro 2017 dans Génétique de la nature , une équipe dirigée par des chercheurs du Joint Genome Institute (JGI) du département américain de l'Énergie (DOE), une installation utilisateur du DOE Office of Science, et le Howard Hughes Medical Institute de l'Université de Caroline du Nord à Chapel Hill (UNC) ont exploité un catalogue de génomes bactériens pour identifier et caractériser les gènes candidats qui aident les bactéries à s'adapter aux environnements végétaux, spécifiquement les gènes impliqués dans la colonisation bactérienne des racines.
La plupart des études sur le terrain à ce jour ont porté sur la structure communautaire du microbiome végétal, c'est-à-dire "qui est là, " et moins sur la fonction, c'est-à-dire "ce qu'ils font, comment et quand ils le font. » Les études précédentes qui ont examiné la fonction ont principalement examiné une seule interaction hôte-microbe, comme celui entre une plante Arabidopsis et un agent pathogène.
« Si nous voulons concevoir le bon microbiome pour soutenir la croissance des plantes, nous devons comprendre la fonction réelle du microbiome et pas seulement les gènes marqueurs de séquence, " a déclaré le co-premier auteur de l'étude Asaf Levy, chercheur au JGI. « Ici, nous avons déployé des efforts considérables en matière de génomique et de calcul pour répondre à la question fondamentale et importante : « Comment le microbiome végétal interagit-il avec la plante ? » »
La plupart des interactions entre les microbes et les plantes se produisent à l'interface entre les racines et le sol. Chercheurs de l'UNC, Laboratoire national d'Oak Ridge, et le Max Planck Institute a isolé de nouvelles bactéries de l'environnement racinaire de Brassicaceae (191), peupliers (135), et le maïs (51). Les génomes de ces 377 isolats bactériens, plus 107 cellules bactériennes individuelles supplémentaires provenant de racines d'A. thaliana, ont ensuite été séquencés, assemblé, et annoté au JGI.
Les auteurs ont ensuite combiné les nouveaux génomes avec des milliers de génomes accessibles au public qui représentent les principaux groupes de bactéries associées aux plantes, et inclus des bactéries provenant de plusieurs environnements végétaux et non végétaux, comme l'intestin humain, en comparaison. La base de données résultante de 3837 génomes, dont 1160 d'origine végétale, a été utilisé dans une analyse génomique comparative.
Les chercheurs ont ensuite identifié des gènes enrichis dans les génomes d'organismes associés aux plantes et aux racines.
« Il est très important pour nous de comprendre les gènes et les fonctions que les microbes utilisent pour coloniser les plantes, car ce n'est qu'alors que nous aurons une chance de concevoir rationnellement des « probiotiques végétaux » utiles pour nous aider à produire plus de cultures vivrières et énergétiques avec moins d'intrants chimiques tels que des engrais et pesticides ou fongicides, " a déclaré l'auteur principal de l'étude Jeff Dangl, un chercheur du Howard Hughes Medical Institute et le professeur de biologie John N. Couch à l'Université de Caroline du Nord à Chapel Hill
L'une des principales conclusions tirées de l'étude était que les génomes associés aux plantes et au sol ont tendance à être plus gros que les génomes témoins du même clade. Cela s'est avéré être dû en partie à l'enrichissement des gènes impliqués dans le métabolisme et le transport du sucre, probablement une adaptation au carbone végétal dérivé de la photosynthèse, générés par les "usines de bonbons" de la nature, ", a déclaré Asaf Levy. Jusqu'à 20% du carbone fixé par les plantes par photosynthèse est exsudé par les racines sous forme de sucres pour attirer les microbes.
De nombreux gènes qui semblent imiter les fonctions des plantes – en codant des « Domaines PA et RA ressemblant à des plantes » ou PREPARADO – ont également été identifiés. « Il est bien connu que les agents pathogènes des plantes utilisent des protéines qui imitent les domaines végétaux nécessaires à la fonction immunitaire, " a déclaré Dangl. "Imaginez que l'agent pathogène injecte directement dans la cellule végétale une protéine qui imite une partie d'une machine particulière du système immunitaire. C'est comme mettre un rouage partiellement défectueux dans une roue :les roues ne peuvent plus tourner. Nous pensons que les domaines protéiques associés aux plantes que nous avons identifiés pourraient fonctionner de la même manière. »
Les gènes en évolution rapide sont souvent la signature d'une course aux armements moléculaires entre des organismes partageant un environnement. Ces gènes sont souvent utilisés en attaque ou en défense contre d'autres organismes. Deux nouvelles familles de protéines à évolution rapide associées à différents « modes de vie » de bactéries associées aux plantes ont été identifiées dans l'étude. Une, trouvé dans les bactéries commensales, a été surnommé "Jekyll"; L'autre, trouvé dans les bactéries pathogènes, s'appelait "Hyde". Avec des collaborateurs de Virginia Tech et de l'ETH (Suisse), Les scientifiques du JGI ont découvert que ces derniers sont très efficaces pour tuer les bactéries concurrentes, potentiellement pour aider ces "Hydes" à prendre le contrôle de la niche des feuilles.
Le catalogue complet des nouveaux génomes et gènes associés aux plantes est disponible pour la communauté de recherche via un portail web dédié :Caractéristiques génomiques de l'adaptation bactérienne aux plantes.
« La base de données est une ressource précieuse pour la communauté des chercheurs qui étudient les interactions plante-microbe, car c'est un moyen impartial d'identifier des gènes potentiellement intéressants impliqués dans l'interaction avec une plante, y compris de nombreux gènes totalement nouveaux. Nous étudions actuellement expérimentalement la fonction de plusieurs des ces gènes pour acquérir une meilleure compréhension fonctionnelle du microbiome végétal." dit Lévy.