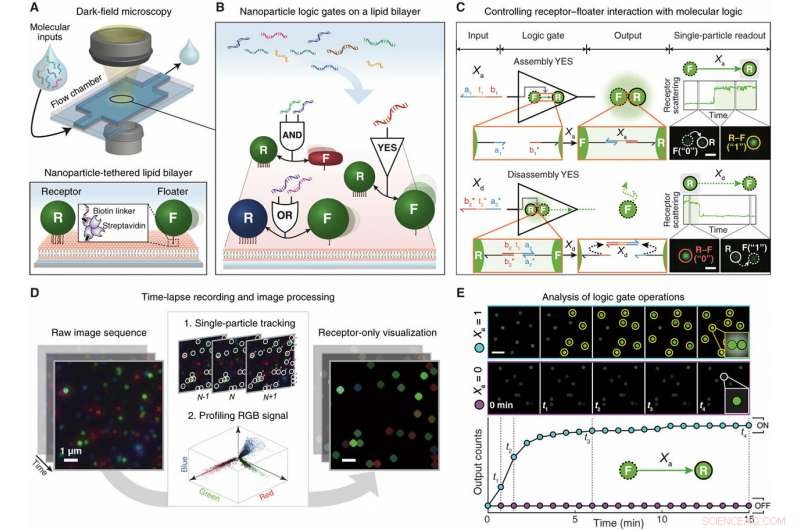
Calcul logique à nanoparticule unique sur LNTs. (A) Schémas de la plate-forme LNT. Deux types de nanoparticules modifiées par l'ADN, récepteur immobile (R) et flotteur mobile (F), sont attachés à un SLB et surveillés par DFM. (B) Paires R–F en tant que portes logiques booléennes de nanoparticules. Chaque porte logique prend l'ADN en entrée et produit soit un assemblage, soit un désassemblage entre les deux particules en sortie. Les flèches bidirectionnelles indiquent les interactions R–F. Les ligands d'ADN de surface ne sont pas représentés. (C) Portes OUI à nanoparticule unique. Les domaines fonctionnels sont représentés par des numéros de couleur et en indice avec des pointes de flèche indiquant leurs extrémités 3'. Les astérisques indiquent la complémentarité. Les cercles lumineux derrière les dimères R–F illustrent le couplage plasmonique. (D) Analyse d'images. Un algorithme de suivi de particule unique identifie d'abord les signaux des récepteurs à partir d'une séquence d'images brutes. Après, les signaux détectés sont échantillonnés et utilisés pour générer un nouveau film à fond noir qui visualise uniquement les signaux des récepteurs. (E) Analyse cinétique. Des instantanés du récepteur uniquement (en haut) et un tracé cinétique (en bas) de la porte Assembly YES sont fournis pour chaque condition d'entrée. Un graphique de cinétique est obtenu en comptant cumulativement le nombre de récepteurs à changement d'état au cours du temps. Crédit :Avancées scientifiques, doi:10.1126/sciadv.aau2124
Les nanoparticules peuvent être utilisées comme substrats pour le calcul, avec un contrôle algorithmique et autonome de leurs propriétés uniques. Cependant, l'architecture évolutive pour former des systèmes informatiques à base de nanoparticules fait actuellement défaut. Dans une étude récente publiée dans Avancées scientifiques , Jinyoung Seo et ses collègues du Département de chimie de l'Université nationale de Séoul en Corée du Sud, rapporté sur une plate-forme de nanoparticules intégrée avec des portes logiques et des circuits au niveau de la particule unique. Ils ont mis en œuvre la plate-forme sur une bicouche lipidique de support. Inspiré des membranes cellulaires en biologie qui compartimentent et contrôlent les réseaux de signalisation, les scientifiques ont appelé la plateforme « lipid nanotablet » (LNT). Pour conduire la nano-bio-informatique, ils ont utilisé une bicouche lipidique comme carte de circuit chimique et les nanoparticules comme unités de calcul.
Sur un nanocomprimé lipidique en solution, Seo et al. ont établi qu'une seule porte logique de nanoparticules détecte les molécules en tant qu'entrées et déclenche l'assemblage ou le désassemblage des particules en tant que sortie. Dans l'étude, ils ont démontré des opérations logiques booléennes aux côtés d'une entrée/sortie de portes logiques et d'un circuit logique combinatoire en tant que multiplexeur. Les scientifiques envisagent que la nouvelle approche serait capable de moduler des circuits de nanoparticules sur des bicouches lipidiques pour concevoir de nouveaux paradigmes et passerelles en informatique moléculaire, circuits de nanoparticules et nanosciences des systèmes, à l'avenir.
La matière peut être fusionnée avec le calcul sur de nombreuses échelles de longueur, allant des gouttelettes microscopiques dans la logique des bulles microfluidiques et des microparticules aux biomolécules et aux machines moléculaires. La mise en œuvre du calcul dans les nanoparticules reste inexplorée, malgré un large éventail d'applications qui pourraient bénéficier de la possibilité de contrôler algorithmiquement la photonique utile, électrique, magnétique, propriétés catalytiques et matérielles des nanoparticules. Ces propriétés sont actuellement inaccessibles via les systèmes moléculaires. Idéalement, des systèmes de nanoparticules équipés de capacités informatiques peuvent former des circuits de nanoparticules pour effectuer de manière autonome des tâches complexes en réponse à des stimuli externes pour combiner le flux de matière et d'informations à l'échelle nanométrique.
Une approche existante pour utiliser les nanoparticules comme substrats pour le calcul consiste à fonctionnaliser les particules avec des ligands sensibles aux stimuli. Un groupe de ces nanoparticules modifiées effectuera ensuite des opérations logiques élémentaires qui répondent à une variété d'entrées chimiques et physiques. Les scientifiques visent à utiliser une nanoparticule individuelle en tant que nanoparties modulaires et à mettre en œuvre un calcul souhaité de manière plug-and-play. Cependant, il y a des difficultés à câbler plusieurs portes logiques intégrées dans la phase de solution car il est difficile de contrôler la diffusion des entrées, portes logiques et sortie dans l'espace 3D. Pour résoudre ce défi, les scientifiques se sont inspirés de la membrane cellulaire; un équivalent biologique d'une carte de circuit imprimé pouvant héberger une variété de protéines réceptrices en tant qu'unités de calcul. Dans la nature, les protéines compartimentées interagissent avec les récepteurs en tant que réseau pour effectuer des fonctions complexes. Les membranes peuvent également permettre à des processus informatiques parallèles de se produire et, par conséquent, les scientifiques des matériaux ont été inspirés pour recâbler le phénomène biologique.

Imagerie en champ sombre time-lapse d'une porte OUI d'assemblage de nanoparticules. Crédit :Avancées scientifiques, doi:10.1126/sciadv.aau2124.
Bioinspiré des membranes cellulaires, dans la présente étude, Seo et al. a démontré une plate-forme de calcul de nanoparticules à base de bicouche lipidique. Comme preuve de principe, ils ont utilisé des nanoparticules plasmoniques diffusant la lumière pour construire des composants de circuit, L'ADN en tant que ligands de surface et entrées moléculaires aux côtés des interactions biotine-streptavidine pour attacher les nanoparticules à la bicouche lipidique. Après fixation des nanoparticules sur une bicouche lipidique supportée (SLB), ils ont fourni plusieurs caractéristiques clés dans les expériences;
Les scientifiques ont mis en œuvre le nano-bio calcul à l'interface des nanostructures et des biomolécules, où l'information moléculaire en solution (entrée) a été traduite en un assemblage/désassemblage dynamique de nanoparticules sur une bicouche lipidique (sortie). En tant que composant clé d'un LNT, Seo et al. conçu une chambre d'écoulement avec une bicouche lipidique enduite au fond du substrat.
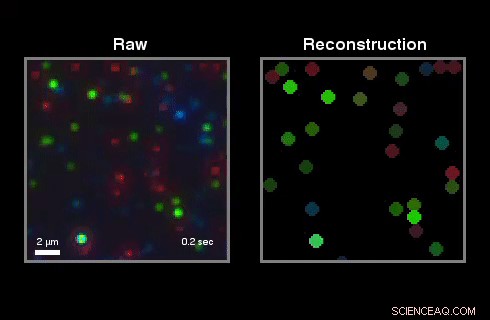
La visualisation par récepteur uniquement d'un film sur fond noir. Crédit :Avancées scientifiques, doi:10.1126/sciadv.aau2124.
Pour construire des nanocomprimés lipidiques dans le dispositif expérimental, les scientifiques ont utilisé trois composants clés - de petites vésicules unilamellaires (SUV), des chambres à flux en verre et des nanoparticules plasmoniques fonctionnalisées par l'ADN. Les nanoparticules modifiées par l'ADN adhéraient à la bicouche lipidique pour former des portes logiques et des circuits qui traitaient l'information moléculaire. Les scientifiques ont classé les nanoparticules fonctionnalisées en récepteurs immobiles (reporters pour le calcul) ou en flotteurs mobiles (porteurs d'information du calcul). Dans ce contexte, les flotteurs étaient des "fils" qui transportaient des informations des portes amont dans les portes aval par une diffusion latérale robuste. Ils ont caractérisé les nanoparticules pour valider leurs propriétés matérielles avant de construire les circuits expérimentaux.
Seo et al. utilisé l'imagerie par microscopie à fond noir (DFM) pour mesurer les performances des portes logiques des nanoparticules en réponse aux entrées moléculaires en solution. Lorsque des séquences d'images en fond noir ont été obtenues à partir des opérations logiques, le scientifique les a traitées et quantifiées à l'aide d'un pipeline d'analyse d'images personnalisé.
Tout à fait, les scientifiques ont conçu des portes logiques booléennes à nanoparticules et des opérations d'assemblage et de désassemblage de portes OUI à nanoparticule unique en temps réel. Les portes OUI à nanoparticule unique ont constitué les exemples les plus simples de l'étude. Pour détecter les signaux de diffusion d'une porte logique à nanoparticules, les scientifiques se sont appuyés sur le couplage plasmonique entre deux particules centrales qui composaient la porte. Pour former les nanoparticules, Seo et al. nanotiges d'or synthétisées avec des coquilles d'argent, nanosphères d'or et nanosphères d'argent sur des graines d'or dites rouges, des nanoparticules vertes et bleues pour présenter du rouge, signaux de diffusion vert et bleu dans l'étude. Les scientifiques ont représenté le comportement des nanoparticules à déclenchement logique d'une manière simple, graphique de réaction de nanoparticules pour montrer une réaction d'assemblage d'un flotteur à un récepteur et une réaction de désassemblage, fournissant une vue intuitive sur le comportement de chaque porte logique de nanoparticule.
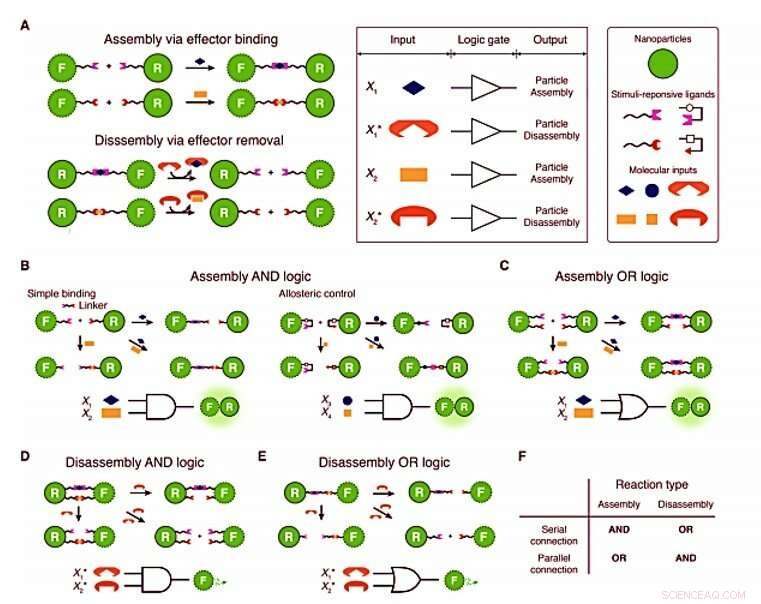
Principes de conception des portes logiques à nanoparticules. (A) Résumé graphique du concept généralisable. L'illustration des portes OUI d'assemblage/désassemblage de nanoparticules à médiation effectrice (à gauche) et la table de vérité pour le concept (à droite) sont fournies. Une paire effecteur-ligand sélective et une paire effecteur-chélateur sont nécessaires pour la construction de portes logiques d'assemblage/désassemblage. Pour construire une porte logique à l'aide de deux nanoparticules, Les interactions de « liaison » dans l'interface récepteur-flotteur doivent être programmées de manière à ce que les liaisons soient formées (via l'assemblage) ou clivées (via le désassemblage) uniquement si deux entrées moléculaires satisfont à la logique ET ou OU. (B) Two-input Assembly AND gate. (C) Two-input Assembly OR gate. Assembly reactions are controlled by AND logic when the bond-forming interaction require the serial activation by the two inputs and by OR logic when the bond-forming interaction is controlled in parallel. (D) Two-input Disassembly AND gate. (E) Two-input Disassembly OR gate. De la même manière, Disassembly reactions are modulated by AND logic via parallel disconnection and by OR logic via serial disconnection. (F) Table summary. These illustrations describe the generalized concept of the interface programming. Dans cette étude, we used sequence recognition and strand displacement of DNA as the mechanisms to implement the logic. Spécifiquement, we used single-stranded DNA molecules as effectors, thiolated oligonucleotides as ligands, and a strand displacement as chelation mechanism. We foresee that this design rules can be potentially applied to other ligand systems and core nanostructures. Crédit :Avancées scientifiques, doi:10.1126/sciadv.aau2124.
The scientists used a sufficiently high density of nanoparticles and incorporated single-particle tracking algorithms to profile the scattering signals and visualize the receptor signals alone in a dark background. To qualitatively understand the overall computing performance of a single nanoparticle logic gate, they used the "receptor-only" view. The results showed that the population of nanoparticle logic gates switched into the ON state in response to performing a YES logic operation. The scientists deduced that a population of nanoparticle logic gates produced high output counts only when the molecular input met TRUE conditions.
To demonstrate two-input, single-nanoparticle logic gates, Seo et al. similarly developed:Assembly AND, Assembly OR, Disassembly AND, and Disassembly OR gates via "interface programming". The scientists showed that the design principles for interface programming were straightforward and could be generalized among circuits. They expanded the interface programming to enable nanoparticle logic gates to process INHIBIT logic.
The scientists then increased the complexity of reactions at the receptor-floater interface but noticed incomplete reactions or spurious interactions occuring in the system. Such anomalous interactions indicated that they could not rely on programming particle interfaces as before to construct complex circuits. Au lieu, they introduced a conceptually distinct approach termed nanoparticle "network programming" to allow two single-particle logic gates to be combined with AND or, OR logic. In the resulting network programming of wired nanoparticle logic gates, the scientists showed the strategy could be implemented to build complex multilayer cascades readily without extensive optimization. Seo et al. successfully implemented the nanoparticle multiplexer to show the ability to design and operate nanoparticle circuits on LNTs in a highly modular and controlled manner.
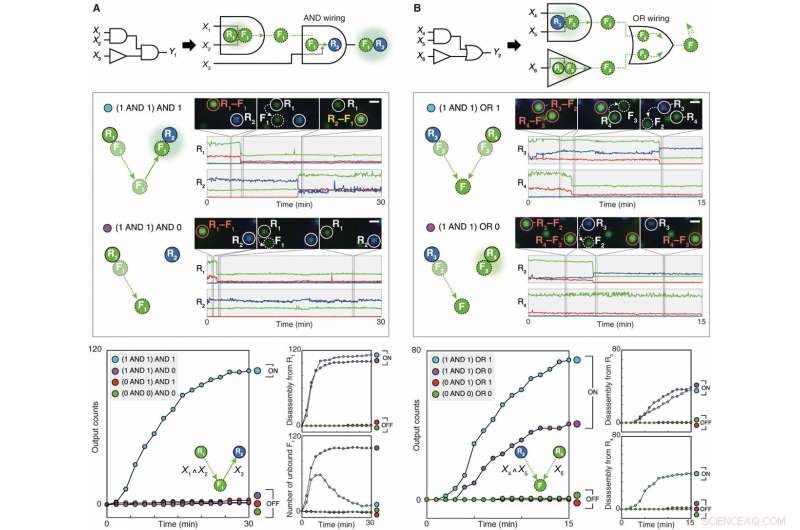
Wiring of nanoparticle logic gates via network programming. (A) Wiring with AND logic. Two logic gates (Disassembly AND gate and Assembly YES gate) are designed to operate in series for AND wiring. The floater F1, which is bound to the first receptor R1 in its initial state, acts as a Disassembly AND logic gate and subsequently as an Assembly YES gate with the second receptor R2. The generation of R2–F1 dimers is an output of the (X1 AND X2) AND X3 circuit. (B) Wiring with OR logic. Two logic gates (Disassembly AND gate and Disassembly YES gate) are designed to operate in parallel for OR wiring. The two gates both release G-NFs as outputs. The generation of the G-NFs is an output of the (X3 AND X4) OR X5 circuit. Circuit diagrams (top), single-particle dark-field analysis (middle), and kinetics analysis of circuits (lower left) and intermediate reactions (lower right). Crédit :Avancées scientifiques, doi:10.1126/sciadv.aau2124
Scientists can expand on the demonstrated scope of lipid bilayer-based nanoparticle computation to advance the existing molecular computing technologies to operate nanoparticle circuits. They can also integrate lipid bilayers with DNA nanostructures to open the development of new molecular circuits by expanding on dynamic inter-origami interactions for more complex and practical molecular computations. Current limits of the experimental setup prevent the construction of arbitrarily large circuits. These can be overcome to generate broader design space for circuit buildup with new modes of communication, dynamic reconfiguration and DNA walkers.
Seo et al. envision that the molecular computing network can be analogously built in a similar approach to silicon-based computers that have improved through the years. The scientists can advance the experimental setup by increasing the nanoparticle density, to increase the computing capacity and expand parallelism, so that each nanoparticle may independently perform its own computation. Pour des applications pratiques, the lipid nanotablet will play a pivotal role in building dynamic, autonomous nanosystems in molecular diagnostics and smart sensors; to sense multiple stimuli and trigger the appropriate response. If such nanocircuits are introduced into living cell membranes, scientists can create novel bioengineered nano-bio interfaces as biologic-inorganic hybrid systems. The particles can also be used separately to study membrane-associated phenomena in living cells. De cette façon, by facilitating communication between nanosystems and cellular systems, the scientists will be able to activate new pathways to navigate complex and dynamic theranostic applications.
© 2019 Réseau Science X