Image colorée au microscope à force atomique de structures arborescentes aléatoires auto-assemblées à la surface de matrices de tuiles d'ADN. Chaque arbre a une seule boucle en tant que "racine". Crédit :Caltech / Grigory Tikhomirov, Philip Petersen et Lulu Qian.
De nombreux systèmes auto-organisés dans la nature exploitent un mélange sophistiqué de processus déterministes et aléatoires. Il n'y a pas deux arbres identiques car la croissance est aléatoire, mais un séquoia peut être facilement distingué d'un jacaranda car les deux espèces suivent des programmes génétiques différents. La valeur du hasard dans les organismes biologiques n'est pas entièrement comprise, mais il a été émis l'hypothèse que cela permet des tailles de génome plus petites, car tous les détails ne doivent pas être codés. Le caractère aléatoire fournit également la variation sous-jacente à l'évolution adaptative.
Contrairement à la biologie, l'ingénierie profite rarement du pouvoir du hasard pour fabriquer des structures complexes. Maintenant, un groupe de scientifiques de Caltech a démontré que le caractère aléatoire de l'auto-assemblage moléculaire peut être combiné à des règles déterministes pour produire des nanostructures complexes à partir d'ADN.
L'oeuvre, fait dans le laboratoire du professeur adjoint de bio-ingénierie Lulu Qian, paraît dans le numéro du 28 novembre de la revue Nature Nanotechnologie .
Les êtres vivants utilisent l'ADN pour stocker des informations génétiques, mais l'ADN peut également être utilisé comme un bloc de construction chimique robuste pour l'ingénierie moléculaire. Les quatre molécules complémentaires qui composent l'ADN, appelés nucléotides, ne se lient que de manière spécifique :les A se lient aux T, et les G se lient aux C. En 2006, Paul Rothemund (BS '94), professeur-chercheur en bio-ingénierie, informatique et sciences mathématiques, et le calcul et les systèmes neuronaux à Caltech, a inventé une technique appelée ADN origami qui tire parti de l'appariement entre de longs brins de nucléotides d'ADN, en les pliant dans tout, des œuvres d'art à l'échelle nanométrique aux dispositifs d'administration de médicaments. Les structures auto-assemblées formées par l'origami d'ADN peuvent être fonctionnelles par elles-mêmes ou elles peuvent être utilisées comme modèles pour organiser d'autres molécules fonctionnelles, telles que les nanotubes de carbone, protéines, nanoparticules métalliques, et des colorants organiques, avec une programmabilité et une précision spatiale sans précédent.
En utilisant l'origami ADN comme bloc de construction, les chercheurs ont fabriqué des nanostructures d'ADN plus grandes, tels que des tableaux périodiques de tuiles d'origami. Cependant, parce que le bloc de construction est simplement répété partout, la complexité des motifs qui peuvent être formés sur ces plus grandes structures est assez limitée. Des processus d'assemblage entièrement déterministes - contrôlant la conception de chaque tuile individuelle et sa position distincte dans le réseau - peuvent donner lieu à des motifs complexes, mais ces processus ne s'étendent pas bien. Inversement, si seuls des processus aléatoires sont impliqués et que les caractéristiques globales du tableau ne sont pas contrôlées par des règles de conception, il est impossible de créer des motifs complexes avec les propriétés souhaitées sans générer simultanément une grande fraction de molécules indésirables qui sont gaspillées. Jusqu'au travail de Qian et de ses collègues, combiner des processus déterministes avec des processus aléatoires n'avait jamais été systématiquement exploré pour créer des nanostructures d'ADN complexes.

La gauche, Les tuiles Truchet ont deux arcs qui sont asymétriques en rotation. Droit, jeux de société populaires inspirés des tuiles Truchet. Crédit :Avec l'aimable autorisation de L. Qian
"Nous recherchions des principes d'auto-assemblage moléculaire qui englobent à la fois les aspects déterministes et aléatoires, ", explique Qian. "Nous avons développé un ensemble simple de règles qui permettent aux tuiles d'ADN de se lier de manière aléatoire, mais uniquement selon des modèles contrôlés spécifiques."
L'approche consiste à concevoir des motifs sur des carreaux individuels, moduler les ratios des différentes tuiles, et déterminer quelles tuiles peuvent se lier pendant l'auto-assemblage. Cela conduit à des caractéristiques émergentes à grande échelle avec des propriétés statistiques réglables, un phénomène que les auteurs appellent « désordre programmable ».
"Les structures que nous pouvons construire ont des aspects aléatoires programmables, " dit Grigori Tikhomirov, un chercheur postdoctoral senior en biologie et génie biologique, et auteur principal de l'article. "Par exemple, nous pouvons faire des structures qui ont des lignes qui prennent des chemins apparemment aléatoires, mais nous pouvons nous assurer qu'ils ne se croisent jamais et finissent toujours par se refermer en boucles."
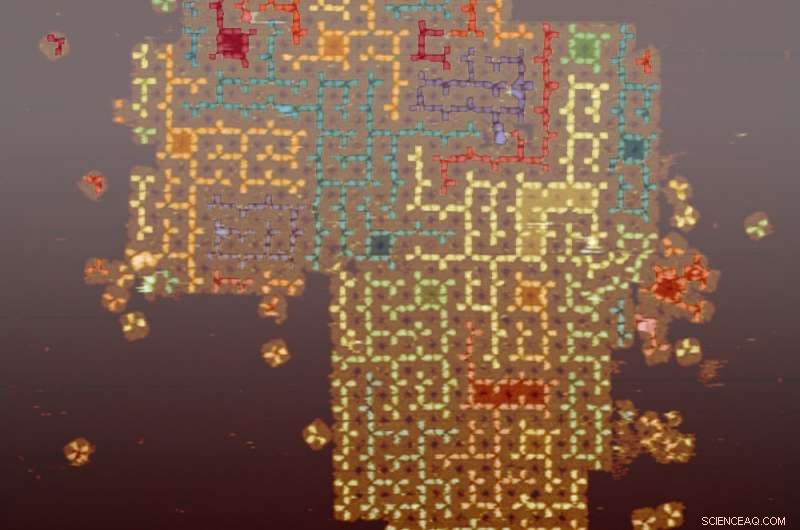
En plus des boucles, l'équipe a choisi deux autres exemples, labyrinthes et arbres, pour démontrer que de nombreuses propriétés non triviales de ces structures peuvent être contrôlées par de simples règles locales. Ils ont trouvé ces exemples intéressants car loop, Labyrinthe, et les structures arborescentes existent largement dans la nature à plusieurs échelles. Par exemple, les poumons sont des structures arborescentes à l'échelle du millimètre au centimètre, et les dendrites neurales sont des structures arborescentes à l'échelle du micromètre au millimètre. Les propriétés contrôlées qu'ils ont montrées incluent les règles de branchement, les axes de croissance, la proximité entre réseaux adjacents, et la distribution des tailles.
Le groupe s'est d'abord inspiré des carreaux classiques Truchet, qui sont des carreaux carrés avec deux arcs d'ADN symétriques en diagonale sur la surface. Il existe deux orientations asymétriques en rotation du motif en arc. Permettant un choix aléatoire des deux orientations des tuiles à chaque emplacement du réseau, le motif continuera à travers les tuiles voisines, soit devenir des boucles de différentes tailles, soit sortir d'un bord du tableau.

Boucle auto-assemblée, Labyrinthe, et les structures arborescentes à la surface des puces à ADN. Rangée du haut, des labyrinthes aléatoires avec des jonctions à trois et quatre voies de distances variables entre des jonctions adjacentes par rapport à seulement des jonctions à trois voies d'une distance fixe entre des jonctions adjacentes. Rangée du milieu, arbres aléatoires (chaque arbre a une seule boucle en tant que "racine") avec des branches plus longues de longueurs variables par rapport à des branches plus courtes de longueurs fixes. Rangée du bas, boucles aléatoires avec des longueurs et un nombre de croisements réglables. Crédit :L. Qian
Pour créer des puces Truchet à l'échelle moléculaire, l'équipe a utilisé la technique de l'origami d'ADN pour plier l'ADN en tuiles carrées, puis a conçu les interactions entre ces tuiles pour les encourager à s'auto-assembler en de grands réseaux bidimensionnels.
"Parce que toutes les molécules se heurtent en flottant dans un tube à essai pendant le processus d'auto-assemblage, les interactions doivent être suffisamment faibles pour permettre aux tuiles de se réorganiser et d'éviter d'être piégés dans des configurations non souhaitées, " dit Philip Petersen, un étudiant diplômé du laboratoire Qian et co-premier auteur de l'article. "D'autre part, les interactions doivent être suffisamment spécifiques pour que les interactions souhaitées soient toujours préférées aux indésirables, interactions parasites. »
Différents types de modèles globaux émergent lorsque les carreaux sont marqués avec différents modèles locaux. Par exemple, si chaque tuile orientée aléatoirement porte un "T" plutôt que deux arcs, le modèle global est un labyrinthe avec des branches et des boucles plutôt que seulement des boucles. Si les règles d'auto-assemblage contraignent l'éventuelle orientation relative des tuiles "T" voisines, il est possible de s'assurer qu'autre qu'une seule "racine, " les branches dans les labyrinthes ne se referment jamais en boucles - produisant des arbres. Pour explorer la pleine généralité de ces principes, L'équipe de Qian a développé un langage de programmation pour les pavages aléatoires d'origami d'ADN.
"Avec ce langage de programmation, le processus de conception commence par une description de haut niveau des tuiles et des tableaux, qui peuvent être automatiquement traduits en diagrammes matriciels abstraits et en simulations numériques, passe ensuite à la conception de tuiles d'origami ADN, y compris la façon dont les tuiles interagissent les unes avec les autres sur leurs bords. Finalement, nous concevons des séquences d'ADN, " dit Qian. "Avec ces séquences d'ADN, il est simple pour les chercheurs d'ordonner les brins d'ADN, les mélanger dans un tube à essai, attendre que les molécules s'auto-assemblent dans les structures conçues pendant la nuit, et obtenir des images des structures à l'aide d'un microscope à force atomique."
La méthode de désordre programmable du groupe a diverses applications futures. Par exemple, il pourrait être utilisé pour créer des environnements de test complexes pour des robots moléculaires de plus en plus sophistiqués, des machines nanométriques basées sur l'ADN qui peuvent se déplacer sur une surface, ramasser ou déposer des protéines ou d'autres types de molécules en tant que cargaisons, et prendre des décisions concernant la navigation et les actions.
« Les applications potentielles sont beaucoup plus larges, " ajoute Qian. Depuis les années 1990, des chaînes unidimensionnelles aléatoires de polymères ont été utilisées pour révolutionner la synthèse chimique et matérielle, l'administration de médicaments, et la chimie des acides nucléiques en créant de vastes bibliothèques combinatoires de molécules candidates, puis en sélectionnant ou en faisant évoluer les meilleures en laboratoire. "Notre travail étend le même principe aux réseaux de molécules bidimensionnels et crée désormais de nouvelles opportunités pour la fabrication de dispositifs moléculaires plus complexes organisés par des nanostructures d'ADN, " elle dit.
L'article s'intitule « Désordre programmable dans les pavages d'ADN aléatoires ».