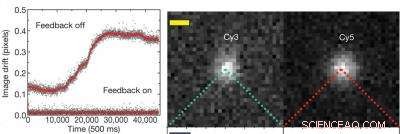
Le graphique de gauche montre qu'avec le système de rétroaction actif éteint, il y a une dérive de résolution d'environ 0,3 pixel ou 19 nanomètres, mais avec le système de rétroaction sur la résolution est maintenue à mieux que 0,01 pixel, ou environ 0,64 nanomètres. L'image de droite montre des molécules individuelles de colorant fluorescent de cyanine (Cy) - Cy3 et Cy5 - utilisées pour marquer 20 paires de bases d'ADN double brin. Crédit :Avec l'aimable autorisation du groupe de recherche Steven Chu
La sagesse conventionnelle soutient que la microscopie optique ne peut pas être utilisée pour "voir" quelque chose d'aussi petit qu'une molécule individuelle. Mais comme il est d'usage, la science intelligente a une fois de plus renversé la sagesse conventionnelle. Secrétaire à l'énergie, Le lauréat du prix Nobel et ancien directeur du Lawrence Berkeley National Laboratory (Berkeley Lab) Steven Chu a dirigé le développement d'une technique qui permet l'utilisation de la microscopie optique pour imager des objets ou la distance entre eux avec des résolutions aussi petites que 0,5 nanomètre - la moitié de un milliardième de mètre, ou un ordre de grandeur plus petit que le meilleur précédent.
« La capacité d'obtenir une résolution inférieure au nanomètre dans des environnements aqueux biologiquement pertinents a le potentiel de révolutionner la biologie, en particulier la biologie structurale, " dit le secrétaire Chu. " L'une des motivations de ce travail, par exemple, était de mesurer les distances entre les protéines qui forment des multi-domaines, structures très complexes, tels que l'assemblage de protéines qui forme le système d'ARN polymérase II humaine, qui initie la transcription de l'ADN."
La secrétaire Chu est co-auteur d'un article qui paraît maintenant dans la revue La nature qui décrit cette recherche. L'article s'intitule "Localisation subnanométrique d'une molécule unique, enregistrement et mesures de distance." Les autres auteurs sont Alexandros Pertsinidis, chercheur post-doctoral et membre du groupe de recherche de Chu à l'Université de Californie (UC) Berkeley, qui est maintenant professeur assistant à l'Institut Sloan-Kettering, et Yunxiang Zhang, membre du groupe de recherche de Chu à l'Université de Stanford.
Selon une loi de la physique connue sous le nom de "limite de diffraction, " la plus petite image qu'un système optique peut résoudre est environ la moitié de la longueur d'onde de la lumière utilisée pour produire cette image. Pour l'optique conventionnelle, cela correspond à environ 200 nanomètres. Par comparaison, une molécule d'ADN mesure environ 2,5 nanomètres de largeur.
Alors que les systèmes d'imagerie non optique, comme les microscopes électroniques, peut bien résoudre les objets à l'échelle subnanométrique, ces systèmes fonctionnent dans des conditions non idéales pour l'étude d'échantillons biologiques. Détecter des marqueurs fluorescents individuels attachés à des molécules biologiques d'intérêt à l'aide de dispositifs à couplage de charge (CCD) - des matrices de puces en silicium qui convertissent la lumière entrante en charge électrique, a donné des résolutions aussi fines que cinq nanomètres. Cependant, Jusqu'à présent, cette technologie était incapable d'imager des molécules uniques ou des distances entre une paire de molécules bien inférieures à 20 nanomètres.
Chu et ses co-auteurs ont pu utiliser la même technologie de fluorescence CCD pour résoudre les distances avec une précision et une précision subnanométriques en corrigeant un truc de la lumière. Les charges électriques dans un réseau CCD sont créées lorsque des photons frappent le silicium et délogent des électrons, la force de la charge étant proportionnelle à l'intensité des photons incidents. Cependant, en fonction de l'endroit précis où un photon frappe la surface d'une puce de silicium, il peut y avoir une légère différence dans la façon dont le photon est absorbé et s'il génère une charge mesurable. Cette non-uniformité dans la réponse de la matrice de silicium CCD aux photons entrants, qui est probablement un artefact du processus de fabrication de la puce, entraîne un flou des pixels qui rend difficile la résolution de deux points situés à quelques nanomètres l'un de l'autre.
"Nous avons développé un système de rétroaction active qui nous permet de placer l'image d'une seule molécule fluorescente n'importe où sur la matrice CCD avec une précision inférieure au pixel, ce qui nous permet à son tour de travailler dans une région plus petite que l'échelle de longueur typique de trois pixels de la non-uniformité du CCD, " dit Pertsinidis, qui est l'auteur principal de l'article sur Nature. "Avec ce système de rétroaction plus l'utilisation de faisceaux optiques supplémentaires pour stabiliser le système de microscope, nous pouvons créer une région calibrée sur le réseau de silicium où l'erreur due à la non-uniformité est réduite à 0,5 nanomètres. En plaçant les molécules que nous voulons mesurer au centre de cette région, nous pouvons obtenir une résolution subnanométrique à l'aide d'un microscope optique conventionnel que vous pouvez trouver dans n'importe quel laboratoire de biologie. »
Chu dit que la possibilité de déplacer la platine d'un microscope sur de petites distances et de calculer le centre géométrique (centroïde) de l'image permet non seulement de mesurer la non-uniformité de la photo-réponse entre les pixels, mais aussi pour mesurer la non-uniformité au sein de chaque pixel individuel.
"La connaissance de cette non-uniformité nous permet alors de faire des corrections entre la position apparente et la position réelle du centroïde de l'image, " dit Chu. " Étant donné que cette réponse non uniforme est intégrée dans la matrice CCD et ne change pas d'un jour à l'autre, notre système de rétroaction active nous permet d'imager à plusieurs reprises à la même position de la matrice CCD."
Pertsinidis continue de travailler avec Chu et d'autres membres du groupe sur le développement et l'application de cette technique de super-résolution. En plus du système d'ARN polymérase II humaine, lui et le groupe l'utilisent pour déterminer la structure des molécules de cadhérine épithéliale qui sont responsables de l'adhésion de cellule à cellule qui maintient ensemble les tissus et autres matériaux biologiques. Pertsinidis, Zhang, et un autre post-doctorant dans le groupe de recherche de Chu, Parc Sang Ryul, utilisent également cette technique pour créer des mesures 3D de l'organisation moléculaire à l'intérieur des cellules du cerveau.
"L'idée est de déterminer la structure et la dynamique du processus de fusion des vésicules qui libère les molécules de neurotransmetteur utilisées par les neurones pour communiquer entre eux, " dit Pertsinidis. " En ce moment, nous obtenons des mesures in situ avec une résolution d'environ 10 nanomètres, mais nous pensons que nous pouvons pousser cette résolution à moins de deux nanomètres."
En collaboration avec Joe Gray, Directeur associé pour les sciences de la vie du Berkeley Lab et chercheur de premier plan sur le cancer, les post-doctorants du groupe de recherche de Chu utilisent également la technique de super-résolution pour étudier la fixation de molécules de signalisation sur la protéine RAS, qui a été liée à un certain nombre de cancers, y compris ceux du sein, pancréas, poumon et côlon. Cette recherche pourrait aider à expliquer pourquoi les thérapies anticancéreuses qui donnent de bons résultats sur certains patients sont inefficaces sur d'autres.
En plus de ses applications biologiques, Pertsinidis, Zhang et Chu dans leur La nature article affirme que leur technique de super-résolution devrait également s'avérer utile pour caractériser et concevoir des systèmes d'imagerie photométrique de précision en physique atomique ou en astronomie, et permettre de nouveaux outils en lithographie optique et en nanométrologie.