Crédit :S. Strauss/MPI de Biochimie
La microscopie à fluorescence à super résolution peut être utilisée pour visualiser des structures inférieures à 200 nanomètres, c'est à dire., en dessous de la limite de diffraction de la lumière. L'une des techniques de microscopie, appelé DNA-PAINT, a été développé par Ralf Jungmann, chef de groupe de recherche au MPI de Biochimie et Professeur de Physique Expérimentale au LMU, avec des collègues. La technique utilise des « imageurs » courts, des brins d'ADN marqués par un colorant qui se lient temporairement à leurs molécules cibles de manière complémentaire pour produire le "clignotement" nécessaire à la reconstruction en super-résolution des images.
"Nous avons récemment amélioré la vitesse d'acquisition traditionnellement plutôt lente de DNA-PAINT d'un ordre de grandeur en optimisant la conception des séquences d'ADN, " dit Jungmann. " Cependant, cela s'est fait au prix de la perte du multiplexage, ce qui signifie que plusieurs structures dans la cellule ne peuvent pas être observées simultanément, " ajoute Jungmann. L'observation simultanée de plusieurs protéines, cependant, est important pour une meilleure compréhension des cascades de signalisation complexes entre les cellules tumorales et normales.
Cette capacité de multiplexage n'était pas réalisable dans DNA-PAINT à vitesse optimisée, car une seule séquence optimisée avec des caractéristiques d'hybridation améliorées était disponible. « Nous nous sommes demandé comment permettre l'imagerie multiplexée et, à la fois, pousser encore plus loin la vitesse d'acquisition des images, " dit Sébastien Strauss, premier auteur de l'article et collaborateur du groupe de Jungmann.
Dans l'étude actuelle, les chercheurs présentent un nouveau concept qui a réussi à améliorer la vitesse d'imagerie. Ils ont profité du fait que la fréquence de liaison des imageurs à leurs brins cibles varie linéairement avec le nombre de sites de liaison disponibles. "Plus il y a de sites de liaison, plus l'acquisition de l'image est rapide. Cependant, la simple concaténation des sites de liaison conduirait à des séquences d'amarrage excessivement longues, réduisant potentiellement la résolution d'image réalisable et augmentant la liaison non spécifique, " dit Strauss. Pour contourner ces problèmes, les chercheurs ont conçu des motifs de séquences répétitives, par exemple. (TCC)n, qui pourraient être concaténés pour fournir des sites de liaison qui se chevauchent tout en n'augmentant que légèrement la longueur des brins. "Nous avons conçu six personnes, motifs de séquences périodiques, ce qui nous permet d'introduire le multiplexage vers DNA-PAINT à vitesse optimisée, " a déclaré Strauss. " En combinaison avec les améliorations précédentes, nous pouvons maintenant accélérer DNA-PAINT d'un facteur 100, " ajoute Jungmann.
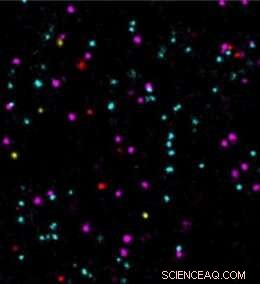
Pour optimiser les nouveaux motifs de séquence et comparer leurs améliorations, le groupe a utilisé des structures d'origami d'ADN, qui sont auto-assemblés, des objets d'ADN de taille nanométrique se repliant de manière autonome dans des formes prédéfinies. Ces structures peuvent être utilisées pour organiser des sites de liaison ADN-PAINT espacés précisément, par ex. distances de 5 nm. Cela a permis aux chercheurs d'évaluer les améliorations apportées à DNA-PAINT dans des conditions définies. "Les nouvelles séquences d'ADN optimisées nous ont permis de résoudre six structures d'origami d'ADN différentes au lieu d'une seule, en quelques minutes, " explique Strauss.
« Nous sommes ravis d'appliquer la vitesse d'imagerie désormais encore améliorée dans DNA-PAINT pour résoudre les questions biologiques. Par exemple, les marqueurs tumoraux ne pouvaient auparavant être examinés que lentement et pas clairement au niveau d'une seule molécule. Dans notre étude, la mesure de quatre marqueurs tumoraux différents confirme une analyse rapide et précise de leurs positions et interactions moléculaires. Cela pourrait fournir des informations importantes pour le développement de médicaments et leurs mécanismes d'action, " conclut Jungmann.