Des chercheurs de l'Université Brown ont montré qu'ils peuvent stocker une variété de fichiers d'images - un dessin de Picasso, une image du dieu égyptien Anubis et d'autres - dans des matrices de mélanges contenant de petites molécules synthétisées sur mesure. Dans tout, les chercheurs ont stocké plus de 200 kilo-octets de données, qui, selon eux, est le plus stocké à ce jour à l'aide de petites molécules. Crédit :Université Brown
Une équipe de chercheurs de l'Université Brown a fait des progrès substantiels dans le but de créer un nouveau type de système de stockage de données moléculaires.
Dans une étude publiée dans Communication Nature , l'équipe a stocké une variété de fichiers d'images :un dessin de Picasso, une image du dieu égyptien Anubis et d'autres, dans des matrices de mélanges contenant de petites molécules synthétisées sur mesure. Dans tout, les chercheurs ont stocké plus de 200 kilo-octets de données, qui, selon eux, est le plus stocké à ce jour à l'aide de petites molécules. Ce n'est pas beaucoup de données par rapport aux moyens de stockage traditionnels, mais c'est un progrès significatif en termes de stockage de petites molécules, disent les chercheurs.
"Je pense que c'est un pas en avant substantiel, " dit Jacob Rosenstein, professeur adjoint à la Brown's School of Engineering et auteur de l'étude. "Le grand nombre de petites molécules uniques, la quantité de données que nous pouvons stocker, et la fiabilité de la lecture des données est vraiment prometteuse pour étendre cela encore plus. »
Alors que l'univers des données continue de s'étendre, beaucoup de travail est fait pour trouver de nouveaux moyens de stockage plus compacts. En codant des données dans des molécules, il peut être possible de stocker l'équivalent de téraoctets de données dans quelques millimètres d'espace seulement. La plupart des recherches sur le stockage moléculaire se sont concentrées sur les polymères à longue chaîne comme l'ADN, qui sont des supports bien connus de données biologiques. Mais il y a des avantages potentiels à utiliser de petites molécules par opposition à de longs polymères. Les petites molécules sont potentiellement plus faciles et moins chères à produire que l'ADN synthétique, et ont en théorie une capacité de stockage encore plus élevée.
L'équipe de recherche Brown, soutenu par une subvention de la Defence Advanced Research Projects Agency (DARPA) des États-Unis dirigée par le professeur de chimie Brenda Rubenstein, a travaillé pour trouver des moyens de rendre le stockage de données de petites molécules réalisable et évolutif.
Pour stocker des données, l'équipe utilise de petites plaques métalliques alignées avec 1, 500 minuscules taches de moins d'un millimètre de diamètre. Chaque spot contient un mélange de molécules. La présence ou l'absence de molécules différentes dans chaque mélange indique les données numériques. Le nombre de bits dans chaque mélange peut être aussi grand que la bibliothèque de molécules distinctes disponibles pour le mélange. Les données peuvent ensuite être lues à l'aide d'un spectromètre de masse, qui permet d'identifier les molécules présentes dans chaque puits.
Dans un article publié l'année dernière, l'équipe Brown a montré qu'elle pouvait stocker des fichiers images de l'ordre du kilo-octet en utilisant certains métabolites courants, les molécules que les organismes utilisent pour réguler le métabolisme. Pour ce nouveau travail, les chercheurs ont pu étendre considérablement la taille de leur bibliothèque, et donc la taille des fichiers qu'ils pouvaient encoder, en synthétisant leurs propres molécules.
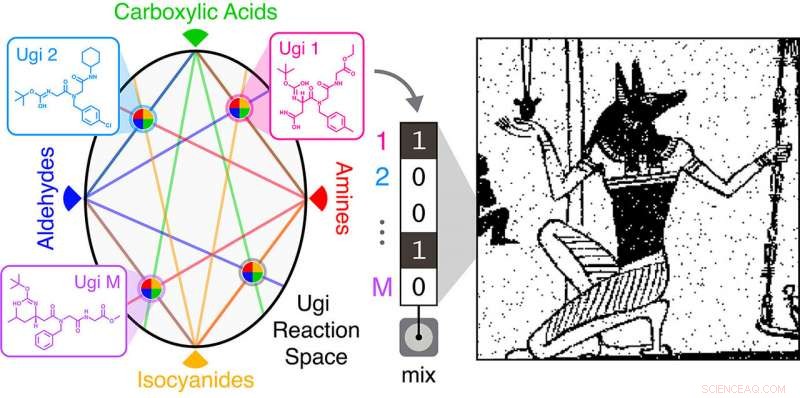
L'équipe a fabriqué ses molécules à l'aide de réactions d'Ugi, une technique souvent utilisée dans l'industrie pharmaceutique pour produire rapidement un grand nombre de composés différents. Les réactions d'Ugi combinent quatre grandes classes de réactifs (une amine, un aldéhyde ou une cétone, un acide carboxylique, et un isocyanure) en une nouvelle molécule. En utilisant des réactifs différents de chaque classe, les chercheurs pourraient rapidement produire un large éventail de molécules distinctes. Pour ce travail, l'équipe a utilisé cinq amines différentes, cinq aldéhydes, 12 acides carboxyliques, et cinq isocyanures dans différentes combinaisons pour créer 1, 500 composés distincts.
"L'avantage ici est l'évolutivité potentielle de la bibliothèque, " Rubenstein a déclaré. "Nous utilisons seulement 27 composants différents pour faire un 1, Banque de 500 molécules en une journée. Cela signifie que nous n'avons pas à sortir et à trouver 1, 500 molécules uniques."
De là, l'équipe a utilisé des sous-bibliothèques de composés pour coder leurs images. Une bibliothèque de 32 composés a été utilisée pour stocker une image binaire du dieu égyptien Anubis. Une bibliothèque de 575 composés a été utilisée pour coder un dessin Picasso de 0,88 mégapixel d'un violon.
Le grand nombre de molécules disponibles pour les bibliothèques chimiques a également permis aux chercheurs d'explorer des schémas de codage alternatifs qui ont rendu la lecture des données plus robuste. Alors que la spectrométrie de masse est très précise, ce n'est pas parfait. Ainsi, comme pour tout système utilisé pour stocker ou transmettre des données, ce système aura besoin d'une certaine forme de correction d'erreur.
"La façon dont nous concevons les bibliothèques et lisons les données inclut des informations supplémentaires qui nous permettent de corriger certaines erreurs, " a déclaré Chris Arcadia, étudiant diplômé de Brown, premier auteur de l'article. « Cela nous a aidés à rationaliser le flux de travail expérimental et à obtenir des taux de précision pouvant atteindre 99 %. »
Il y a encore du travail à faire pour porter cette idée à une échelle utile, disent les chercheurs. Mais la possibilité de créer de grandes bibliothèques chimiques et de les utiliser pour encoder des fichiers toujours plus volumineux suggère que l'approche peut en effet être étendue.
"Nous ne sommes plus limités par la taille de notre chimiothèque, ce qui est vraiment important, " Rosenstein a déclaré. "C'est le plus grand pas en avant ici. Lorsque nous avons commencé ce projet il y a quelques années, nous avons eu quelques débats pour savoir si quelque chose de cette échelle était même expérimentalement réalisable. C'est donc vraiment encourageant que nous ayons pu le faire. »