Les scientifiques associés au projet logiciel international MZmine, dirigé par le Dr Robin Schmid et le Dr Tomáš Pluskal de l'Institut de chimie organique et de biochimie de l'Académie tchèque des sciences, ont mis au point un nouveau logiciel qui accélère et simplifie considérablement l'identification de produits chimiques dans les tissus.
Il permet aux chercheurs de reconnaître en toute confiance les produits chimiques et de visualiser leur distribution dans les organes. Par rapport aux flux de travail établis, le nouveau pipeline nécessite moins de travail en laboratoire pour obtenir des informations similaires sur la chimie se produisant, par exemple, dans les tumeurs et les lésions inflammatoires. L'article présentant ce logiciel à la communauté scientifique mondiale vient d'être publié dans la revue Nature Communications .
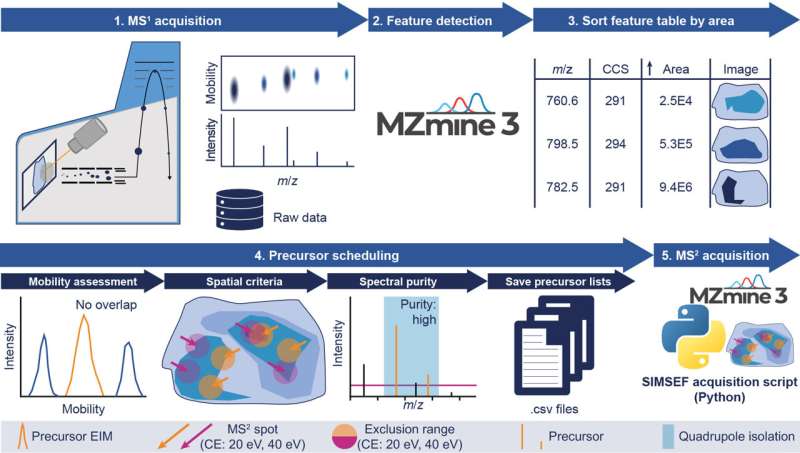
À l'IOCB Prague, le Dr Robin Schmid est principalement impliqué dans le développement du logiciel SIMSEF (fragmentation exhaustive programmée par mobilité ionique spatiale) avec ses collègues du groupe du professeur Dr. Karst de l'Université de Münster, en Allemagne, où il était auparavant basé.
Le travail est effectué en collaboration avec les développeurs du spectromètre de masse de pointe timsTOF fleX de Bruker Daltonics, qui permet l'élucidation de haut niveau de la composition des molécules en mesurant la mobilité des ions.
"Jusqu'à présent, les scientifiques pouvaient déterminer avec un spectromètre de masse qu'il s'agissait de la formule de la molécule qu'ils étudiaient, mais lorsqu'ils examinaient la base de données et essayaient d'identifier cette substance, c'était très difficile. La raison en est que les données biologiques les échantillons peuvent contenir de nombreux lipides distincts et des combinaisons de molécules qui sont souvent biologiquement très différentes."
"Maintenant, grâce au nouvel algorithme, il est possible de regarder à l'intérieur de la molécule, de savoir de quoi elle est composée et même de comparer les images entre elles", explique Robin Schmid. Cela peut fournir des informations importantes, par exemple qu'une partie spécifique du cerveau contient un type de lipide différent d'une autre partie de cet organe essentiel et que ce lipide particulier est absent dans d'autres tissus.
L'algorithme SIMSEF a été développé en collaboration avec des experts médicaux d'universités allemandes et suisses. La détection de biomarqueurs cliniques à des fins de diagnostic est également considérablement accélérée, ce qui est crucial pour les médecins. Savoir très rapidement, par exemple, s'il s'agit d'un lipide ou d'un métabolite que l'on trouve uniquement dans les tissus cancéreux, joue un rôle essentiel dans la décision d'un traitement ultérieur. Inutile de dire que cela a de grandes implications sur son résultat.
Le nouvel algorithme fait partie du progiciel open source MZmine, qui aide les experts du monde entier à analyser les données de spectrométrie de masse depuis 2005. Steffen Heuckeroth de l'équipe MZmine et de l'Institut de chimie inorganique et analytique de l'Université de Münster a été le principal développeur de l'algorithme SIMSEF et est le premier auteur de l'article maintenant publié dans Nature Communications .
Il ajoute :"L'article en question ne contient aucune découverte spécifique dans le domaine de la biologie. Cependant, grâce à notre nouvelle méthode, de nombreux scientifiques peuvent améliorer et accélérer leurs travaux, ce qui ouvre la voie à de nouvelles découvertes."
"Je trouve toujours formidable de permettre à d'autres personnes d'améliorer les conditions de travail de leurs recherches et également de les communiquer à d'autres scientifiques. Grâce aux retours de la communauté scientifique utilisant MZmine, le programme lui-même fait directement avancer leurs recherches", ajoute Robin Schmid du IOCB Prague.
Le directeur de l'IOCB Prague, le professeur Jan Konvalinka, souligne :« De nos jours, tout le monde parle d'apprentissage automatique et d'intelligence artificielle. Nos collègues de l'équipe du Dr Pluskal les utilisent pour résoudre des problèmes très spécifiques dans la recherche ainsi que dans la pratique clinique.
La troisième génération de MZmine, rapportée au printemps 2023 dans la revue Nature Biotechnology , peut traiter des milliers d’échantillons par heure. Les algorithmes nouvellement développés par cette équipe internationale améliorent ainsi encore les capacités existantes pour relier différents types de données, y compris la combinaison de données provenant de méthodes d'analyse et d'imagerie, ce qui jusqu'à présent n'était possible avec aucun logiciel académique ou commercial.
Plus d'informations : Steffen Heuckeroth et al, Bioimagerie MALDI-TIMS-MS2 dépendant de l'ensemble de données sur les tissus, Nature Communications (2023). DOI :10.1038/s41467-023-43298-9
Informations sur le journal : Communications naturelles , Biotechnologie naturelle
Fourni par l'Institut de chimie organique et de biochimie de l'Académie tchèque des sciences (IOCB Prague)