La rétrosynthèse vise à prédire un ensemble de réactifs pour produire des molécules données, qui jouent un rôle important dans le domaine de la biochimie, comme la conception de voies moléculaires et la découverte de médicaments. La plupart des méthodes existantes ne bénéficient que d'un seul type d'informations plutôt que de considérer davantage les divers aspects de l'information moléculaire.
Pour résoudre ce problème, une équipe de recherche a mené une étude, désormais publiée dans Frontiers of Computer Science. .
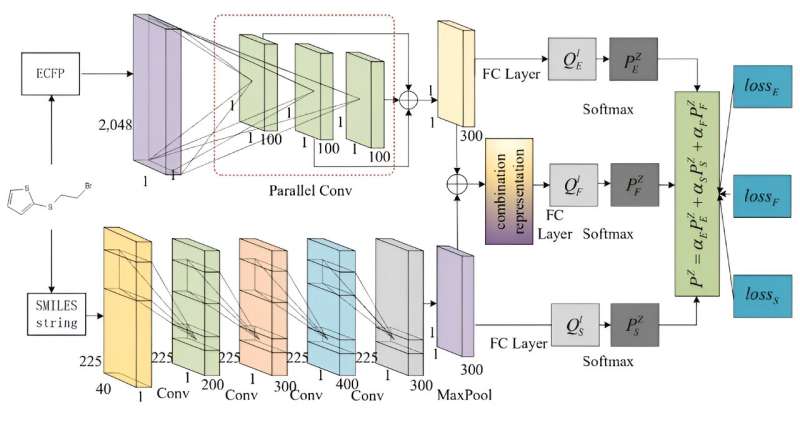
L'équipe a proposé un réseau multi-flux pour la prédiction de la rétrosynthèse en décrivant les molécules sous plusieurs perspectives en utilisant leurs descripteurs SMILES et ECFP.
MSNR se compose de trois modules principaux :(i) Le CNN parallèle et le CNN texte qui prennent les ECFP et les SMILES après un codage à chaud comme entrée pour produire les fonctionnalités approfondies. (ii) La représentation combinée est conçue en fusionnant les deux types de caractéristiques profondes des ECFP et des SMILES, qui offrent une perspective approfondie de la représentation moléculaire. (iii) Trois classificateurs denses ont été mis en œuvre pour prédire la probabilité de réactifs pour les molécules, qui exploitent respectivement les caractéristiques profondes extraites par différents flux comme représentation moléculaire.
En fusionnant ces résultats de prédiction multi-flux avec des poids variables, le modèle arrive à une prédiction de rétrosynthèse finale. De plus, le modèle est entraîné à l'aide d'une fonction de perte globale capable d'exploiter les diverses informations disponibles pour chaque type de fonctionnalité approfondie.
Plus d'informations : Qiang Zhang et al, Un réseau multi-flux pour la prédiction de la rétrosynthèse, Frontiers of Computer Science (2023). DOI :10.1007/s11704-023-3103-z
Fourni par Frontiers Journals