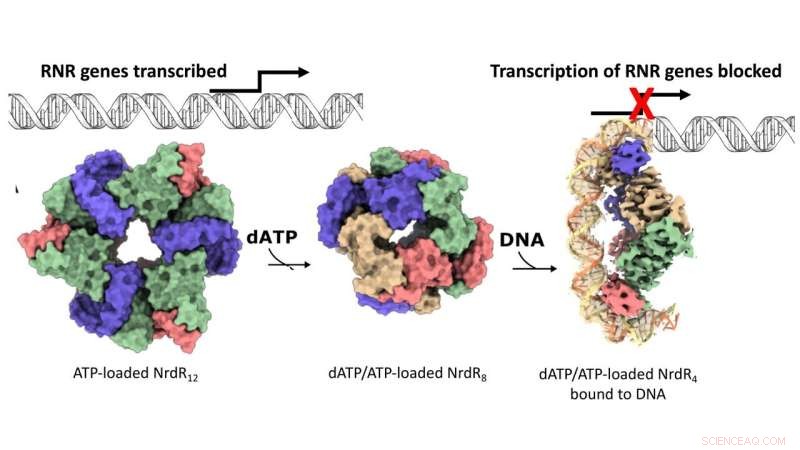
Le mécanisme d'action du répresseur spécifique du RNR NrdR. Représentation de surface des cartes cryo-EM pour les structures NrdR tétramères dodécamériques, octamériques et liées à l'ADN. Les monomères NrdR dans chaque assemblage tétramère sont de couleur beige, bleu, vert et rose. L'ADN est représenté schématiquement, à l'exception de la partie résolue par cryo-EM où les deux brins d'ADN antiparallèles sont respectivement orange et jaune clair. Crédit :Inna Rozman Grinberg
Dans une nouvelle étude publiée dans Nature Communications , des chercheurs de l'Université de Stockholm montrent pour la première fois comment le NrdR se lie à l'ADN pour inhiber le RNR. Le nouveau mécanisme pourrait aider les scientifiques à concevoir de meilleurs antibiotiques en ciblant la capacité d'un agent pathogène à se reproduire.
"Nous avons découvert le NrdR il y a plus de dix ans, mais son fonctionnement était une énigme. Dans cet article, nous avons combiné des études biochimiques et structurelles pour découvrir comment le NrdR se lie à l'ADN", explique Inna Rozman Grinberg, chercheuse au Département de biochimie et Biophysique à l'Université de Stockholm et auteur principal de l'étude.
La synthèse de l'ADN est le processus de création de nouvelles molécules d'ADN qui a lieu lorsqu'une cellule se divise. Dans pratiquement tous les organismes vivants, l'enzyme ribonucléotide réductase (RNR) fournit des blocs de construction pour les nouveaux brins d'ADN. Chez de nombreuses bactéries, y compris des agents pathogènes bien connus tels que Mycobacterium tuberculosis, la synthèse de RNR est désactivée par la protéine répresseur NrdR. Cependant, le mécanisme qui sous-tend l'interaction entre RNR et NrdR est resté insaisissable. En général, lorsque les cellules bactériennes se divisent, le RNR est activé pour fournir des éléments de base pour la fabrication d'un nouvel ADN. Entre-temps, NrdR reste désactivé. Ensuite, lorsque la synthèse de l'ADN est terminée, NrdR se lie à l'ADN, empêchant ainsi la synthèse de RNR.
Mais comment les cellules détectent-elles qu'il est temps de désactiver le RNR ? La réponse réside dans le rapport de concentration relative entre deux molécules clés, à savoir l'adénosine triphosphate (ATP) et la désoxyadénosine triphosphate (dATP). Les chercheurs ont montré que certains RNR peuvent s'éteindre lorsque des niveaux élevés de dATP sont présents. NrdR, d'autre part, ne se lie à l'ADN et inhibe la synthèse de RNR que lorsque les niveaux de dATP sont élevés. Ce mécanisme de détection, qui est presque exclusivement utilisé par RNR et NrdR, est médié par un domaine protéique appelé "ATP-cone".
"Ce mécanisme met en évidence un aspect passionnant de l'évolution des protéines", déclare Inna Rozman Grinberg.
Selon Inna Rozman Grinberg, puisque le NrdR est abondant chez les bactéries mais absent chez l'homme, le cône ATP pourrait être utilisé comme cible antibiotique contre les agents pathogènes avec un seul RNR, ou contre les agents pathogènes avec NrdR pour contrôler tous leurs RNR. "À notre connaissance, c'est le seul exemple d'un domaine protéique mobile évolutif qui contrôle à la fois l'expression et l'activité d'une enzyme." Le "commutateur" RNR offre de l'espoir dans la lutte contre les bactéries résistantes aux antibiotiques