Crédit :Université de Kanazawa
Des chercheurs de l'Université de Kanazawa rapportent dans le Journal of Physical Chemistry Letters comment simuler des images de microscopie à force atomique 3D de systèmes hors équilibre impliquant des biomolécules. L'approche utilise une célèbre équation de la thermodynamique applicable aux situations de non-équilibre.
La microscopie tridimensionnelle à force atomique (3D-AFM) est une technique utilisée pour sonder la distribution des molécules de solvant aux interfaces solide-liquide. Initialement appliquée pour étudier des situations où le solvant est de l'eau, la méthode est maintenant également utilisée pour d'autres molécules. Un développement récent consiste à utiliser la 3D-AFM pour résoudre l'organisation des biopolymères tels que les chromosomes ou les protéines dans les cellules. En raison de la complexité de ces systèmes, cependant, des simulations du processus d'imagerie 3D-AFM sont nécessaires pour aider à son interprétation. Les méthodes de simulation développées jusqu'à présent ont supposé que le système sondé est en équilibre pendant le cycle de balayage AFM. Cela limite leur validité aux situations où les molécules de solvant se déplacent beaucoup plus rapidement que la sonde de balayage. Maintenant, Takeshi Fukuma de l'Université de Kanazawa et ses collègues ont développé une approche de simulation 3D-AFM qui fonctionne pour les systèmes non en équilibre, applicable aux situations où le mouvement moléculaire se produit sur des échelles de temps comparables ou supérieures à celles du cycle de sondage AFM.
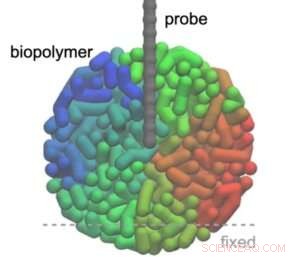
Le principe de base de l'AFM est de fabriquer une très petite pointe, attachée à un porte-à-faux, pour balayer la surface d'un échantillon. La réponse de la pointe aux différences de hauteur dans la surface scannée fournit des informations structurelles sur l'échantillon. En 3D-AFM, la pointe est amenée à pénétrer dans l'échantillon, et la force subie par la pointe est le résultat d'interactions avec des (parties de) molécules proches. Pour une position horizontale (xy) donnée de la pointe, la dépendance de la force F sur la position verticale (z) de la pointe lorsqu'elle pénètre dans l'échantillon est capturée dans une courbe force-distance (F contre z). La combinaison de toutes les courbes force-distance obtenues lors du balayage xy donne l'image 3D-AFM.
Fukuma et ses collègues ont examiné la situation où une pointe AFM sonde un biopolymère globulaire et ont modélisé à la fois la pointe et la molécule sous forme de billes reliées par des ressorts (2 000 billes pour la molécule, 50 billes pour la pointe). Ils ont calculé les courbes force-distance en utilisant l'égalité dite de Jarzynski, une équation qui relie la différence d'énergie libre entre deux états d'un système au travail (proportionnel à la force) nécessaire pour passer d'un état à l'autre. Il est important de noter que l'égalité est valable pour les situations de non-équilibre.
Les chercheurs ont pu montrer que les simulations reproduisaient la structure interne du biopolymère, certaines caractéristiques des fibres étant clairement observables. Ils ont également examiné comment la vitesse de balayage affecte les résultats de la simulation et ont découvert qu'il existe une plage de vitesse optimale pour le balayage vertical (z). Enfin, Fukuma et ses collègues ont simulé des images 3D-AFM de fibres de cytosquelette pour lesquelles des images 3D-AFM obtenues expérimentalement existent, et ont constaté que les simulations concordaient bien avec l'expérience. Les scientifiques ont donc conclu que leur méthode "est applicable à diverses fibres dans les cellules telles que l'ADN et ainsi de suite en modifiant des paramètres tels que la rigidité, fournissant une base théorique importante pour de telles mesures expérimentales". Affectation de caractéristiques mobiles en microscopie à force atomique à grande vitesse